+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3374 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex | |||||||||
 マップデータ マップデータ | Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex. Post-processed with RELION, using auto-masking with initial binarization threshold of 0.011. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | T4 / baseplate-tail tube complex / pre-attachment / bacteriophage / bacterial virus / hexagonal / membrane-piercing / cell attachment / infection | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peptidoglycan beta-N-acetylmuramidase activity / symbiont genome ejection through host cell envelope, contractile tail mechanism / symbiont entry into host cell via disruption of host cell wall peptidoglycan / virus tail, tube / virus tail, baseplate / virus tail, fiber / viral tail assembly / symbiont entry into host cell via disruption of host cell envelope / virus tail / symbiont entry into host ...peptidoglycan beta-N-acetylmuramidase activity / symbiont genome ejection through host cell envelope, contractile tail mechanism / symbiont entry into host cell via disruption of host cell wall peptidoglycan / virus tail, tube / virus tail, baseplate / virus tail, fiber / viral tail assembly / symbiont entry into host cell via disruption of host cell envelope / virus tail / symbiont entry into host / viral release from host cell / peptidoglycan catabolic process / cell wall macromolecule catabolic process / lysozyme / lysozyme activity / killing of cells of another organism / entry receptor-mediated virion attachment to host cell / defense response to bacterium / symbiont entry into host cell / structural molecule activity / metal ion binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.11 Å | |||||||||
 データ登録者 データ登録者 | Taylor NMI / Guerrero-Ferreira RC / Goldie KN / Stahlberg H / Leiman PG | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Structure of the T4 baseplate and its function in triggering sheath contraction. 著者: Nicholas M I Taylor / Nikolai S Prokhorov / Ricardo C Guerrero-Ferreira / Mikhail M Shneider / Christopher Browning / Kenneth N Goldie / Henning Stahlberg / Petr G Leiman /   要旨: Several systems, including contractile tail bacteriophages, the type VI secretion system and R-type pyocins, use a multiprotein tubular apparatus to attach to and penetrate host cell membranes. This ...Several systems, including contractile tail bacteriophages, the type VI secretion system and R-type pyocins, use a multiprotein tubular apparatus to attach to and penetrate host cell membranes. This macromolecular machine resembles a stretched, coiled spring (or sheath) wound around a rigid tube with a spike-shaped protein at its tip. A baseplate structure, which is arguably the most complex part of this assembly, relays the contraction signal to the sheath. Here we present the atomic structure of the approximately 6-megadalton bacteriophage T4 baseplate in its pre- and post-host attachment states and explain the events that lead to sheath contraction in atomic detail. We establish the identity and function of a minimal set of components that is conserved in all contractile injection systems and show that the triggering mechanism is universally conserved. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3374.map.gz emd_3374.map.gz | 38.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3374-v30.xml emd-3374-v30.xml emd-3374.xml emd-3374.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3374_fsc.xml emd_3374_fsc.xml | 16.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3374.png emd_3374.png | 149.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3374 http://ftp.pdbj.org/pub/emdb/structures/EMD-3374 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3374 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3374 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3374.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3374.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-electron microscopy structure of the hexagonal pre-attachment T4 baseplate-tail tube complex. Post-processed with RELION, using auto-masking with initial binarization threshold of 0.011. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.326 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
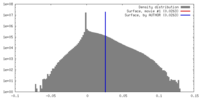
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Hexagonal pre-attachment T4 baseplate-tail tube complex
| 全体 | 名称: Hexagonal pre-attachment T4 baseplate-tail tube complex |
|---|---|
| 要素 |
|
-超分子 #1000: Hexagonal pre-attachment T4 baseplate-tail tube complex
| 超分子 | 名称: Hexagonal pre-attachment T4 baseplate-tail tube complex タイプ: sample / ID: 1000 詳細: In addition to hexagonal pre-attachment baseplate-tail tube complexes, the sample also contained some star-shaped, hubless post-attachment baseplates.The current reconstruction is the overall ...詳細: In addition to hexagonal pre-attachment baseplate-tail tube complexes, the sample also contained some star-shaped, hubless post-attachment baseplates.The current reconstruction is the overall reconstruction of the hexagonal pre-attachment baseplate-tail tube complex. 集合状態: (gp3)6(gp5)3(gp5.4)1(gp6)12(gp7)6(gp8)12(gp9)18(gp10)18(gp11)18(gp12)18(gp19)126(gp25)6(gp27)3(gp29)X(gp48)6(gp53)6(gp54)6 Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 8.7 MDa |
-分子 #1: Hexagonal pre-attachment T4 baseplate-tail tube complex
| 分子 | 名称: Hexagonal pre-attachment T4 baseplate-tail tube complex タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage T4 (ファージ) / 株: am18/am23 mutant Enterobacteria phage T4 (ファージ) / 株: am18/am23 mutant |
| 分子量 | 理論値: 8.7 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 50 mM Tris-Hcl pH 8.0, 100 mM NaCl, 8 mM MgSO4 |
| グリッド | 詳細: Quantifoil 300 mesh carbon-coated copper grids glow-discharged for 20 seconds |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 手法: Applied 3.5 ul of sample and blotting 3 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 平均: 80 K |
| アライメント法 | Legacy - 非点収差: The astigmatism was corrected at high magnification |
| 特殊光学系 | エネルギーフィルター - 名称: Quantum-LS Gatan Image Filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2015年5月7日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 1621 / 平均電子線量: 60 e/Å2 詳細: Individual frames were aligned with 2dx_automator. 40 frames were recorded in total, and the 2 first frames were discarded. ビット/ピクセル: 32 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 37700 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















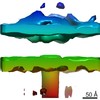








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)