+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-32195 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Coxsackievirus B3(VP3-234N) incubate with CD55 at pH7.4 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CVB3 / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / regulation of complement-dependent cytotoxicity / regulation of complement activation / respiratory burst / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / Class B/2 (Secretin family receptors) / ficolin-1-rich granule membrane / complement activation, classical pathway ...regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / regulation of complement-dependent cytotoxicity / regulation of complement activation / respiratory burst / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / Class B/2 (Secretin family receptors) / ficolin-1-rich granule membrane / complement activation, classical pathway / transport vesicle / side of membrane / COPI-mediated anterograde transport / endoplasmic reticulum-Golgi intermediate compartment membrane / secretory granule membrane / Regulation of Complement cascade / positive regulation of T cell cytokine production / virus receptor activity / positive regulation of cytosolic calcium ion concentration / membrane raft / Golgi membrane / innate immune response / lipid binding / Neutrophil degranulation / cell surface / extracellular exosome / extracellular region / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Coxsackievirus B3 (コクサッキーウイルス) / Coxsackievirus B3 (コクサッキーウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
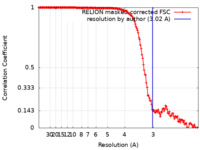
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.02 Å | |||||||||
 データ登録者 データ登録者 | Wang QL / Liu CC | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Molecular basis of differential receptor usage for naturally occurring CD55-binding and -nonbinding coxsackievirus B3 strains. 著者: Qingling Wang / Qian Yang / Congcong Liu / Guoqing Wang / Hao Song / Guijun Shang / Ruchao Peng / Xiao Qu / Sheng Liu / Yingzi Cui / Peiyi Wang / Wenbo Xu / Xin Zhao / Jianxun Qi / Mengsu Yang / George F Gao /  要旨: Receptor usage defines cell tropism and contributes to cell entry and infection. Coxsackievirus B (CVB) engages coxsackievirus and adenovirus receptor (CAR), and selectively utilizes the decay- ...Receptor usage defines cell tropism and contributes to cell entry and infection. Coxsackievirus B (CVB) engages coxsackievirus and adenovirus receptor (CAR), and selectively utilizes the decay-accelerating factor (DAF; CD55) to infect cells. However, the differential receptor usage mechanism for CVB remains elusive. This study identified VP3-234 residues (234Q/N/V/D/E) as critical population selection determinants during CVB3 virus evolution, contributing to diverse binding affinities to CD55. Cryoelectron microscopy (cryo-EM) structures of CD55-binding/nonbinding isolates and their complexes with CD55 or CAR were obtained under both neutral and acidic conditions, and the molecular mechanism of VP3-234 residues determining CD55 affinity/specificity for naturally occurring CVB3 strains was elucidated. Structural and biochemical studies in vitro revealed the dynamic entry process of CVB3 and the function of the uncoating receptor CAR with different pH preferences. This work provides detailed insight into the molecular mechanism of CVB infection and contributes to an in-depth understanding of enterovirus attachment receptor usage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32195.map.gz emd_32195.map.gz | 84.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32195-v30.xml emd-32195-v30.xml emd-32195.xml emd-32195.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32195_fsc.xml emd_32195_fsc.xml | 15.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32195.png emd_32195.png | 133.7 KB | ||
| マスクデータ |  emd_32195_msk_1.map emd_32195_msk_1.map | 307.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-32195.cif.gz emd-32195.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32195 http://ftp.pdbj.org/pub/emdb/structures/EMD-32195 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32195 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32195 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32195_validation.pdf.gz emd_32195_validation.pdf.gz | 682.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32195_full_validation.pdf.gz emd_32195_full_validation.pdf.gz | 681.7 KB | 表示 | |
| XML形式データ |  emd_32195_validation.xml.gz emd_32195_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_32195_validation.cif.gz emd_32195_validation.cif.gz | 19.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32195 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32195 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32195 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32195 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7vy6MC  7vxhC  7vxzC  7vy0C  7vy5C  7vykC  7vylC  7vymC  7w14C  7w17C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32195.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32195.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_32195_msk_1.map emd_32195_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Coxsackievirus B3
| 全体 | 名称:   Coxsackievirus B3 (コクサッキーウイルス) Coxsackievirus B3 (コクサッキーウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Coxsackievirus B3
| 超分子 | 名称: Coxsackievirus B3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus B3 (コクサッキーウイルス) Coxsackievirus B3 (コクサッキーウイルス) |
-超分子 #3: Complement decay-accelerating factor
| 超分子 | 名称: Complement decay-accelerating factor / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5 |
|---|
-超分子 #2: Homo sapiens
| 超分子 | 名称: Homo sapiens / タイプ: virus / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 / NCBI-ID: 9606 / 生物種: Homo sapiens / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus B3 (コクサッキーウイルス) Coxsackievirus B3 (コクサッキーウイルス) |
| 分子量 | 理論値: 31.703477 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPVEDVITAA IGRVADTVGT GPTNSEAIPA LTAAETGHTS QVVPGDTMQT RHVKNYHSRS ESTVENFLCR SACVYFTEYK NSGSKRYAE WVVTTRQAAQ LRRKLEFFTY IRFDLELTFV ITSTQQPSTT QNQDAQILTH QIMYVPPGGP VPDKVDSYVW Q TSTNPSVF ...文字列: GPVEDVITAA IGRVADTVGT GPTNSEAIPA LTAAETGHTS QVVPGDTMQT RHVKNYHSRS ESTVENFLCR SACVYFTEYK NSGSKRYAE WVVTTRQAAQ LRRKLEFFTY IRFDLELTFV ITSTQQPSTT QNQDAQILTH QIMYVPPGGP VPDKVDSYVW Q TSTNPSVF WTEGNAPPRM SIPFLSIGNA YSNFYDGWSE FSRNGVYGIN TLNNMGTLYA RHVNTGSTGP IKSTIRIYFK PK HVKAWIP RPPRLCQYEK AKNVNFQPSG VTTTRQSITT MTNTGAF |
-分子 #2: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus B3 (コクサッキーウイルス) Coxsackievirus B3 (コクサッキーウイルス) |
| 分子量 | 理論値: 28.862561 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SPTVEECGYS DRVRSITLGN STITTQECAN VVVGYGVWPD YLKDSEATAE DQPTQPDVAT CRFYTLDSVQ WQKTSPGWWW KLPDALSNL GLFGQNMQYH YLGRTGYTIH VQCNASKFHQ GCLLVVCVPE AEMGCATLDN TPSSAELLGG DAAKEFAGEP I ASGSNKLV ...文字列: SPTVEECGYS DRVRSITLGN STITTQECAN VVVGYGVWPD YLKDSEATAE DQPTQPDVAT CRFYTLDSVQ WQKTSPGWWW KLPDALSNL GLFGQNMQYH YLGRTGYTIH VQCNASKFHQ GCLLVVCVPE AEMGCATLDN TPSSAELLGG DAAKEFAGEP I ASGSNKLV QRVVYNAGMG IGVGNLTIFP HQWINLRTNN SATIVMPYTN SVPMDNMFRH NNITLMVIPF VPLDYCPGST TY VPITVTI APMCAEYNGL RLASHQ |
-分子 #3: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus B3 (コクサッキーウイルス) Coxsackievirus B3 (コクサッキーウイルス) |
| 分子量 | 理論値: 26.128633 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GLPTMNTPGS CQFLTSDDFQ SPSAMPQYDV TPEMKIPGEV KNLMEIAEVD SVVPVQNVGE KVNSMEAYQI PVRSNEGSGT QVFGFPLQP GYSSVFSRTL LGEILNYYTH WSGSIKLTFM FCGSAMATGK FLLAYSPPGA GAPTKRVDAM LGTHVVWDVG L QSSCVLCI ...文字列: GLPTMNTPGS CQFLTSDDFQ SPSAMPQYDV TPEMKIPGEV KNLMEIAEVD SVVPVQNVGE KVNSMEAYQI PVRSNEGSGT QVFGFPLQP GYSSVFSRTL LGEILNYYTH WSGSIKLTFM FCGSAMATGK FLLAYSPPGA GAPTKRVDAM LGTHVVWDVG L QSSCVLCI PWISQTHYRY VASDEYTAGG FITCWYQTNI VVPADAQSSC YIMCFVSACN DFSVRLLKDT PFISQNSFFQ |
-分子 #4: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus B3 (コクサッキーウイルス) Coxsackievirus B3 (コクサッキーウイルス) |
| 分子量 | 理論値: 7.437209 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGAQVSTQKT GAHETGLNAS GNSIIHYTNI NYYKDAASNS ATRQDFAQDP GKFTEPVKDI MIKSLPALN |
-分子 #5: Complement decay-accelerating factor
| 分子 | 名称: Complement decay-accelerating factor / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.641074 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DCGLPPDVPN AQPALEGRTS FPEDTVITYK CEESFVKIPG EKDSVICLKG SQWSDIEEFC NRSCEVPTRL NSASLKQPYI TQNYFPVGT VVEYECRPGY RREPSLSPKL TCLQNLKWST AVEFCKKKSC PNPGEIRNGQ IDVPGGILFG ATISFSCNTG Y KLFGSTSS ...文字列: DCGLPPDVPN AQPALEGRTS FPEDTVITYK CEESFVKIPG EKDSVICLKG SQWSDIEEFC NRSCEVPTRL NSASLKQPYI TQNYFPVGT VVEYECRPGY RREPSLSPKL TCLQNLKWST AVEFCKKKSC PNPGEIRNGQ IDVPGGILFG ATISFSCNTG Y KLFGSTSS FCLISGSSVQ WSDPLPECRE IYCPAPPQID NGIIQGERDH YGYRQSVTYA CNKGFTMIGE HSIYCTVNND EG EWSGPPP ECRGHHHHHH UniProtKB: Complement decay-accelerating factor |
-分子 #6: PALMITIC ACID
| 分子 | 名称: PALMITIC ACID / タイプ: ligand / ID: 6 / コピー数: 1 / 式: PLM |
|---|---|
| 分子量 | 理論値: 256.424 Da |
| Chemical component information |  ChemComp-PLM: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均露光時間: 1.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 1.8 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||||||
| 得られたモデル |  PDB-7vy6: |
 ムービー
ムービー コントローラー
コントローラー
































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)