+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31258 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | STRUCTURE OF PHOTOSYNTHETIC LH1-RC SUPER-COMPLEX OF RHODOSPIRILLUM RUBRUM | |||||||||||||||||||||
 マップデータ マップデータ | LH1-RC complex of Rsp. rubrum | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | LH1-RC COMPLEX / PHOTOSYNTHESIS / PURPLE BACTERIA | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / : / metal ion binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Rhodospirillum rubrum (バクテリア) / Rhodospirillum rubrum (バクテリア) /  Rhodospirillum rubrum (strain ATCC 11170 / ATH 1.1.1 / DSM 467 / LMG 4362 / NCIB 8255 / S1) (バクテリア) Rhodospirillum rubrum (strain ATCC 11170 / ATH 1.1.1 / DSM 467 / LMG 4362 / NCIB 8255 / S1) (バクテリア) | |||||||||||||||||||||
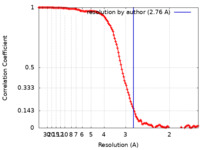
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.76 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Tani K / Kanno R | |||||||||||||||||||||
| 資金援助 |  日本, 6件 日本, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2021 ジャーナル: Biochemistry / 年: 2021タイトル: Cryo-EM Structure of the Photosynthetic LH1-RC Complex from . 著者: Kazutoshi Tani / Ryo Kanno / Xuan-Cheng Ji / Malgorzata Hall / Long-Jiang Yu / Yukihiro Kimura / Michael T Madigan / Akira Mizoguchi / Bruno M Humbel / Zheng-Yu Wang-Otomo /    要旨: () is one of the most widely used model organisms in bacterial photosynthesis. This purple phototroph is characterized by the presence of both rhodoquinone (RQ) and ubiquinone as electron carriers ... () is one of the most widely used model organisms in bacterial photosynthesis. This purple phototroph is characterized by the presence of both rhodoquinone (RQ) and ubiquinone as electron carriers and bacteriochlorophyll (BChl) esterified at the propionic acid side chain by geranylgeraniol (BChl ) instead of phytol. Despite intensive efforts, the structure of the light-harvesting-reaction center (LH1-RC) core complex from remains at low resolutions. Using cryo-EM, here we present a robust new view of the LH1-RC at 2.76 Å resolution. The LH1 complex forms a closed, slightly elliptical ring structure with 16 αβ-polypeptides surrounding the RC. Our biochemical analysis detected RQ molecules in the purified LH1-RC, and the cryo-EM density map specifically positions RQ at the Q site in the RC. The geranylgeraniol side chains of BChl coordinated by LH1 β-polypeptides exhibit a highly homologous tail-up conformation that allows for interactions with the bacteriochlorin rings of nearby LH1 α-associated BChls . The structure also revealed key protein-protein interactions in both N- and C-terminal regions of the LH1 αβ-polypeptides, mainly within a face-to-face structural subunit. Our high-resolution LH1-RC structure provides new insight for evaluating past experimental and computational results obtained with this old organism over many decades and lays the foundation for more detailed exploration of light-energy conversion, quinone transport, and structure-function relationships in this pigment-protein complex. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31258.map.gz emd_31258.map.gz | 166.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31258-v30.xml emd-31258-v30.xml emd-31258.xml emd-31258.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31258_fsc.xml emd_31258_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31258.png emd_31258.png | 168.8 KB | ||
| Filedesc metadata |  emd-31258.cif.gz emd-31258.cif.gz | 7.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31258 http://ftp.pdbj.org/pub/emdb/structures/EMD-31258 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31258 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31258 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31258.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31258.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LH1-RC complex of Rsp. rubrum | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Photosynthetic LH1-RC complex from the purple phototrophic bacter...
+超分子 #1: Photosynthetic LH1-RC complex from the purple phototrophic bacter...
+分子 #1: Reaction center protein L chain
+分子 #2: Reaction center protein M chain
+分子 #3: Photoreaction center protein H
+分子 #4: Light-harvesting protein B-870 alpha chain
+分子 #5: Light-harvesting protein B-870 beta chain
+分子 #6: Trans-Geranyl BACTERIOCHLOROPHYLL A
+分子 #7: Trans-Geranyl BACTERIOPHEOPHYTIN A
+分子 #8: UBIQUINONE-10
+分子 #9: (1R)-2-{[{[(2S)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
+分子 #10: DODECYL-BETA-D-MALTOSIDE
+分子 #11: FE (III) ION
+分子 #12: 2-azanyl-5-[(2~{E},6~{E},8~{E},10~{E},12~{E},14~{E},18~{E},22~{E}...
+分子 #13: SPIRILLOXANTHIN
+分子 #14: CARDIOLIPIN
+分子 #15: DI-PALMITOYL-3-SN-PHOSPHATIDYLETHANOLAMINE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/2 / 材質: MOLYBDENUM / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均露光時間: 30.6 sec. / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)