+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31178 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Lassa virus polymerase in complex with 3'-vRNA and Z mutant (F36A) | |||||||||
 マップデータ マップデータ | The EM map of Lassa L in complex with 3'-vRNA and Z mutant (F36A) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Lassa virus / Polymerase / Z protein / replication regulation / VIRAL PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative stranded viral RNA replication / viral budding via host ESCRT complex / cap snatching / viral budding from plasma membrane / virion component / host cell / 加水分解酵素; エステル加水分解酵素 / host cell cytoplasm / hydrolase activity / host cell perinuclear region of cytoplasm ...negative stranded viral RNA replication / viral budding via host ESCRT complex / cap snatching / viral budding from plasma membrane / virion component / host cell / 加水分解酵素; エステル加水分解酵素 / host cell cytoplasm / hydrolase activity / host cell perinuclear region of cytoplasm / RNA-directed RNA polymerase / RNA-directed RNA polymerase activity / nucleotide binding / host cell plasma membrane / RNA binding / zinc ion binding / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) | |||||||||
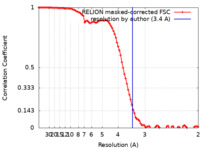
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Xu X / Peng R | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2021 ジャーナル: Nat Microbiol / 年: 2021タイトル: Cryo-EM structures of Lassa and Machupo virus polymerases complexed with cognate regulatory Z proteins identify targets for antivirals. 著者: Xin Xu / Ruchao Peng / Qi Peng / Min Wang / Ying Xu / Sheng Liu / Xiaolin Tian / Haiteng Deng / Yimin Tong / Xiaoyou Hu / Jin Zhong / Peiyi Wang / Jianxun Qi / George F Gao / Yi Shi /  要旨: Zoonotic arenaviruses can lead to life-threating diseases in humans. These viruses encode a large (L) polymerase that transcribes and replicates the viral genome. At the late stage of replication, ...Zoonotic arenaviruses can lead to life-threating diseases in humans. These viruses encode a large (L) polymerase that transcribes and replicates the viral genome. At the late stage of replication, the multifunctional Z protein interacts with the L polymerase to shut down RNA synthesis and initiate virion assembly. However, the mechanism by which the Z protein regulates the activity of L polymerase is unclear. Here, we used cryo-electron microscopy to resolve the structures of both Lassa and Machupo virus L polymerases in complex with their cognate Z proteins, and viral RNA, to 3.1-3.9 Å resolutions. These structures reveal that Z protein binding induces conformational changes in two catalytic motifs of the L polymerase, and restrains their conformational dynamics to inhibit RNA synthesis, which is supported by hydrogen-deuterium exchange mass spectrometry analysis. Importantly, we show, by in vitro polymerase reactions, that Z proteins of Lassa and Machupo viruses can cross-inhibit their L polymerases, albeit with decreased inhibition efficiencies. This cross-reactivity results from a highly conserved determinant motif at the contacting interface, but is affected by other variable auxiliary motifs due to the divergent evolution of Old World and New World arenaviruses. These findings could provide promising targets for developing broad-spectrum antiviral drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31178.map.gz emd_31178.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31178-v30.xml emd-31178-v30.xml emd-31178.xml emd-31178.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31178_fsc.xml emd_31178_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31178.png emd_31178.png | 55.9 KB | ||
| Filedesc metadata |  emd-31178.cif.gz emd-31178.cif.gz | 7.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31178 http://ftp.pdbj.org/pub/emdb/structures/EMD-31178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31178 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31178_validation.pdf.gz emd_31178_validation.pdf.gz | 392.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31178_full_validation.pdf.gz emd_31178_full_validation.pdf.gz | 392.5 KB | 表示 | |
| XML形式データ |  emd_31178_validation.xml.gz emd_31178_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  emd_31178_validation.cif.gz emd_31178_validation.cif.gz | 13.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31178 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31178 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31178 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31178 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31178.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31178.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The EM map of Lassa L in complex with 3'-vRNA and Z mutant (F36A) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Lassa virus polymerase in complex with 3'-vRNA and Z mutant (F36A).
| 全体 | 名称: Lassa virus polymerase in complex with 3'-vRNA and Z mutant (F36A). |
|---|---|
| 要素 |
|
-超分子 #1: Lassa virus polymerase in complex with 3'-vRNA and Z mutant (F36A).
| 超分子 | 名称: Lassa virus polymerase in complex with 3'-vRNA and Z mutant (F36A). タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 270 KDa |
-超分子 #2: Lassa virus Z protein
| 超分子 | 名称: Lassa virus Z protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|
-超分子 #3: 3-'vRNA promoter
| 超分子 | 名称: 3-'vRNA promoter / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) / Synthetically produced: Yes Lassa mammarenavirus (ウイルス) / Synthetically produced: Yes |
-超分子 #4: Lassa virus polymerase
| 超分子 | 名称: Lassa virus polymerase / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|
-分子 #1: RING finger protein Z
| 分子 | 名称: RING finger protein Z / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 10.665365 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGNKQVKAPE ARNSPRASLI PDATHLGPQF CKSCWAENKG LVECNNHYLC LNCLTLLLGV SSRCPICKMP LPTRLRPSAA PTAPPAEAG DNTRPPPYSP UniProtKB: RING finger protein Z |
-分子 #3: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 253.505828 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) |
| 配列 | 文字列: MEEDIACVKD LVSKYLANNE RLSRQKLAFL VQTEPRMLLM EGLKLLSLCI EVDSCNANGC EHNSEDKSVE RILHDHGVLT PSLCFVVPD GYKLTGNVLI LLECFVRSSP ANFEQKYVED FKKLEQLKED LKSVDINLIP LIDGRTSFYN EQIPDWVNDK L RDTLFSLL ...文字列: MEEDIACVKD LVSKYLANNE RLSRQKLAFL VQTEPRMLLM EGLKLLSLCI EVDSCNANGC EHNSEDKSVE RILHDHGVLT PSLCFVVPD GYKLTGNVLI LLECFVRSSP ANFEQKYVED FKKLEQLKED LKSVDINLIP LIDGRTSFYN EQIPDWVNDK L RDTLFSLL KYAQESNSLF EESEYSRLCE SLSMTSGRLS GVESLNALLD NRSNHYEEVI ASCHQGINNK LTAHEVKLQI EE EYQVFRN RLRKGEIEGQ FLKVEKNQLL NEFNNLYADK VAEKDSVEHL THQFKRASPI LRFLYANISK GDNGEGNLII GEC QMQCWR SFLNKVKSMR ILNTRRKLLL IFDALILLAS KHDQVRKKPL RGWLGTCFVS VNDRLVSLES TKKDLKKWVE RRQQ VERSR TMQSFQCPSK NQILNSIFQK TISKATTALR DVGISVDHYK IDMEVICPDG YDLIMDFDVS GVTPTISYQR SEEEA FPYI MGDVDLLKTT DLERLSSLSL ALVNSMKTSS TVKLRQNEFG PARYQVVKCK EAYCQEFSLG ETEFQLIYQK TGECSK CYA INDNRVGEVC SFYADPKRYF PAIFSAEVLQ TTVSTMISWI EDCNELEEQL DKIRSLTKMI LILILAHPSK RSQKLLQ NL RYFIMAYVSD YYHKDLIDKV REELITDVEF LLYRLLRTLM GLVLSEDVKS MMTNRFKFIL NISYMCHFIT KETPDRLT D QIKCFEKFLE PKVKFGHVSI NPADTATEEE LDDMVYNAKK FLSKGGCTSA KGPSYKKPGV SKKYLSLLTS SFNNGSLFK EREVKKEIKD PLITSGCATA LDLASNKSVV VNKYTDGSRV LNYDFNKLTA LAVSQLTEVF SRKGKHLLNK QDYEYKVQQA MSNLVLGSK QHKGDADEAD LDEILLDGGA STYFNQLKET VEKIVDQYRE PVKMGSGSND GDQPSINDLD EIVSNKFYIR L IKGELSNH MVEDFDHDVL PDKFYEEFCD AVYENSKLKE KYFYCGHMSQ CPIGELTKAV STRTYLDHEY FQCFKSILLI MN ANALMGK YTHYKSRNLN FKFDMGKLSD DARISERESN SEALSKALSL TNCTTAMLKN LCFYSQESPQ SYNSVGPDTG RLK FSLSYK EQVGGNRELY IGDLRTKMFT RLIEDYFEAL SSQLSGSCLN NEKEFENAIL SMKLNVSMAH VSYSMDHSKW GPMM CPFLF LTVLQNLIFL SKDLQADIKG RDYLSTLLMW HMHKMVEIPF NVVSAMMKSF IKAQLGLRKK TKQSITEDFF YSNFQ IGVV PSHISSILDM GQGILHNTSD FYALITERFI NYAISCVCGG TIDAYTSSDD QISLFDQTLT ELLHRDPEEF RALMEF HYY MSDQLNKFVS PKSVIGRFVA EFKSRFFVWG DEVPLLTKFV AAALHNIKCK EPHQLAETID TIVDQSVANG VPVHLCN LI QIRTLSLLQY ARYPIDPFLL NCETDVRDWV DGNRSYRIMR QIEGLIPDAC SKIRSMLRRL YNRLKTGQLH EEFTTNYL S SEHLSSLKNL CELLGVEPPS ESDLEYSWLN LAAHHPLRMV LRQKIIYSGA VNLDDEKIPT IVKTIQNKLS STFTRGAQK LLSEAINKSA FQSSIASGFV GLCRTLGSKC VRGPNKENLY IKSIQSLITG TQGIELLTNS IGVQYWRVPL GLRNKSESVV SYFRPLLWD YMCISLSTAI ELGAWVLGDP KTTKALDFFK HNPCDYFPLK PTASKLLEDR VGLNHIIHSL RRLYPSVFEK H ILPFMSDL ASTKMKWSPR IKFLDLCVAL DVNCEALSLV SHIVKWKREE HYIVLSSELR FSHTRTHEPM VEERVVSTSD AV DNFMRQI YFESYVRPFV ATTRTLGSFT WFPHRTSIPE GEGLHRLGPF SSFVEKVIHK GVERPMFKHD LMMGYAWIDF DIE PARFNQ NQLIASGLVD SKFDSLEDFF DAVASLPTGS AKLSQTVRFR IKSQDASFRE SFAIHLDYIG SMNQQAKYLV HDVT AMYSG AVSPCVLSDC WRLVLSGPTF KGKPVWYVDT EVINEFLVDT NQLGHVTPVE VVVDMEKLQF AEYDFMLVGP CAEPV PLVV RRGGLWECEK KLASFTPVIQ DQDLEMFVRE VGDTSSDLLI RALSDMITDR LGLRMQWSGV DIVSTLRAAA PGNAEV LSA VLEVVDNWVE FKGYALCYSK SRGRVMVQSS SGKLRLKGRT CEELTEGGEH VEDIE UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: 3-'vRNA promoter
| 分子 | 名称: 3-'vRNA promoter / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 6.069649 KDa |
| 配列 | 文字列: GCCUAGGAUC CACUGUGCG |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #5: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: NICKEL/TITANIUM / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY 詳細: The film was designed with regular 1.2/1.3 micron holey partterns, similar to the Quantifoil 1.2/1.3 holey grids. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 3-18 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient | ||||||
| 得られたモデル |  PDB-7ela: |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)