+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2946 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of HET-s prion infectious form | |||||||||
 マップデータ マップデータ | Reconstruction of HET-s prion amyloid assembled at pH3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | amyloid / prion / cross-beta structure | |||||||||
| 機能・相同性 | Het-s prion-forming domain / Het-s prion-forming domain / Prion-inhibition and propagation, HeLo domain / HeLo domain superfamily / Het-s 218-289 / Prion-inhibition and propagation / identical protein binding / cytoplasm / Heterokaryon incompatibility protein s 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Podospora anserina (菌類) Podospora anserina (菌類) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 8.5 Å | |||||||||
 データ登録者 データ登録者 | Mizuno N / Baxa U / Steven AC | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2011 ジャーナル: Proc Natl Acad Sci U S A / 年: 2011タイトル: Structural dependence of HET-s amyloid fibril infectivity assessed by cryoelectron microscopy. 著者: Naoko Mizuno / Ulrich Baxa / Alasdair C Steven /  要旨: HET-s is a prion protein of the fungus Podospora anserina which, in the prion state, is active in a self/nonself recognition process called heterokaryon incompatibility. Its prionogenic properties ...HET-s is a prion protein of the fungus Podospora anserina which, in the prion state, is active in a self/nonself recognition process called heterokaryon incompatibility. Its prionogenic properties reside in the C-terminal "prion domain." The HET-s prion domain polymerizes in vitro into amyloid fibrils whose properties depend on the pH of assembly; above pH 3, infectious singlet fibrils are produced, and below pH 3, noninfectious triplet fibrils. To investigate the correlation between structure and infectivity, we performed cryo-EM analyses. Singlet fibrils have a helical pitch of approximately 410 Å and a left-handed twist. Triplet fibrils have three protofibrils whose lateral dimensions (36 × 25 Å) and axial packing (one subunit per 9.4 Å) match those of singlets but differ in their supercoiling. At 8.5-Å resolution, the cross-section of the singlet fibril reconstruction is largely consistent with that of a β-solenoid model previously determined by solid-state NMR. Reconstructions of the triplet fibrils show three protofibrils coiling around a common axis and packed less tightly at pH 3 than at pH 2, eventually peeling off. Taken together with the earlier observation that fragmentation of triplet fibrils by sonication does not increase infectivity, these observations suggest a novel mechanism for self-propagation, whereby daughter fibrils nucleate on the lateral surface of singlet fibrils. In triplets, this surface is occluded, blocking nucleation and thereby explaining their lack of infectivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2946.map.gz emd_2946.map.gz | 548.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2946-v30.xml emd-2946-v30.xml emd-2946.xml emd-2946.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  2946-HET-s.tif 2946-HET-s.tif | 62.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2946 http://ftp.pdbj.org/pub/emdb/structures/EMD-2946 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2946 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2946 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2946_validation.pdf.gz emd_2946_validation.pdf.gz | 173.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2946_full_validation.pdf.gz emd_2946_full_validation.pdf.gz | 172.6 KB | 表示 | |
| XML形式データ |  emd_2946_validation.xml.gz emd_2946_validation.xml.gz | 4.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2946 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2946 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2946 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2946 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2946.map.gz / 形式: CCP4 / 大きさ: 708 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2946.map.gz / 形式: CCP4 / 大きさ: 708 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of HET-s prion amyloid assembled at pH3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
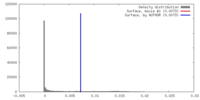
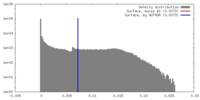
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HET-s prion domain (218-289) assembled at pH3
| 全体 | 名称: HET-s prion domain (218-289) assembled at pH3 |
|---|---|
| 要素 |
|
-超分子 #1000: HET-s prion domain (218-289) assembled at pH3
| 超分子 | 名称: HET-s prion domain (218-289) assembled at pH3 / タイプ: sample / ID: 1000 集合状態: helical polymer with a rise of 9.4 A per protein unit Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 8 KDa |
-分子 #1: Heterokaryon incompatibility protein s
| 分子 | 名称: Heterokaryon incompatibility protein s / タイプ: protein_or_peptide / ID: 1 / Name.synonym: HET-s 詳細: HET-s proteins are assembled to make a single strand helix, with a rise of 9.4 A per protein. 集合状態: Helical filament / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Podospora anserina (菌類) Podospora anserina (菌類) |
| 分子量 | 実験値: 8 KDa / 理論値: 8 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Heterokaryon incompatibility protein s / InterPro: Het-s prion-forming domain |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 3 / 詳細: 20 mM citrate acid |
| グリッド | 詳細: Glow discharged Quantifoil R2/2 was used. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: FEI VITROBOT MARK II / 手法: Blot for 5 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 最低: 90 K / 最高: 100 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 120,000 times magnification |
| 日付 | 2008年4月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 100 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | The reconstruction was performed using SPIDER in combination with IHRSR. |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 18.8 Å 想定した対称性 - らせんパラメータ - ΔΦ: 17.1 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 8.5 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIder, EMAN, IHRSR, bsoft |
| CTF補正 | 詳細: Each micrograph |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)