+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2880 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of the human mitochondrial ribosome (small subunit, masked) | |||||||||
 マップデータ マップデータ | Small subunit of the human mitochondrial ribosome (masked). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mitochondrial ribosome / 55S | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Amunts A / Brown A / Toots J / Scheres SH / Ramakrishnan V | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2015 ジャーナル: Science / 年: 2015タイトル: Ribosome. The structure of the human mitochondrial ribosome. 著者: Alexey Amunts / Alan Brown / Jaan Toots / Sjors H W Scheres / V Ramakrishnan /  要旨: The highly divergent ribosomes of human mitochondria (mitoribosomes) synthesize 13 essential proteins of oxidative phosphorylation complexes. We have determined the structure of the intact ...The highly divergent ribosomes of human mitochondria (mitoribosomes) synthesize 13 essential proteins of oxidative phosphorylation complexes. We have determined the structure of the intact mitoribosome to 3.5 angstrom resolution by means of single-particle electron cryogenic microscopy. It reveals 80 extensively interconnected proteins, 36 of which are specific to mitochondria, and three ribosomal RNA molecules. The head domain of the small subunit, particularly the messenger (mRNA) channel, is highly remodeled. Many intersubunit bridges are specific to the mitoribosome, which adopts conformations involving ratcheting or rolling of the small subunit that are distinct from those seen in bacteria or eukaryotes. An intrinsic guanosine triphosphatase mediates a contact between the head and central protuberance. The structure provides a reference for analysis of mutations that cause severe pathologies and for future drug design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2880.map.gz emd_2880.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2880-v30.xml emd-2880-v30.xml emd-2880.xml emd-2880.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2880_fsc.xml emd_2880_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  EMD-2880.png EMD-2880.png | 122.5 KB | ||
| その他 |  emd_2880_half_map_1.map.gz emd_2880_half_map_1.map.gz emd_2880_half_map_2.map.gz emd_2880_half_map_2.map.gz | 107 MB 107 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2880 http://ftp.pdbj.org/pub/emdb/structures/EMD-2880 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2880 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2880 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2880_validation.pdf.gz emd_2880_validation.pdf.gz | 252.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2880_full_validation.pdf.gz emd_2880_full_validation.pdf.gz | 251.6 KB | 表示 | |
| XML形式データ |  emd_2880_validation.xml.gz emd_2880_validation.xml.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2880 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2880 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2880 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2880 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2880.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2880.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Small subunit of the human mitochondrial ribosome (masked). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
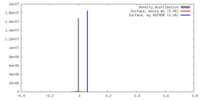
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 2880 half map 1.map
| ファイル | emd_2880_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
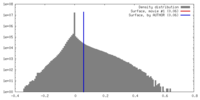
| 密度ヒストグラム |
-添付マップデータ: emd 2880 half map 2.map
| ファイル | emd_2880_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
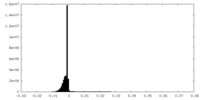
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human mitochondrial ribosome
| 全体 | 名称: Human mitochondrial ribosome |
|---|---|
| 要素 |
|
-超分子 #1000: Human mitochondrial ribosome
| 超分子 | 名称: Human mitochondrial ribosome / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: Multimer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1.0 MDa / 理論値: 1.0 MDa / 手法: Sedimentation |
-超分子 #1: Small subunit of the human mitochondrial ribosome
| 超分子 | 名称: Small subunit of the human mitochondrial ribosome / タイプ: complex / ID: 1 / Name.synonym: 28S subunit / 組換発現: No / Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: human / 組織: kidney / 細胞: HEK 293 / Organelle: mitochondria / 細胞中の位置: Inner mitochondrial membrane Homo sapiens (ヒト) / 別称: human / 組織: kidney / 細胞: HEK 293 / Organelle: mitochondria / 細胞中の位置: Inner mitochondrial membrane |
| 分子量 | 実験値: 1.0 MDa / 理論値: 1.0 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.23 mg/mL |
|---|---|
| 緩衝液 | pH: 7.45 詳細: 20 mM Hepes-KOH pH 7.45, 100 mM KCl, 20 mM MgOAc, 2 mM DTT |
| グリッド | 詳細: 30 s on glow-discharged holey carbon grids (Quantifoil R2/2), onto which a home-made continuous carbon film |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 90 K / 装置: FEI VITROBOT MARK II / 手法: Blot 2.5 seconds before plunging |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 80 K / 最高: 90 K / 平均: 85 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 59,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2014年4月3日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 7526 / 平均電子線量: 25 e/Å2 詳細: Every image is the average of 17 frames recorded by the direct electron detector |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 104478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| Microscopy ID | 2 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 80 K / 最高: 90 K / 平均: 85 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 59,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2014年4月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 7526 / 平均電子線量: 25 e/Å2 詳細: Every image is the average of 17 frames recorded by the direct electron detector |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 104478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #3
電子顕微鏡法 #3
| Microscopy ID | 3 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 80 K / 最高: 90 K / 平均: 85 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 59,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2014年5月10日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 7526 / 平均電子線量: 25 e/Å2 詳細: Every image is the average of 17 frames recorded by the direct electron detector |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 104478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #4
電子顕微鏡法 #4
| Microscopy ID | 4 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 80 K / 最高: 90 K / 平均: 85 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 59,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2014年5月30日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 7526 / 平均電子線量: 25 e/Å2 詳細: Every image is the average of 17 frames recorded by the direct electron detector |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 104478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)