+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-25905 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of W6 possum enterovirus | |||||||||
 マップデータ マップデータ | Enterovirus EV-F4 - isolated from Bushtail Possum | |||||||||
 試料 試料 | Enterovirus != Possum enterovirus W6 Enterovirus
| |||||||||
 キーワード キーワード | enterovirus / icosahedral / VIRUS | |||||||||
| 生物種 |  Possum enterovirus W6 (エンテロウイルス) Possum enterovirus W6 (エンテロウイルス) | |||||||||
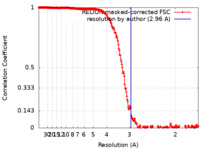
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.96 Å | |||||||||
 データ登録者 データ登録者 | Wang I / Jayawardena N | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022タイトル: Cryo-EM Structure of a Possum Enterovirus. 著者: Ivy Wang / Sandeep K Gupta / Guillaume Ems / Nadishka Jayawardena / Mike Strauss / Mihnea Bostina /     要旨: Enteroviruses (EVs) represent a substantial concern to global health. Here, we present the cryo-EM structure of a non-human enterovirus, EV-F4, isolated from the Australian brushtail possum to assess ...Enteroviruses (EVs) represent a substantial concern to global health. Here, we present the cryo-EM structure of a non-human enterovirus, EV-F4, isolated from the Australian brushtail possum to assess the structural diversity of these picornaviruses. The capsid structure, determined to ~3 Å resolution by single particle analysis, exhibits a largely smooth surface, similar to EV-F3 (formerly BEV-2). Although the cellular receptor is not known, the absence of charged residues on the outer surface of the canyon suggest a different receptor type than for EV-F3. Density for the pocket factor is clear, with the entrance to the pocket being smaller than for other enteroviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25905.map.gz emd_25905.map.gz | 920.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25905-v30.xml emd-25905-v30.xml emd-25905.xml emd-25905.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25905_fsc.xml emd_25905_fsc.xml | 22.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25905.png emd_25905.png | 117.4 KB | ||
| Filedesc metadata |  emd-25905.cif.gz emd-25905.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25905 http://ftp.pdbj.org/pub/emdb/structures/EMD-25905 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25905 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25905 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25905_validation.pdf.gz emd_25905_validation.pdf.gz | 753.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25905_full_validation.pdf.gz emd_25905_full_validation.pdf.gz | 753 KB | 表示 | |
| XML形式データ |  emd_25905_validation.xml.gz emd_25905_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_25905_validation.cif.gz emd_25905_validation.cif.gz | 25.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25905 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25905 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25905 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25905 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25905.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25905.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Enterovirus EV-F4 - isolated from Bushtail Possum | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.857 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus
| 全体 | 名称:  Enterovirus (エンテロウイルス) Enterovirus (エンテロウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Possum enterovirus W6
| 超分子 | 名称: Possum enterovirus W6 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 263532 / 生物種: Possum enterovirus W6 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Possum enterovirus W6 (エンテロウイルス) Possum enterovirus W6 (エンテロウイルス) |
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Possum enterovirus W6 (エンテロウイルス) Possum enterovirus W6 (エンテロウイルス) |
| 分子量 | 理論値: 30.311832 KDa |
| 配列 | 文字列: GETGQLIKDT IKNTVENTVE STHSISTEAT PALQAAETGA TSNASDESMI ETRNVVNTHG VAETSLEGFY GRAGLVAMFT TEGGIRSWY INFGKYVQLR AKLELLTYAR FDVEFTIVAQ VVDEQAKVKD FNVDYQVMYV PPGASAPDGQ DSFQWQSSCN P SVISNTGL ...文字列: GETGQLIKDT IKNTVENTVE STHSISTEAT PALQAAETGA TSNASDESMI ETRNVVNTHG VAETSLEGFY GRAGLVAMFT TEGGIRSWY INFGKYVQLR AKLELLTYAR FDVEFTIVAQ VVDEQAKVKD FNVDYQVMYV PPGASAPDGQ DSFQWQSSCN P SVISNTGL PPARVSVPFM SSANAYSFSY DGYIQFGDTS SSSYGILPIH YLGQLVVRTC EDLDSARLRV RIYAKPKHMR GW IPRSPRM RPYVSRFTGI YTDAPSFCVN RESITTAG |
-分子 #2: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Possum enterovirus W6 (エンテロウイルス) Possum enterovirus W6 (エンテロウイルス) |
| 分子量 | 理論値: 26.577887 KDa |
| 配列 | 文字列: SAEACGYSDR VAQLTLGNST ITTQEAANIV VAYGRWPSGL RDTDATAVDK PTQPGVSAER FYTLPSVQWT TSFTGHYWKL PDALSDLGL FGQNLQFHYL YRGGWAIHVQ CNATKFHQGT LLVVAVPEHK IQAQSNPSFG RTNPGEAGAA CQFPFTFEDG T ALGNALIY ...文字列: SAEACGYSDR VAQLTLGNST ITTQEAANIV VAYGRWPSGL RDTDATAVDK PTQPGVSAER FYTLPSVQWT TSFTGHYWKL PDALSDLGL FGQNLQFHYL YRGGWAIHVQ CNATKFHQGT LLVVAVPEHK IQAQSNPSFG RTNPGEAGAA CQFPFTFEDG T ALGNALIY PHQWINLRTN NSATLVLPYV NAIPMDSGIK HNNWTLLVIP IVPLEYAVGA TTFVPITVTI APMCTEYNGL RA AVTQ |
-分子 #3: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Possum enterovirus W6 (エンテロウイルス) Possum enterovirus W6 (エンテロウイルス) |
| 分子量 | 理論値: 26.997758 KDa |
| 配列 | 文字列: GCPTLYTPGS GQFLTTDDFQ TPCMLPKFQP TPVIDIPGEV KNFLEVIQVE SLVEINNVSG VEGVARYRIP LNVQDAMDGQ IMAVRVDPG ADGPMQSTLL GVFTRYYTQW SGSLDFTFMF CGTFMTTGKV IIAYTPPGGD QPGSRQQAML GTHVVWDFGL Q SSITLVVP ...文字列: GCPTLYTPGS GQFLTTDDFQ TPCMLPKFQP TPVIDIPGEV KNFLEVIQVE SLVEINNVSG VEGVARYRIP LNVQDAMDGQ IMAVRVDPG ADGPMQSTLL GVFTRYYTQW SGSLDFTFMF CGTFMTTGKV IIAYTPPGGD QPGSRQQAML GTHVVWDFGL Q SSITLVVP WISSGHFRGT SLDNTIYKYR YYEAGYITMW YQTNMVVPPN FPTEASILMF VAAQPNFSLR ILKDRPDITQ VA SLQ |
-分子 #4: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Possum enterovirus W6 (エンテロウイルス) Possum enterovirus W6 (エンテロウイルス) |
| 分子量 | 理論値: 7.63234 KDa |
| 配列 | 文字列: MGAQLSKNTA GSHTTGTYAT GGSSIHYTNI NYYENAASNS LNKQDFTQDP DKFTRPVADI MKEAAVPLKS P |
-分子 #5: SPHINGOSINE
| 分子 | 名称: SPHINGOSINE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SPH |
|---|---|
| 分子量 | 理論値: 299.492 Da |
| Chemical component information |  ChemComp-SPH: |
-分子 #6: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 21 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 1.37 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)