+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24683 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the Sodium-driven Chloride/Bicarbonate Exchanger NDCBE (SLC4A8) | |||||||||
 マップデータ マップデータ | postprocess 3d map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NDCBE / sodium-driven chloride/bicarbonate exchanger / cryo-EM / MEMBRANE PROTEIN | |||||||||
| 生物種 |  | |||||||||
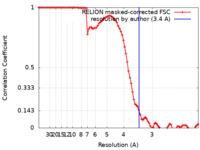
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Wang WG / Tsirulnikov K | |||||||||
| 資金援助 |  米国, 米国,  カナダ, 2件 カナダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Cryo-EM structure of the sodium-driven chloride/bicarbonate exchanger NDCBE. 著者: Weiguang Wang / Kirill Tsirulnikov / Hristina R Zhekova / Gülru Kayık / Hanif Muhammad Khan / Rustam Azimov / Natalia Abuladze / Liyo Kao / Debbie Newman / Sergei Yu Noskov / Z Hong Zhou / ...著者: Weiguang Wang / Kirill Tsirulnikov / Hristina R Zhekova / Gülru Kayık / Hanif Muhammad Khan / Rustam Azimov / Natalia Abuladze / Liyo Kao / Debbie Newman / Sergei Yu Noskov / Z Hong Zhou / Alexander Pushkin / Ira Kurtz /   要旨: SLC4 transporters play significant roles in pH regulation and cellular sodium transport. The previously solved structures of the outward facing (OF) conformation for AE1 (SLC4A1) and NBCe1 (SLC4A4) ...SLC4 transporters play significant roles in pH regulation and cellular sodium transport. The previously solved structures of the outward facing (OF) conformation for AE1 (SLC4A1) and NBCe1 (SLC4A4) transporters revealed an identical overall fold despite their different transport modes (chloride/bicarbonate exchange versus sodium-carbonate cotransport). However, the exact mechanism determining the different transport modes in the SLC4 family remains unknown. In this work, we report the cryo-EM 3.4 Å structure of the OF conformation of NDCBE (SLC4A8), which shares transport properties with both AE1 and NBCe1 by mediating the electroneutral exchange of sodium-carbonate with chloride. This structure features a fully resolved extracellular loop 3 and well-defined densities corresponding to sodium and carbonate ions in the tentative substrate binding pocket. Further, we combine computational modeling with functional studies to unravel the molecular determinants involved in NDCBE and SLC4 transport. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24683.map.gz emd_24683.map.gz | 54.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24683-v30.xml emd-24683-v30.xml emd-24683.xml emd-24683.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24683_fsc.xml emd_24683_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24683.png emd_24683.png | 104.4 KB | ||
| Filedesc metadata |  emd-24683.cif.gz emd-24683.cif.gz | 6.6 KB | ||
| その他 |  emd_24683_additional_1.map.gz emd_24683_additional_1.map.gz emd_24683_half_map_1.map.gz emd_24683_half_map_1.map.gz emd_24683_half_map_2.map.gz emd_24683_half_map_2.map.gz | 54.5 MB 43.9 MB 43.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24683 http://ftp.pdbj.org/pub/emdb/structures/EMD-24683 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24683 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24683 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24683_validation.pdf.gz emd_24683_validation.pdf.gz | 813.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24683_full_validation.pdf.gz emd_24683_full_validation.pdf.gz | 813.3 KB | 表示 | |
| XML形式データ |  emd_24683_validation.xml.gz emd_24683_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_24683_validation.cif.gz emd_24683_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24683 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24683 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24683 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24683 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24683.map.gz / 形式: CCP4 / 大きさ: 58.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24683.map.gz / 形式: CCP4 / 大きさ: 58.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocess 3d map. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
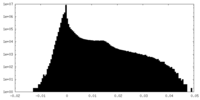
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: postprocess 3d map.
| ファイル | emd_24683_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocess 3d map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
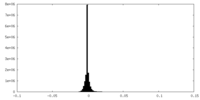
| 密度ヒストグラム |
-ハーフマップ: half map1.
| ファイル | emd_24683_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
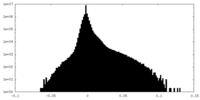
| 密度ヒストグラム |
-ハーフマップ: half map2.
| ファイル | emd_24683_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
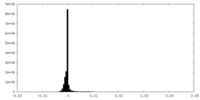
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NDCBE(SLC4A8) dimer
| 全体 | 名称: NDCBE(SLC4A8) dimer |
|---|---|
| 要素 |
|
-超分子 #1: NDCBE(SLC4A8) dimer
| 超分子 | 名称: NDCBE(SLC4A8) dimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Electroneutral sodium bicarbonate exchanger 1
| 分子 | 名称: Electroneutral sodium bicarbonate exchanger 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 64.304094 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LFGGLVLDVK RKAPWYWSDY RDALSLQCLA SFLFLYCACM SPVITFGGLL GEATEGRISA IESLFGASMT GIAYSLFAGQ PLTILGSTG PVLVFEKILF KFCKDYALSY LSLRACIGLW TAFLCIVLVA TDASSLVCYI TRFTEEAFAS LICIIFIYEA I EKLIHLAE ...文字列: LFGGLVLDVK RKAPWYWSDY RDALSLQCLA SFLFLYCACM SPVITFGGLL GEATEGRISA IESLFGASMT GIAYSLFAGQ PLTILGSTG PVLVFEKILF KFCKDYALSY LSLRACIGLW TAFLCIVLVA TDASSLVCYI TRFTEEAFAS LICIIFIYEA I EKLIHLAE TYPIHMHSQL DHLSLYYCRC ALPENPNNHT LQYWKEHSIP TADVNWANLT VSECQEMHGE FIGSACGHHG PY TPDVLFW SCILFFATFI VSSTLKTFKT SRYFPTRVRS TVSDFAVFLT IFTMVILDFL IGVPSPKLQV PSVFKPTRDD RGW FISPIG PNPWWTVIAA IIPALLCTIL IFMDQQITAV IINRKEHKLK KGCGYHLDLL VVAIMLGVCS LMGLPWFVAA TVLS ITHVN SLKLESECSA PGEQPKFLGI REQRVTGLMI FVLMGCSVFM TAVLKFIPMP VLYGVFLYMG VSSLQGIQFF DRLKL FGMP AKHQPDFIYL RHVPLRKVHL FTLVQLTCLV LLWVIKASPA AIVFPMMVLA LVFVRKVMDL CFSKRELSWL DDLMPE SKK KKLDDAKK |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #5: CARBONATE ION
| 分子 | 名称: CARBONATE ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: CO3 |
|---|---|
| 分子量 | 理論値: 60.009 Da |
| Chemical component information |  ChemComp-CO3: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER / 詳細: Glow discharged with Easyglow. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot time 2 seconds. blot force 0. | ||||||||||||
| 詳細 | sample in 0.01% LMNG. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 2 / 実像数: 7545 / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 130000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7rtm: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)