登録情報 データベース : EMDB / ID : EMD-23916タイトル The HER2/HER3/NRG1b Heterodimer Main map from non-uniform refinement in cryosparc, filtered to 2.92A in resolution and sharpened with a b-factor of 94.3. 複合体 : HER2/HER3/NRG1b Heterocomplex in the context of near full-length receptors複合体 : HER3タンパク質・ペプチド : Receptor tyrosine-protein kinase erbB-3複合体 : HER2-MBP Fusionタンパク質・ペプチド : Receptor tyrosine-protein kinase erbB-2,Maltose/maltodextrin-binding periplasmic protein複合体 : neuregulin-1タンパク質・ペプチド : Isoform 6 of Pro-neuregulin-1, membrane-bound isoformリガンド : 2-acetamido-2-deoxy-beta-D-glucopyranose / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
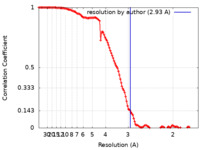
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / Escherichia coli (大腸菌)手法 / / 解像度 : 2.93 Å Diwanji D / Trenker R 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) National Institutes of Health/National Cancer Institute (NIH/NCI) 1F30CA247147 German Research Foundation (DFG) TR 1668/1-1
ジャーナル : Nature / 年 : 2021タイトル : Structures of the HER2-HER3-NRG1β complex reveal a dynamic dimer interface.著者 : Devan Diwanji / Raphael Trenker / Tarjani M Thaker / Feng Wang / David A Agard / Kliment A Verba / Natalia Jura / 要旨 : Human epidermal growth factor receptor 2 (HER2) and HER3 form a potent pro-oncogenic heterocomplex upon binding of growth factor neuregulin-1β (NRG1β). The mechanism by which HER2 and HER3 interact ... Human epidermal growth factor receptor 2 (HER2) and HER3 form a potent pro-oncogenic heterocomplex upon binding of growth factor neuregulin-1β (NRG1β). The mechanism by which HER2 and HER3 interact remains unknown in the absence of any structures of the complex. Here we isolated the NRG1β-bound near full-length HER2-HER3 dimer and, using cryo-electron microscopy, reconstructed the extracellulardomain module, revealing unexpected dynamics at the HER2-HER3 dimerization interface. We show that the dimerization arm of NRG1β-bound HER3 is unresolved because the apo HER2 monomer does not undergo a ligand-induced conformational change needed to establish a HER3 dimerization arm-binding pocket. In a structure of the oncogenic extracellular domain mutant HER2(S310F), we observe a compensatory interaction with the HER3 dimerization arm that stabilizes the dimerization interface. Both HER2-HER3 and HER2(S310F)-HER3 retain the capacity to bind to the HER2-directed therapeutic antibody trastuzumab, but the mutant complex does not bind to pertuzumab. Our structure of the HER2(S310F)-HER3-NRG1β-trastuzumab Fab complex reveals that the receptor dimer undergoes a conformational change to accommodate trastuzumab. Thus, similar to oncogenic mutations, therapeutic agents exploit the intrinsic dynamics of the HER2-HER3 heterodimer. The unique features of a singly liganded HER2-HER3 heterodimer underscore the allosteric sensing of ligand occupancy by the dimerization interface and explain why extracellular domains of HER2 do not homo-associate via a canonical active dimer interface. 履歴 登録 2021年4月30日 - ヘッダ(付随情報) 公開 2021年10月27日 - マップ公開 2021年10月27日 - 更新 2024年11月13日 - 現状 2024年11月13日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) / 
 データ登録者
データ登録者 米国,
米国,  ドイツ, 3件
ドイツ, 3件  引用
引用 ジャーナル: Nature / 年: 2021
ジャーナル: Nature / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_23916.map.gz
emd_23916.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-23916-v30.xml
emd-23916-v30.xml emd-23916.xml
emd-23916.xml EMDBヘッダ
EMDBヘッダ emd_23916_fsc.xml
emd_23916_fsc.xml FSCデータファイル
FSCデータファイル emd_23916.png
emd_23916.png emd_23916_msk_1.map
emd_23916_msk_1.map マスクマップ
マスクマップ emd-23916.cif.gz
emd-23916.cif.gz emd_23916_half_map_1.map.gz
emd_23916_half_map_1.map.gz emd_23916_half_map_2.map.gz
emd_23916_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-23916
http://ftp.pdbj.org/pub/emdb/structures/EMD-23916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23916
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23916 emd_23916_validation.pdf.gz
emd_23916_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_23916_full_validation.pdf.gz
emd_23916_full_validation.pdf.gz emd_23916_validation.xml.gz
emd_23916_validation.xml.gz emd_23916_validation.cif.gz
emd_23916_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23916
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23916 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23916
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23916 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_23916.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_23916.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_23916_msk_1.map
emd_23916_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)

 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)