+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23845 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Half integration complex of Cas4/Cas1/Cas2 and Cas4 still in the Non-PAM side | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR/Cas / Cas4 / PAM recognition / half integration / HYDROLASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報5' to 3' exodeoxyribonuclease (nucleoside 3'-phosphate-forming) / CRISPR-cas system / exonuclease activity / maintenance of CRISPR repeat elements / RNA endonuclease activity / 4 iron, 4 sulfur cluster binding / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Geobacter sulfurreducens (バクテリア) / Geobacter sulfurreducens Geobacter sulfurreducens (バクテリア) / Geobacter sulfurreducens | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.8 Å | |||||||||
 データ登録者 データ登録者 | Hu CY / Ke AK | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Mechanism for Cas4-assisted directional spacer acquisition in CRISPR-Cas. 著者: Chunyi Hu / Cristóbal Almendros / Ki Hyun Nam / Ana Rita Costa / Jochem N A Vink / Anna C Haagsma / Saket R Bagde / Stan J J Brouns / Ailong Ke /    要旨: Prokaryotes adapt to challenges from mobile genetic elements by integrating spacers derived from foreign DNA in the CRISPR array. Spacer insertion is carried out by the Cas1-Cas2 integrase complex. A ...Prokaryotes adapt to challenges from mobile genetic elements by integrating spacers derived from foreign DNA in the CRISPR array. Spacer insertion is carried out by the Cas1-Cas2 integrase complex. A substantial fraction of CRISPR-Cas systems use a Fe-S cluster containing Cas4 nuclease to ensure that spacers are acquired from DNA flanked by a protospacer adjacent motif (PAM) and inserted into the CRISPR array unidirectionally, so that the transcribed CRISPR RNA can guide target searching in a PAM-dependent manner. Here we provide a high-resolution mechanistic explanation for the Cas4-assisted PAM selection, spacer biogenesis and directional integration by type I-G CRISPR in Geobacter sulfurreducens, in which Cas4 is naturally fused with Cas1, forming Cas4/Cas1. During biogenesis, only DNA duplexes possessing a PAM-embedded 3'-overhang trigger Cas4/Cas1-Cas2 assembly. During this process, the PAM overhang is specifically recognized and sequestered, but is not cleaved by Cas4. This 'molecular constipation' prevents the PAM-side prespacer from participating in integration. Lacking such sequestration, the non-PAM overhang is trimmed by host nucleases and integrated to the leader-side CRISPR repeat. Half-integration subsequently triggers PAM cleavage and Cas4 dissociation, allowing spacer-side integration. Overall, the intricate molecular interaction between Cas4 and Cas1-Cas2 selects PAM-containing prespacers for integration and couples the timing of PAM processing with the stepwise integration to establish directionality. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23845.map.gz emd_23845.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23845-v30.xml emd-23845-v30.xml emd-23845.xml emd-23845.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23845.png emd_23845.png | 82.1 KB | ||
| Filedesc metadata |  emd-23845.cif.gz emd-23845.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23845 http://ftp.pdbj.org/pub/emdb/structures/EMD-23845 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23845 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23845 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23845_validation.pdf.gz emd_23845_validation.pdf.gz | 413.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23845_full_validation.pdf.gz emd_23845_full_validation.pdf.gz | 412.8 KB | 表示 | |
| XML形式データ |  emd_23845_validation.xml.gz emd_23845_validation.xml.gz | 5.4 KB | 表示 | |
| CIF形式データ |  emd_23845_validation.cif.gz emd_23845_validation.cif.gz | 6.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23845 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23845 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23845 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23845 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23845.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23845.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.18625 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
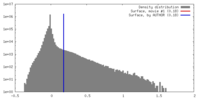
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Half integration complex of Cas4/Cas1/Cas2 and Cas4 still in the ...
| 全体 | 名称: Half integration complex of Cas4/Cas1/Cas2 and Cas4 still in the PAM side |
|---|---|
| 要素 |
|
-超分子 #1: Half integration complex of Cas4/Cas1/Cas2 and Cas4 still in the ...
| 超分子 | 名称: Half integration complex of Cas4/Cas1/Cas2 and Cas4 still in the PAM side タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 / 詳細: half integration in cas4 containing system |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 300 KDa |
-分子 #1: DNA (31-MER)
| 分子 | 名称: DNA (31-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 9.510105 KDa |
| 配列 | 文字列: (DG)(DT)(DC)(DG)(DT)(DA)(DG)(DC)(DT)(DG) (DA)(DG)(DG)(DC)(DC)(DT)(DC)(DA)(DG)(DC) (DT)(DA)(DC)(DG)(DA)(DC)(DT)(DT)(DT) (DT)(DT) |
-分子 #4: DNA (64-MER)
| 分子 | 名称: DNA (64-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 19.659529 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(DT)(DG)(DC)(DC)(DG)(DT)(DC) (DC)(DG)(DT)(DA)(DA)(DC)(DG)(DT)(DT)(DG) (DT)(DC)(DG)(DA)(DT)(DT)(DT)(DT)(DT) (DG)(DT)(DA)(DT)(DT)(DC)(DC)(DG)(DG)(DG) (DG) (DC)(DC)(DA)(DT)(DG) ...文字列: (DC)(DT)(DG)(DT)(DG)(DC)(DC)(DG)(DT)(DC) (DC)(DG)(DT)(DA)(DA)(DC)(DG)(DT)(DT)(DG) (DT)(DC)(DG)(DA)(DT)(DT)(DT)(DT)(DT) (DG)(DT)(DA)(DT)(DT)(DC)(DC)(DG)(DG)(DG) (DG) (DC)(DC)(DA)(DT)(DG)(DA)(DT)(DG) (DC)(DC)(DC)(DC)(DG)(DG)(DC)(DC)(DT)(DC) (DA)(DT) (DT)(DG)(DA)(DA) |
-分子 #5: DNA (5'-D(P*CP*GP*GP*AP*AP*AP*AP*GP*AP*GP*CP*C)-3')
| 分子 | 名称: DNA (5'-D(P*CP*GP*GP*AP*AP*AP*AP*GP*AP*GP*CP*C)-3') / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 3.705445 KDa |
| 配列 | 文字列: (DC)(DG)(DG)(DA)(DA)(DA)(DA)(DG)(DA)(DG) (DC)(DC) |
-分子 #6: DNA (45-MER)
| 分子 | 名称: DNA (45-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: Geobacter sulfurreducens |
| 分子量 | 理論値: 13.855857 KDa |
| 配列 | 文字列: (DC)(DA)(DA)(DT)(DG)(DA)(DG)(DG)(DC)(DC) (DG)(DG)(DG)(DG)(DC)(DA)(DT)(DC)(DA)(DT) (DG)(DG)(DC)(DC)(DC)(DC)(DG)(DG)(DA) (DA)(DT)(DA)(DC)(DG)(DG)(DC)(DT)(DC)(DT) (DT) (DT)(DT)(DC)(DC)(DG) |
-分子 #2: CRISPR-associated exonuclease Cas4/endonuclease Cas1 fusion
| 分子 | 名称: CRISPR-associated exonuclease Cas4/endonuclease Cas1 fusion タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) / 株: ATCC 51573 / DSM 12127 / PCA Geobacter sulfurreducens (バクテリア) / 株: ATCC 51573 / DSM 12127 / PCA |
| 分子量 | 理論値: 62.598496 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAETDGSIPL IPVRMLNEHV YCPRLAYLMW VQGEFSHNEF TVDGVIRHRR VDAGGGVLPS ETQEDSRIHA RSVSLSSERL GITAKIDLV EGEGAYVSPV DYKRGKRPHV AGGAYEPERV QLCAQGLLLR EHGFASDGGA LYFVASRERV PVAFDDELIG R TLAAIDEM ...文字列: MAETDGSIPL IPVRMLNEHV YCPRLAYLMW VQGEFSHNEF TVDGVIRHRR VDAGGGVLPS ETQEDSRIHA RSVSLSSERL GITAKIDLV EGEGAYVSPV DYKRGKRPHV AGGAYEPERV QLCAQGLLLR EHGFASDGGA LYFVASRERV PVAFDDELIG R TLAAIDEM GRTALSGTMP PPLEDSPKCP RCSLVGICLP DEVRFLSHLS VEPRPIIPAD GRGLPLYVQS PKAYVRKDGD CL VIEEERV RVAEARLGET SQVALFGNAT LTTAALHECL RREIPVTWLS YGGWFMGHTV STGHRNVETR TYQYQRSFDP ETC LNLARR WIVAKIANCR TLLRRNWRGE GDEAKAPPGL LMSLQDDMRH AMRAPSLEVL LGIEGASAGR YFQHFSRMLR GGDG EGMGF DFTTRNRRPP KDPVNALLSF AYAMLTREWT VALAAVGLDP YRGFYHQPRF GRPALALDMM EPFRPLIADS TVLMA INNG EIRTGDFVRS AGGCNLTDSA RKRFIAGFER RMEQEVTHPI FKYTISYRRL LEVQARLLTR YLSGEIPAYP NFVTR UniProtKB: CRISPR-associated exonuclease Cas4/endonuclease Cas1 fusion |
-分子 #3: CRISPR-associated endoribonuclease Cas2
| 分子 | 名称: CRISPR-associated endoribonuclease Cas2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) / 株: ATCC 51573 / DSM 12127 / PCA Geobacter sulfurreducens (バクテリア) / 株: ATCC 51573 / DSM 12127 / PCA |
| 分子量 | 理論値: 11.190176 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEHLYIVSYD IRNQRRWRRL FKTMHGFGCW LQLSVFQCRL DRIRIIKMEA AINEIVNHAE DHVLILDLGP AENVKPKVSS IGKTFDPIL RQAVIV UniProtKB: CRISPR-associated endoribonuclease Cas2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 150.0 mM / 構成要素 - 式: NaCl / 構成要素 - 名称: sodium chloride / 詳細: with 5 mM DTT |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: 6 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 1200 / 平均露光時間: 0.35 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / Cs: 2.7 mm / 最小 デフォーカス(公称値): 1.5 µm |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7mib: |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)