+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23519 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer | |||||||||
 マップデータ マップデータ | Cryo-EM map of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Fab-dimerized / glycan-reactive / antibodies / HIV-1 / DH898.1 / Fab-dimer / glycan-reactive neutralizing antibody / macaque HIV-1 vaccine-induced / B cell lineage / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane fusion involved in viral entry into host cell / symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope ...membrane fusion involved in viral entry into host cell / symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Simian-Human immunodeficiency virus (ウイルス) / Simian-Human immunodeficiency virus (ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Edwards RJ / Manne K / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: Fab-dimerized glycan-reactive antibodies are a structural category of natural antibodies. 著者: Wilton B Williams / R Ryan Meyerhoff / R J Edwards / Hui Li / Kartik Manne / Nathan I Nicely / Rory Henderson / Ye Zhou / Katarzyna Janowska / Katayoun Mansouri / Sophie Gobeil / Tyler ...著者: Wilton B Williams / R Ryan Meyerhoff / R J Edwards / Hui Li / Kartik Manne / Nathan I Nicely / Rory Henderson / Ye Zhou / Katarzyna Janowska / Katayoun Mansouri / Sophie Gobeil / Tyler Evangelous / Bhavna Hora / Madison Berry / A Yousef Abuahmad / Jordan Sprenz / Margaret Deyton / Victoria Stalls / Megan Kopp / Allen L Hsu / Mario J Borgnia / Guillaume B E Stewart-Jones / Matthew S Lee / Naomi Bronkema / M Anthony Moody / Kevin Wiehe / Todd Bradley / S Munir Alam / Robert J Parks / Andrew Foulger / Thomas Oguin / Gregory D Sempowski / Mattia Bonsignori / Celia C LaBranche / David C Montefiori / Michael Seaman / Sampa Santra / John Perfect / Joseph R Francica / Geoffrey M Lynn / Baptiste Aussedat / William E Walkowicz / Richard Laga / Garnett Kelsoe / Kevin O Saunders / Daniela Fera / Peter D Kwong / Robert A Seder / Alberto Bartesaghi / George M Shaw / Priyamvada Acharya / Barton F Haynes /   要旨: Natural antibodies (Abs) can target host glycans on the surface of pathogens. We studied the evolution of glycan-reactive B cells of rhesus macaques and humans using glycosylated HIV-1 envelope (Env) ...Natural antibodies (Abs) can target host glycans on the surface of pathogens. We studied the evolution of glycan-reactive B cells of rhesus macaques and humans using glycosylated HIV-1 envelope (Env) as a model antigen. 2G12 is a broadly neutralizing Ab (bnAb) that targets a conserved glycan patch on Env of geographically diverse HIV-1 strains using a unique heavy-chain (V) domain-swapped architecture that results in fragment antigen-binding (Fab) dimerization. Here, we describe HIV-1 Env Fab-dimerized glycan (FDG)-reactive bnAbs without V-swapped domains from simian-human immunodeficiency virus (SHIV)-infected macaques. FDG Abs also recognized cell-surface glycans on diverse pathogens, including yeast and severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) spike. FDG precursors were expanded by glycan-bearing immunogens in macaques and were abundant in HIV-1-naive humans. Moreover, FDG precursors were predominately mutated IgMIgDCD27, thus suggesting that they originated from a pool of antigen-experienced IgM or marginal zone B cells. #1:  ジャーナル: bioRxiv / 年: 2020 ジャーナル: bioRxiv / 年: 2020タイトル: Fab-dimerized glycan-reactive antibodies neutralize HIV and are prevalent in humans and rhesus macaques 著者: Acharya P / Williams W / Henderson R / Janowska K / Manne K / Parks R / Deyton M / Sprenz J / Stalls V / Kopp M / Mansouri K / Edwards RJ / Meyerhoff RR / Oguin T / Sempowski G / Saunders K / Haynes BF | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23519.map.gz emd_23519.map.gz | 6.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23519-v30.xml emd-23519-v30.xml emd-23519.xml emd-23519.xml | 24.7 KB 24.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23519.png emd_23519.png | 129.5 KB | ||
| Filedesc metadata |  emd-23519.cif.gz emd-23519.cif.gz | 7.6 KB | ||
| その他 |  emd_23519_half_map_1.map.gz emd_23519_half_map_1.map.gz emd_23519_half_map_2.map.gz emd_23519_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23519 http://ftp.pdbj.org/pub/emdb/structures/EMD-23519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23519 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23519_validation.pdf.gz emd_23519_validation.pdf.gz | 815.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23519_full_validation.pdf.gz emd_23519_full_validation.pdf.gz | 814.9 KB | 表示 | |
| XML形式データ |  emd_23519_validation.xml.gz emd_23519_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_23519_validation.cif.gz emd_23519_validation.cif.gz | 14.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23519 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23519 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23519 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23519 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23519.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23519.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0665 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Cryo-EM map of DH898.1 Fab-dimer bound near the...
| ファイル | emd_23519_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
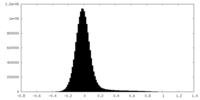
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM half-map B of DH898.1 Fab-dimer bound near...
| ファイル | emd_23519_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM half-map B of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
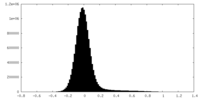
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of DH898.1 Fab-dimer bound near the CD4 binding...
| 全体 | 名称: Cryo-EM structure of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer. |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of DH898.1 Fab-dimer bound near the CD4 binding...
| 超分子 | 名称: Cryo-EM structure of DH898.1 Fab-dimer bound near the CD4 binding site of HIV-1 Env CH848 SOSIP trimer. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: CH848 SOSIP gp120
| 分子 | 名称: CH848 SOSIP gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 52.06193 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKEAKT TLFCASDARA YEKEVHNVWA THACVPTDPS PQELVLGNVT ENFNMWKNDM VDQMHEDIIS LWDQSLKPC VKLTPLCVTL ICSNATVKNG TVEEMKNCSF NTTTEIRDKE KKEYALFYKP DIVPLSETNN TSEYRLINCN T SACTQACP ...文字列: AENLWVTVYY GVPVWKEAKT TLFCASDARA YEKEVHNVWA THACVPTDPS PQELVLGNVT ENFNMWKNDM VDQMHEDIIS LWDQSLKPC VKLTPLCVTL ICSNATVKNG TVEEMKNCSF NTTTEIRDKE KKEYALFYKP DIVPLSETNN TSEYRLINCN T SACTQACP KVTFEPIPIH YCAPAGYAIL KCNDETFNGT GPCSNVSTVQ CTHGIRPVVS TQLLLNGSLA EKEIVIRSEN LT NNAKIII VHLHTPVEIV CTRPNNNTRK SVRIGPGQTF YATGDIIGDI KQAHCNISEE KWNDTLQKVG IELQKHFPNK TIK YNQSAG GDMEITTHSF NCGGEFFYCN TSNLFNGTYN GTYISTNSSA NSTSTITLQC RIKQIINMWQ GVGRCMYAPP IAGN ITCRS NITGLLLTRD GGTNSNETET FRPAGGDMRD NWRSELYKYK VVKIEPLGVA PTRCKRRVVG R UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Env polyprotein
| 分子 | 名称: Env polyprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Simian-Human immunodeficiency virus (ウイルス) Simian-Human immunodeficiency virus (ウイルス) |
| 分子量 | 理論値: 16.578803 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: FLGFLGAAGS TMGAASMTLT VQARNLLSGI VQQQSNLLRA PEAQQHLLKL TVWGIKQLQA RVLAVERYLR DQQLLGIWGC SGKLICCTN VPWNSSWSNR NLSEIWDNMT WLQWDKEISN YTQIIYGLLE ESQNQQEKNE QDLLALD UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: DH898.1 light chain
| 分子 | 名称: DH898.1 light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.55325 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVMTQTPLS LSVTPGEPAS LSCRSSASLL HGNGNTYLHW YLRKAGQSPQ LLIFGGSKRV PGISDRFIGS GAGTNFTLKI SSVEADDVG FYYCAQGVAF PWTFGQGTKV EIKRAVAAPS VFIFPPSEDQ VKSGTVSVVC LLNNFYPREA SVKWKVDGVL K TGNSQESV ...文字列: EIVMTQTPLS LSVTPGEPAS LSCRSSASLL HGNGNTYLHW YLRKAGQSPQ LLIFGGSKRV PGISDRFIGS GAGTNFTLKI SSVEADDVG FYYCAQGVAF PWTFGQGTKV EIKRAVAAPS VFIFPPSEDQ VKSGTVSVVC LLNNFYPREA SVKWKVDGVL K TGNSQESV TEQDSKDNTY SLSSTLTLSN TDYQSHNVYA CEVTHQGLSS PVTKSFNRGE |
-分子 #4: DH898.1 heavy chain
| 分子 | 名称: DH898.1 heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.596594 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QDLLLQSGAE VREPGASVTV SCQASNYTFP DYYIHWVRLV PGQGLEWLGE MKPKVGVTNV SKKIRDRLFM TADTSTDTAY MVLSALTPG DTAIYYCTRL EPDFLSGWAH WGKGVLVTVS PASTKGPSVF PLAPSSRSTS ESTAALGCLV KDYFPEPVTV S WNSGSLTS ...文字列: QDLLLQSGAE VREPGASVTV SCQASNYTFP DYYIHWVRLV PGQGLEWLGE MKPKVGVTNV SKKIRDRLFM TADTSTDTAY MVLSALTPG DTAIYYCTRL EPDFLSGWAH WGKGVLVTVS PASTKGPSVF PLAPSSRSTS ESTAALGCLV KDYFPEPVTV S WNSGSLTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYVCNVNHK PSNTKVDKRV E |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 17 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 293 K / 装置: LEICA EM GP / 詳細: blot for 2.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 93.15 K / 最高: 93.15 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3230 / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7lua: |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)