登録情報 データベース : EMDB / ID : EMD-23290タイトル native AMPA receptor 複合体 : GluA1/A2/A3-asymmetric-conformation1 with bound antibody fragments機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mus musculus (ハツカネズミ)手法 / / 解像度 : 7.5 Å Yu J / Rao P 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of Neurological Disorders and Stroke (NIH/NINDS) R01NS038631
ジャーナル : Science / 年 : 2019タイトル : Architecture and subunit arrangement of native AMPA receptors elucidated by cryo-EM.著者 : Yan Zhao / Shanshuang Chen / Adam C Swensen / Wei-Jun Qian / Eric Gouaux / 要旨 : Glutamate-gated AMPA receptors mediate the fast component of excitatory signal transduction at chemical synapses throughout all regions of the mammalian brain. AMPA receptors are tetrameric ... Glutamate-gated AMPA receptors mediate the fast component of excitatory signal transduction at chemical synapses throughout all regions of the mammalian brain. AMPA receptors are tetrameric assemblies composed of four subunits, GluA1-GluA4. Despite decades of study, the subunit composition, subunit arrangement, and molecular structure of native AMPA receptors remain unknown. Here we elucidate the structures of 10 distinct native AMPA receptor complexes by single-particle cryo-electron microscopy (cryo-EM). We find that receptor subunits are arranged nonstochastically, with the GluA2 subunit preferentially occupying the B and D positions of the tetramer and with triheteromeric assemblies comprising a major population of native AMPA receptors. Cryo-EM maps define the structure for S2-M4 linkers between the ligand-binding and transmembrane domains, suggesting how neurotransmitter binding is coupled to ion channel gating. 履歴 登録 2021年1月14日 - ヘッダ(付随情報) 公開 2022年1月19日 - マップ公開 2022年1月19日 - 更新 2022年8月10日 - 現状 2022年8月10日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
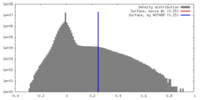
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Science / 年: 2019
ジャーナル: Science / 年: 2019
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_23290.map.gz
emd_23290.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-23290-v30.xml
emd-23290-v30.xml emd-23290.xml
emd-23290.xml EMDBヘッダ
EMDBヘッダ emd_23290.png
emd_23290.png http://ftp.pdbj.org/pub/emdb/structures/EMD-23290
http://ftp.pdbj.org/pub/emdb/structures/EMD-23290 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23290
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23290 emd_23290_validation.pdf.gz
emd_23290_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_23290_full_validation.pdf.gz
emd_23290_full_validation.pdf.gz emd_23290_validation.xml.gz
emd_23290_validation.xml.gz emd_23290_validation.cif.gz
emd_23290_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23290
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23290 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23290
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23290 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_23290.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_23290.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)