+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13460 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the 70S ribosome with tRNAs in hybrid state 2 (H2) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | EF-G / robosome / 70S / apramycin / translocation / RIBOSOME | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to reactive oxygen species / response to radiation / mRNA 5'-UTR binding / ribosome biogenesis / large ribosomal subunit / transferase activity / small ribosomal subunit / small ribosomal subunit rRNA binding / ribosomal large subunit assembly / cytosolic small ribosomal subunit ...response to reactive oxygen species / response to radiation / mRNA 5'-UTR binding / ribosome biogenesis / large ribosomal subunit / transferase activity / small ribosomal subunit / small ribosomal subunit rRNA binding / ribosomal large subunit assembly / cytosolic small ribosomal subunit / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / tRNA binding / negative regulation of translation / rRNA binding / structural constituent of ribosome / ribosome / translation / ribonucleoprotein complex / response to antibiotic / mRNA binding / metal ion binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
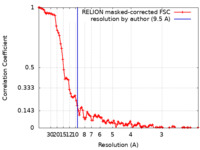
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.5 Å | |||||||||
 データ登録者 データ登録者 | Petrychenko V / Peng BZ | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural mechanism of GTPase-powered ribosome-tRNA movement. 著者: Valentyn Petrychenko / Bee-Zen Peng / Ana C de A P Schwarzer / Frank Peske / Marina V Rodnina / Niels Fischer /  要旨: GTPases are regulators of cell signaling acting as molecular switches. The translational GTPase EF-G stands out, as it uses GTP hydrolysis to generate force and promote the movement of the ribosome ...GTPases are regulators of cell signaling acting as molecular switches. The translational GTPase EF-G stands out, as it uses GTP hydrolysis to generate force and promote the movement of the ribosome along the mRNA. The key unresolved question is how GTP hydrolysis drives molecular movement. Here, we visualize the GTPase-powered step of ongoing translocation by time-resolved cryo-EM. EF-G in the active GDP-Pi form stabilizes the rotated conformation of ribosomal subunits and induces twisting of the sarcin-ricin loop of the 23 S rRNA. Refolding of the GTPase switch regions upon Pi release initiates a large-scale rigid-body rotation of EF-G pivoting around the sarcin-ricin loop that facilitates back rotation of the ribosomal subunits and forward swiveling of the head domain of the small subunit, ultimately driving tRNA forward movement. The findings demonstrate how a GTPase orchestrates spontaneous thermal fluctuations of a large RNA-protein complex into force-generating molecular movement. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13460.map.gz emd_13460.map.gz | 83.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13460-v30.xml emd-13460-v30.xml emd-13460.xml emd-13460.xml | 80.4 KB 80.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13460_fsc.xml emd_13460_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13460.png emd_13460.png | 148.9 KB | ||
| マスクデータ |  emd_13460_msk_1.map emd_13460_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13460.cif.gz emd-13460.cif.gz | 16 KB | ||
| その他 |  emd_13460_half_map_1.map.gz emd_13460_half_map_1.map.gz emd_13460_half_map_2.map.gz emd_13460_half_map_2.map.gz | 71.1 MB 71.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13460 http://ftp.pdbj.org/pub/emdb/structures/EMD-13460 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13460 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13460 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13460_validation.pdf.gz emd_13460_validation.pdf.gz | 164.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13460_full_validation.pdf.gz emd_13460_full_validation.pdf.gz | 164.5 KB | 表示 | |
| XML形式データ |  emd_13460_validation.xml.gz emd_13460_validation.xml.gz | 572 B | 表示 | |
| CIF形式データ |  emd_13460_validation.cif.gz emd_13460_validation.cif.gz | 484 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13460 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13460 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13460 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13460 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7pjuMC  7pjsC  7pjtC  7pjvC  7pjwC  7pjxC  7pjyC  7pjzC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10792 (タイトル: Structural mechanism of GTPase-powered ribosome-tRNA movement EMPIAR-10792 (タイトル: Structural mechanism of GTPase-powered ribosome-tRNA movementData size: 987.5 Data #1: Motion-corrected, dose-weighted micrographs [micrographs - single frame] Data #2: Shiny particles of non-rotated ribosome particles (C state), obtained by Bayesian polishing in Relion 3.1 [picked particles - single frame - processed] Data #3: Shiny particles of rotated ribosome particles, obtained by Bayesian polishing in Relion 3.1 [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13460.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13460.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13460_msk_1.map emd_13460_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13460_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13460_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : E. coli ribosome complex 70S-Apr-EF-G-GDP-Pi-fMet-Phe-tRNAPhe-tRN...
+超分子 #1: E. coli ribosome complex 70S-Apr-EF-G-GDP-Pi-fMet-Phe-tRNAPhe-tRN...
+分子 #1: 50S ribosomal protein L32
+分子 #2: 50S ribosomal protein L33
+分子 #3: 50S ribosomal protein L34
+分子 #4: 50S ribosomal protein L35
+分子 #5: 50S ribosomal protein L36
+分子 #6: 50S ribosomal protein L10
+分子 #7: 50S ribosomal protein L31
+分子 #10: 50S ribosomal protein L2
+分子 #11: 50S ribosomal protein L3
+分子 #12: 50S ribosomal protein L4
+分子 #13: 50S ribosomal protein L5
+分子 #14: 50S ribosomal protein L6
+分子 #15: 50S ribosomal protein L9
+分子 #16: Ribosomal protein L11
+分子 #17: 50S ribosomal protein L13
+分子 #18: 50S ribosomal protein L14
+分子 #19: 50S ribosomal protein L15
+分子 #20: 50S ribosomal protein L16
+分子 #21: 50S ribosomal protein L17
+分子 #22: 50S ribosomal protein L18
+分子 #23: 50S ribosomal protein L19
+分子 #24: 50S ribosomal protein L20
+分子 #25: 50S ribosomal protein L21
+分子 #26: Ribosomal protein L22
+分子 #27: 50S ribosomal protein L23
+分子 #28: Ribosomal protein L24
+分子 #29: 50S ribosomal protein L25
+分子 #30: 50S ribosomal protein L27
+分子 #31: 50S ribosomal protein L28
+分子 #32: 50S ribosomal protein L29
+分子 #33: 50S ribosomal protein L30
+分子 #35: 30S ribosomal protein S2
+分子 #36: 30S ribosomal protein S3
+分子 #37: Ribosomal protein S4
+分子 #38: 30S ribosomal protein S5
+分子 #39: 30S ribosomal protein S6, fully modified isoform
+分子 #40: 30S ribosomal protein S7
+分子 #41: 30S ribosomal protein S8
+分子 #42: 30S ribosomal protein S9
+分子 #43: 30S ribosomal protein S10
+分子 #44: 30S ribosomal protein S11
+分子 #45: 30S ribosomal protein S12
+分子 #46: 30S ribosomal protein S13
+分子 #47: 30S ribosomal protein S14
+分子 #48: 30S ribosomal protein S15
+分子 #49: 30S ribosomal protein S16
+分子 #50: 30S ribosomal protein S17
+分子 #51: 30S ribosomal protein S18
+分子 #52: 30S ribosomal protein S19
+分子 #53: 30S ribosomal protein S20
+分子 #54: 30S ribosomal protein S21
+分子 #57: Dipeptide (FME-PHE)
+分子 #8: 23S ribosomal RNA
+分子 #9: 5S ribosomal RNA
+分子 #34: 16S ribosomal RNA
+分子 #55: P-site tRNA(fMet)
+分子 #56: P-site fMet-Phe-tRNA(Phe)
+分子 #58: mRNA
+分子 #59: ZINC ION
+分子 #60: APRAMYCIN
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: 50 mM HEPES, 70 mM NH4Cl, 30 mM KCl, 3.5 mM MgCl2, 0.6 mM spermine, 0.4 mM spermidine |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER / 詳細: Manual blotting & plunge-freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Aberration corrections performed using Cs image corrector (CEOS company) |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 8221 / 平均露光時間: 1.0 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 1.2 µm / 最小 デフォーカス(補正後): 0.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | Instead of Chimera ChimeraX was used. | ||||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: RSCC | ||||||||
| 得られたモデル |  PDB-7pju: |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)