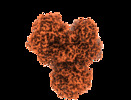
登録情報 データベース : EMDB / ID : EMD-12661タイトル Respiratory complex I from Escherichia coli - focused refinement of cytoplasmic arm 複合体 : Respiratory complex I from Escherichia coli - focused refinement of cytoplasmic armリガンド : x 5種機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Escherichia coli B (大腸菌)手法 / / 解像度 : 2.1 Å Kolata P / Efremov RG 資金援助 Organization Grant number 国 European Research Council (ERC) 00281ROEF
ジャーナル : Elife / 年 : 2021タイトル : Structure of respiratory complex I reconstituted into lipid nanodiscs reveals an uncoupled conformation.著者 : Piotr Kolata / Rouslan G Efremov / 要旨 : Respiratory complex I is a multi-subunit membrane protein complex that reversibly couples NADH oxidation and ubiquinone reduction with proton translocation against transmembrane potential. Complex I ... Respiratory complex I is a multi-subunit membrane protein complex that reversibly couples NADH oxidation and ubiquinone reduction with proton translocation against transmembrane potential. Complex I from is among the best functionally characterized complexes, but its structure remains unknown, hindering further studies to understand the enzyme coupling mechanism. Here, we describe the single particle cryo-electron microscopy (cryo-EM) structure of the entire catalytically active complex I reconstituted into lipid nanodiscs. The structure of this mesophilic bacterial complex I displays highly dynamic connection between the peripheral and membrane domains. The peripheral domain assembly is stabilized by unique terminal extensions and an insertion loop. The membrane domain structure reveals novel dynamic features. Unusual conformation of the conserved interface between the peripheral and membrane domains suggests an uncoupled conformation of the complex. Considering constraints imposed by the structural data, we suggest a new simple hypothetical coupling mechanism for the molecular machine. 履歴 登録 2021年3月23日 - ヘッダ(付随情報) 公開 2021年8月18日 - マップ公開 2021年8月18日 - 更新 2021年8月18日 - 現状 2021年8月18日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ベルギー, 1件
ベルギー, 1件  引用
引用 ジャーナル: Elife / 年: 2021
ジャーナル: Elife / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_12661.map.gz
emd_12661.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-12661-v30.xml
emd-12661-v30.xml emd-12661.xml
emd-12661.xml EMDBヘッダ
EMDBヘッダ emd_12661_fsc.xml
emd_12661_fsc.xml FSCデータファイル
FSCデータファイル emd_12661.png
emd_12661.png emd_12661_half_map_1.map.gz
emd_12661_half_map_1.map.gz emd_12661_half_map_2.map.gz
emd_12661_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-12661
http://ftp.pdbj.org/pub/emdb/structures/EMD-12661 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12661
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12661 emd_12661_validation.pdf.gz
emd_12661_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_12661_full_validation.pdf.gz
emd_12661_full_validation.pdf.gz emd_12661_validation.xml.gz
emd_12661_validation.xml.gz emd_12661_validation.cif.gz
emd_12661_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12661
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12661 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12661
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12661 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_12661.map.gz / 形式: CCP4 / 大きさ: 21 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_12661.map.gz / 形式: CCP4 / 大きさ: 21 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN ムービー
ムービー コントローラー
コントローラー

































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)