+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1105 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Global conformational rearrangements during the activation of the GDP/GTP exchange factor Vav3. | |||||||||
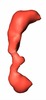
 マップデータ マップデータ | 3D volume of a truncated form of Vav3, Vav3(d1-144) missing the first 144 aminoacids and which displays oncogenic potential | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | small GTPase-mediated signal transduction 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 22.0 Å | |||||||||
 データ登録者 データ登録者 | Llorca O / Arias-Palomo E / Zugaza JL / Bustelo XR | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2005 ジャーナル: EMBO J / 年: 2005タイトル: Global conformational rearrangements during the activation of the GDP/GTP exchange factor Vav3. 著者: Oscar Llorca / Ernesto Arias-Palomo / José L Zugaza / Xosé R Bustelo /  要旨: Activation of Rho/Rac GTPases during cell signaling requires the participation of GDP/GTP exchange factors of the Dbl family. Although the structure of the catalytic core of Dbl proteins has been ...Activation of Rho/Rac GTPases during cell signaling requires the participation of GDP/GTP exchange factors of the Dbl family. Although the structure of the catalytic core of Dbl proteins has been established recently, the molecular changes that the full-length proteins experience during normal or oncogenic conditions of stimulation are still unknown. Here, we have used single-particle electron microscopy to solve the structures of the inactive (unphosphorylated), active (phosphorylated), and constitutively active (N-terminally deleted) versions of the exchange factor Vav3. Comparison of these forms has revealed the interdomain interactions maintaining the inactive Vav3 state and the dynamic changes that the overall Vav3 structure undergoes upon tyrosine phosphorylation. We have also found that the conformations of phosphorylated Vav3 and N-terminally deleted Vav3 are distinct, indicating that the acquisition of constitutive activity by exchange factors is structurally more complex than the mere elimination of inhibitory interactions between structural domains. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1105.map.gz emd_1105.map.gz | 39.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1105-v30.xml emd-1105-v30.xml emd-1105.xml emd-1105.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1105.gif 1105.gif emd_1105.tif emd_1105.tif | 3.1 KB 182 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1105 http://ftp.pdbj.org/pub/emdb/structures/EMD-1105 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1105 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1105 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1105_validation.pdf.gz emd_1105_validation.pdf.gz | 184.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1105_full_validation.pdf.gz emd_1105_full_validation.pdf.gz | 183.8 KB | 表示 | |
| XML形式データ |  emd_1105_validation.xml.gz emd_1105_validation.xml.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1105 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1105 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1105 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1105 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1105.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1105.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D volume of a truncated form of Vav3, Vav3(d1-144) missing the first 144 aminoacids and which displays oncogenic potential | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : N-terminally truncated form of Vav3 missing the first 144 aminoacids
| 全体 | 名称: N-terminally truncated form of Vav3 missing the first 144 aminoacids |
|---|---|
| 要素 |
|
-超分子 #1000: N-terminally truncated form of Vav3 missing the first 144 aminoacids
| 超分子 | 名称: N-terminally truncated form of Vav3 missing the first 144 aminoacids タイプ: sample / ID: 1000 / 集合状態: monomer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 81 KDa / 手法: from its sequence |
-分子 #1: Vav3 with 1-144 deletion
| 分子 | 名称: Vav3 with 1-144 deletion / タイプ: protein_or_peptide / ID: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞: baculovirus-infected S. frugiperda cells / 細胞中の位置: cytoplam Homo sapiens (ヒト) / 別称: Human / 細胞: baculovirus-infected S. frugiperda cells / 細胞中の位置: cytoplam |
| 分子量 | 実験値: 81 KDa |
| 組換発現 | 生物種: baculovirus-infected S. frugiperda cells |
| 配列 | GO: small GTPase-mediated signal transduction |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50 mM Tris-Hcl, 100 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: Glow-discharged carbon-coated grids and negatively stained with 1% W/v uranyl acetate |
| グリッド | 詳細: 400 mesh copper-rhodium grids |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1230 |
|---|---|
| 温度 | 平均: 293 K |
| アライメント法 | Legacy - 非点収差: correction with CCD camera |
| 詳細 | Electron Microscope:JEOL JEM-1230. Specimen holder:JEOL type M, 207EM-11020. |
| 日付 | 2003年3月2日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK 4489 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 10 µm / 詳細: Digitiser used: Minolta Dimage Scan Multi Pro / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.9 mm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 22.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 使用した粒子像数: 4604 |
|---|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)


























