+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10663 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Focused refinement CF in eiPIC | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.54 Å | |||||||||
 データ登録者 データ登録者 | Pilsl M / Engel C | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural basis of RNA polymerase I pre-initiation complex formation and promoter melting. 著者: Michael Pilsl / Christoph Engel /  要旨: Transcription of the ribosomal RNA precursor by RNA polymerase (Pol) I is a prerequisite for the biosynthesis of ribosomes in eukaryotes. Compared to Pols II and III, the mechanisms underlying ...Transcription of the ribosomal RNA precursor by RNA polymerase (Pol) I is a prerequisite for the biosynthesis of ribosomes in eukaryotes. Compared to Pols II and III, the mechanisms underlying promoter recognition, initiation complex formation and DNA melting by Pol I substantially diverge. Here, we report the high-resolution cryo-EM reconstruction of a Pol I early initiation intermediate assembled on a double-stranded promoter scaffold that prevents the establishment of downstream DNA contacts. Our analyses demonstrate how efficient promoter-backbone interaction is achieved by combined re-arrangements of flexible regions in the 'core factor' subunits Rrn7 and Rrn11. Furthermore, structure-function analysis illustrates how destabilization of the melted DNA region correlates with contraction of the polymerase cleft upon transcription activation, thereby combining promoter recruitment with DNA-melting. This suggests that molecular mechanisms and structural features of Pol I initiation have co-evolved to support the efficient melting, initial transcription and promoter clearance required for high-level rRNA synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10663.map.gz emd_10663.map.gz | 258.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10663-v30.xml emd-10663-v30.xml emd-10663.xml emd-10663.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10663.png emd_10663.png | 182.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10663 http://ftp.pdbj.org/pub/emdb/structures/EMD-10663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10663 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10663_validation.pdf.gz emd_10663_validation.pdf.gz | 253 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10663_full_validation.pdf.gz emd_10663_full_validation.pdf.gz | 252.1 KB | 表示 | |
| XML形式データ |  emd_10663_validation.xml.gz emd_10663_validation.xml.gz | 6.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10663 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10663 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10663 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10663 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10663.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10663.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
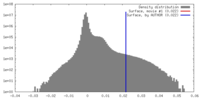
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : early intermediate RNA-Polymerase I Pre-initiation Complex - eiPIC
| 全体 | 名称: early intermediate RNA-Polymerase I Pre-initiation Complex - eiPIC |
|---|---|
| 要素 |
|
-超分子 #1: early intermediate RNA-Polymerase I Pre-initiation Complex - eiPIC
| 超分子 | 名称: early intermediate RNA-Polymerase I Pre-initiation Complex - eiPIC タイプ: complex / ID: 1 / 親要素: 0 |
|---|
-超分子 #2: RNA polymerase
| 超分子 | 名称: RNA polymerase / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: transcription initiation factor
| 超分子 | 名称: transcription initiation factor / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-超分子 #4: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 4 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種: Synthetic construct (人工物) |
| 組換発現 | 生物種: Synthetic construct (人工物) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 / 構成要素 - 式: KCl |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 4088 / 平均電子線量: 1.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)