+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10495 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of NADH reduced form of NAD+-dependent Formate Dehydrogenase from Rhodobacter capsulatus | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | molybdoenzyme / formate oxidation / NAD+-dependent / OXIDOREDUCTASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報formate dehydrogenase complex / formate metabolic process / formate dehydrogenase / formate dehydrogenase / formate dehydrogenase (NAD+) activity / oxidoreductase complex / molybdopterin cofactor binding / NADH dehydrogenase activity / NADH dehydrogenase (ubiquinone) activity / respiratory electron transport chain ...formate dehydrogenase complex / formate metabolic process / formate dehydrogenase / formate dehydrogenase / formate dehydrogenase (NAD+) activity / oxidoreductase complex / molybdopterin cofactor binding / NADH dehydrogenase activity / NADH dehydrogenase (ubiquinone) activity / respiratory electron transport chain / 2 iron, 2 sulfur cluster binding / FMN binding / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / metal ion binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Rhodobacter capsulatus (バクテリア) Rhodobacter capsulatus (バクテリア) | ||||||||||||
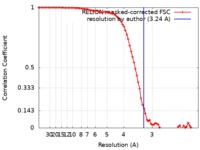
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.24 Å | ||||||||||||
 データ登録者 データ登録者 | Wendler P / Radon C | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Cryo-EM structures reveal intricate Fe-S cluster arrangement and charging in Rhodobacter capsulatus formate dehydrogenase. 著者: Christin Radon / Gerd Mittelstädt / Benjamin R Duffus / Jörg Bürger / Tobias Hartmann / Thorsten Mielke / Christian Teutloff / Silke Leimkühler / Petra Wendler /   要旨: Metal-containing formate dehydrogenases (FDH) catalyse the reversible oxidation of formate to carbon dioxide at their molybdenum or tungsten active site. They display a diverse subunit and cofactor ...Metal-containing formate dehydrogenases (FDH) catalyse the reversible oxidation of formate to carbon dioxide at their molybdenum or tungsten active site. They display a diverse subunit and cofactor composition, but structural information on these enzymes is limited. Here we report the cryo-electron microscopic structures of the soluble Rhodobacter capsulatus FDH (RcFDH) as isolated and in the presence of reduced nicotinamide adenine dinucleotide (NADH). RcFDH assembles into a 360 kDa dimer of heterotetramers revealing a putative interconnection of electron pathway chains. In the presence of NADH, the RcFDH structure shows charging of cofactors, indicative of an increased electron load. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10495.map.gz emd_10495.map.gz | 4.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10495-v30.xml emd-10495-v30.xml emd-10495.xml emd-10495.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10495_fsc.xml emd_10495_fsc.xml | 8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10495.png emd_10495.png | 93 KB | ||
| Filedesc metadata |  emd-10495.cif.gz emd-10495.cif.gz | 7.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10495 http://ftp.pdbj.org/pub/emdb/structures/EMD-10495 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10495 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10495 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10495_validation.pdf.gz emd_10495_validation.pdf.gz | 431.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10495_full_validation.pdf.gz emd_10495_full_validation.pdf.gz | 431.1 KB | 表示 | |
| XML形式データ |  emd_10495_validation.xml.gz emd_10495_validation.xml.gz | 10.1 KB | 表示 | |
| CIF形式データ |  emd_10495_validation.cif.gz emd_10495_validation.cif.gz | 13.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10495 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10495 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10495 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10495 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10495.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10495.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Formate dehydrogenase, NADH reduced
+超分子 #1: Formate dehydrogenase, NADH reduced
+分子 #1: Formate dehydrogenase subunit alpha
+分子 #2: Formate dehydrogenase subunit beta
+分子 #3: Formate dehydrogenase subunit gamma
+分子 #4: NAD-dependent formate dehydrogenase subunit delta
+分子 #5: 2-AMINO-5,6-DIMERCAPTO-7-METHYL-3,7,8A,9-TETRAHYDRO-8-OXA-1,3,9,1...
+分子 #6: MOLYBDENUM(VI) ION
+分子 #7: FE2/S2 (INORGANIC) CLUSTER
+分子 #8: IRON/SULFUR CLUSTER
+分子 #9: HYDROSULFURIC ACID
+分子 #10: FLAVIN MONONUCLEOTIDE
+分子 #11: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R2/4 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI POLARA 300 |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 64.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE | ||||||||
| 得られたモデル |  PDB-6tg9: |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)