+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0690 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The reconstruction of apo-state streptavidin at 3.3 Angstrom resolution | |||||||||
 マップデータ マップデータ | apo-state streptavidin | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | streptavidin / CYTOSOLIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Streptomyces avidinii (バクテリア) Streptomyces avidinii (バクテリア) | |||||||||
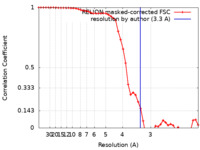
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Fan X / Wang J / Lei JL / Wang HW | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Single particle cryo-EM reconstruction of 52 kDa streptavidin at 3.2 Angstrom resolution. 著者: Xiao Fan / Jia Wang / Xing Zhang / Zi Yang / Jin-Can Zhang / Lingyun Zhao / Hai-Lin Peng / Jianlin Lei / Hong-Wei Wang /  要旨: The fast development of single-particle cryogenic electron microscopy (cryo-EM) has made it more feasible to obtain the 3D structure of well-behaved macromolecules with a molecular weight higher than ...The fast development of single-particle cryogenic electron microscopy (cryo-EM) has made it more feasible to obtain the 3D structure of well-behaved macromolecules with a molecular weight higher than 300 kDa at ~3 Å resolution. However, it remains a challenge to obtain the high-resolution structures of molecules smaller than 200 kDa using single-particle cryo-EM. In this work, we apply the Cs-corrector-VPP-coupled cryo-EM to study the 52 kDa streptavidin (SA) protein supported on a thin layer of graphene and embedded in vitreous ice. We are able to solve both the apo-SA and biotin-bound SA structures at near-atomic resolution using single-particle cryo-EM. We demonstrate that the method has the potential to determine the structures of molecules as small as 39 kDa. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0690.map.gz emd_0690.map.gz | 916.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0690-v30.xml emd-0690-v30.xml emd-0690.xml emd-0690.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0690_fsc.xml emd_0690_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0690.png emd_0690.png | 68.5 KB | ||
| Filedesc metadata |  emd-0690.cif.gz emd-0690.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0690 http://ftp.pdbj.org/pub/emdb/structures/EMD-0690 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0690 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0690 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0690_validation.pdf.gz emd_0690_validation.pdf.gz | 392.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0690_full_validation.pdf.gz emd_0690_full_validation.pdf.gz | 392.1 KB | 表示 | |
| XML形式データ |  emd_0690_validation.xml.gz emd_0690_validation.xml.gz | 7.9 KB | 表示 | |
| CIF形式データ |  emd_0690_validation.cif.gz emd_0690_validation.cif.gz | 10 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0690 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0690 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0690 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0690 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6j6kMC  0689C  6j6jC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10269 (タイトル: Single particle reconstruction of 52 kDa apo-state streptavidin at 3.3 Angstrom resolution EMPIAR-10269 (タイトル: Single particle reconstruction of 52 kDa apo-state streptavidin at 3.3 Angstrom resolutionData size: 2.4 TB Data #1: Uncorrected apo-state streptavidin movie stacks, binning 2 from super-resolution stacks. [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0690.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0690.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | apo-state streptavidin | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.053 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
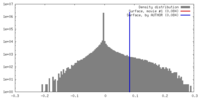
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Streptavidin
| 全体 | 名称: Streptavidin |
|---|---|
| 要素 |
|
-超分子 #1: Streptavidin
| 超分子 | 名称: Streptavidin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptomyces avidinii (バクテリア) Streptomyces avidinii (バクテリア) |
| 分子量 | 理論値: 52 KDa |
-分子 #1: Streptavidin
| 分子 | 名称: Streptavidin / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptomyces avidinii (バクテリア) Streptomyces avidinii (バクテリア) |
| 分子量 | 理論値: 12.596641 KDa |
| 配列 | 文字列: GITGTWYNQL GSTFIVTAGA DGALTGTYES AVGNAESRYV LTGRYDSAPA TDGSGTALGW TVAWKNNYRN AHSATTWSGQ YVGGAEARI NTQWLLTSGT TEANAWKSTL VGHDTFTKVK UniProtKB: Streptavidin |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 285 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / 球面収差補正装置: spherical aberration corrector |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-32 / 撮影したグリッド数: 1 / 実像数: 1450 / 平均露光時間: 2.56 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): -0.8 µm / 最小 デフォーカス(公称値): -0.8 µm / 倍率(公称値): 215000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)