+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | Proline utilization A from Desulfovibrio vulgaris 4.5 mg/mL
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proline dehydrogenase / proline dehydrogenase activity / oxidoreductase activity, acting on the aldehyde or oxo group of donors, NAD or NADP as acceptor / L-glutamate gamma-semialdehyde dehydrogenase / L-glutamate gamma-semialdehyde dehydrogenase activity / L-proline catabolic process to L-glutamate / : / DNA-binding transcription factor activity / DNA binding 類似検索 - 分子機能 |
| 生物種 |  Desulfovibrio vulgaris (strain RCH1) (バクテリア) Desulfovibrio vulgaris (strain RCH1) (バクテリア) |
 引用 引用 |  ジャーナル: FEBS J / 年: 2017 ジャーナル: FEBS J / 年: 2017タイトル: Biophysical investigation of type A PutAs reveals a conserved core oligomeric structure. 著者: David A Korasick / Harkewal Singh / Travis A Pemberton / Min Luo / Richa Dhatwalia / John J Tanner /  要旨: Many enzymes form homooligomers, yet the functional significance of self-association is seldom obvious. Herein, we examine the connection between oligomerization and catalytic function for proline ...Many enzymes form homooligomers, yet the functional significance of self-association is seldom obvious. Herein, we examine the connection between oligomerization and catalytic function for proline utilization A (PutA) enzymes. PutAs are bifunctional enzymes that catalyze both reactions of proline catabolism. Type A PutAs are the smallest members of the family, possessing a minimal domain architecture consisting of N-terminal proline dehydrogenase and C-terminal l-glutamate-γ-semialdehyde dehydrogenase modules. Type A PutAs form domain-swapped dimers, and in one case (Bradyrhizobium japonicum PutA), two of the dimers assemble into a ring-shaped tetramer. Whereas the dimer has a clear role in substrate channeling, the functional significance of the tetramer is unknown. To address this question, we performed structural studies of four-type A PutAs from two clades of the PutA tree. The crystal structure of Bdellovibrio bacteriovorus PutA covalently inactivated by N-propargylglycine revealed a fold and substrate-channeling tunnel similar to other PutAs. Small-angle X-ray scattering (SAXS) and analytical ultracentrifugation indicated that Bdellovibrio PutA is dimeric in solution, in contrast to the prediction from crystal packing of a stable tetrameric assembly. SAXS studies of two other type A PutAs from separate clades also suggested that the dimer predominates in solution. To assess whether the tetramer of B. japonicum PutA is necessary for catalytic function, a hot spot disruption mutant that cleanly produces dimeric protein was generated. The dimeric variant exhibited kinetic parameters similar to the wild-type enzyme. These results implicate the domain-swapped dimer as the core structural and functional unit of type A PutAs. ENZYMES: Proline dehydrogenase (EC 1.5.5.2); l-glutamate-γ-semialdehyde dehydrogenase (EC 1.2.1.88). DATABASES: The atomic coordinates and structure factor amplitudes have been deposited in the Protein Data Bank under accession number 5UR2. The SAXS data have been deposited in the SASBDB under the ...DATABASES: The atomic coordinates and structure factor amplitudes have been deposited in the Protein Data Bank under accession number 5UR2. The SAXS data have been deposited in the SASBDB under the following accession codes: SASDCP3 (BbPutA), SASDCQ3 (DvPutA 1.5 mg·mL ), SASDCX3 (DvPutA 3.0 mg·mL ), SASDCY3 (DvPutA 4.5 mg·mL ), SASDCR3 (LpPutA 3.0 mg·mL ), SASDCV3 (LpPutA 5.0 mg·mL ), SASDCW3 (LpPutA 8.0 mg·mL ), SASDCS3 (BjPutA 2.3 mg·mL ), SASDCT3 (BjPutA 4.7 mg·mL ), SASDCU3 (BjPutA 7.0 mg·mL ), SASDCZ3 (R51E 2.3 mg·mL ), SASDC24 (R51E 4.7 mg·mL ), SASDC34 (R51E 7.0 mg·mL ). |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-Data source
| SASBDBのページ |  SASDCY3 SASDCY3 |
|---|
-関連構造データ
| 関連構造データ |  5ur2C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- 外部リンク
外部リンク
| 「今月の分子」の関連する項目 |
|---|
-モデル
- 試料
試料
 試料 試料 | 名称: Proline utilization A from Desulfovibrio vulgaris 4.5 mg/mL 試料濃度: 4.5 mg/ml |
|---|---|
| バッファ | 名称: 50 mM Tris-HCl, 50 mM NaCl, 0.5 mM EDTA, and 0.5 mM THP at pH 7.5. pH: 7.5 |
| 要素 #679 | 名称: DvPutA / タイプ: protein / 記述: Bifunctional protein PutA / 分子量: 114.496 / 分子数: 2 / 由来: Desulfovibrio vulgaris (strain RCH1) / 参照: UniProt: A0A0E0T6R2 配列: MHHHHHHSSG VDLGTENLYF QSMDQQHLDG KVVERGKEFF RSISGEAPSI FNKGWWTGKV MDWAMQNEDF KVQLFRFVDV LPYLNTSESL LRHIREYFAT EDADIPPVLK WGAGKAGIGG ALTAKLMGMT IRSNIEGMAR QFIIGDNSKE AVKGLAKLRK DGFTFTVDLL ...配列: MHHHHHHSSG VDLGTENLYF QSMDQQHLDG KVVERGKEFF RSISGEAPSI FNKGWWTGKV MDWAMQNEDF KVQLFRFVDV LPYLNTSESL LRHIREYFAT EDADIPPVLK WGAGKAGIGG ALTAKLMGMT IRSNIEGMAR QFIIGDNSKE AVKGLAKLRK DGFTFTVDLL GEATVSEEES EAYAQGYHEV VDAIAREQEK WKALPGNGPV EGFDWGATPK VNVSIKPSAL YSQAKPVDVE GSVRGILSRL VPIYRKVVAM GGFLCIDMEQ LKYKEMTLEL FKRLRSDPEF RHYPHLSIVL QAYLRDTEKD LDDLLHWARS EKLPIGIRLV KGAYWDYETV IAKQNGWEIP VWTDKPESDI AYEKLAHRIL ENSDIVYFAC ASHNVRTIAA VMETALALNV PEHRYEFQVL YGMAEPVRKG LKNVAGRVRL YCPYGELIPG MAYLVRRLLE NTANESFLRQ SFAEGAALER LLENPQKTLH RLLAARPEPR AVEPGPGGLP PFTNDAMIDF TVPDNRKAFV EALADVRSRF GQTVPLYIGG RDVTTADLIP TTNPAKPAEV VASICQAGRP EIDDAIAAAK KAALTWRDTS PADRAAYLRR AADICRKRIW ELSAWQVVEV GKQWDQAYHD VTEGIDFLEY YAREMLRLGA PRRMGRAPGE HNHLFYQPKG IAAVIAPWNF PFAIAIGMAS AAIVTGNPVI FKPSSISSRI GYNLAEVFRE AGLPEGVFNY CPGRSSIMGD YLVEHPDISL ICFTGSMEVG LRIQEKAAKV QPGQRQCKRV IAEMGGKNAT IIDDDADLDE AVLQVLYSAF GFQGQKCSAC SRVIVLDAIY DRFIERLVKA ASSIHIGPSE DPSNYMGPVA DATLQKNVSD YIRIAEEEGR VLLKRTDLPA EGCYVPLTIV GDIRPEHRIA QEEIFGPVLA VMRAATFDEA LSIANGTRFA LTGAVFSRSP EHLDKARREF RVGNLYLNKG STGALVERQP FGGFAMSGVG SKTGGPDYLL QFMDPRVVTE NTMRRGFTPI DEDDDWIV |
-実験情報
| ビーム | 設備名称: Advanced Light Source (ALS) 12.3.1 (SIBYLS) / 地域: Berkeley, CA / 国: USA  / 線源: X-ray synchrotron / 線源: X-ray synchrotron | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: MAR 165 CCD | |||||||||||||||||||||||||||||||||
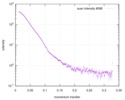
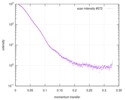
| スキャン | 測定日: 2012年6月8日 / 単位: 1/A /
| |||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー