+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6i00 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
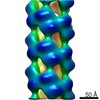
| タイトル | Cryo-EM informed directed evolution of Nitrilase 4 leads to a change in quaternary structure. | |||||||||
 要素 要素 | Bifunctional nitrilase/nitrile hydratase NIT4 | |||||||||
 キーワード キーワード | HYDROLASE / NITRILASE / Cyanide detoxification / Beta-cyano-L-alanine hydrolase / Helical filament | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cyanoalanine nitrilase / 3-cyanoalanine hydratase / cyanoalanine nitrilase activity / 3-cyanoalanine hydratase activity / cyanide metabolic process / nitrilase / indole-3-acetonitrile nitrilase activity / detoxification of nitrogen compound / nitrilase activity / nitrile hydratase activity ...cyanoalanine nitrilase / 3-cyanoalanine hydratase / cyanoalanine nitrilase activity / 3-cyanoalanine hydratase activity / cyanide metabolic process / nitrilase / indole-3-acetonitrile nitrilase activity / detoxification of nitrogen compound / nitrilase activity / nitrile hydratase activity / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子顕微鏡法 / らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Mulelu, A.E. / Woodward, J.D. | |||||||||
| 資金援助 |  南アフリカ, 南アフリカ,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2019 ジャーナル: Commun Biol / 年: 2019タイトル: Cryo-EM and directed evolution reveal how nitrilase specificity is influenced by its quaternary structure. 著者: Andani E Mulelu / Angela M Kirykowicz / Jeremy D Woodward /  要旨: Nitrilases are helical enzymes that convert nitriles to acids and/or amides. All plants have a nitrilase 4 homolog specific for ß-cyanoalanine, while in some plants neofunctionalization has produced ...Nitrilases are helical enzymes that convert nitriles to acids and/or amides. All plants have a nitrilase 4 homolog specific for ß-cyanoalanine, while in some plants neofunctionalization has produced nitrilases with altered specificity. Plant nitrilase substrate size and specificity correlate with helical twist, but molecular details of this relationship are lacking. Here we determine, to our knowledge, the first close-to-atomic resolution (3.4 Å) cryo-EM structure of an active helical nitrilase, the nitrilase 4 from . We apply site-saturation mutagenesis directed evolution to three residues (R95, S224, and L169) and generate a mutant with an altered helical twist that accepts substrates not catalyzed by known plant nitrilases. We reveal that a loop between α2 and α3 limits the length of the binding pocket and propose that it shifts position as a function of helical twist. These insights will allow us to start designing nitrilases for chemoenzymatic synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6i00.cif.gz 6i00.cif.gz | 665.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6i00.ent.gz pdb6i00.ent.gz | 553.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6i00.json.gz 6i00.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6i00_validation.pdf.gz 6i00_validation.pdf.gz | 431.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6i00_full_validation.pdf.gz 6i00_full_validation.pdf.gz | 467 KB | 表示 | |
| XML形式データ |  6i00_validation.xml.gz 6i00_validation.xml.gz | 68.1 KB | 表示 | |
| CIF形式データ |  6i00_validation.cif.gz 6i00_validation.cif.gz | 102.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i0/6i00 https://data.pdbj.org/pub/pdb/validation_reports/i0/6i00 ftp://data.pdbj.org/pub/pdb/validation_reports/i0/6i00 ftp://data.pdbj.org/pub/pdb/validation_reports/i0/6i00 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 39766.113 Da / 分子数: 12 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: NIT4, At5g22300, MWD9.8 発現宿主:  参照: UniProt: P46011, nitrilase, cyanoalanine nitrilase, 3-cyanoalanine hydratase |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: らせん対称体再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Active helical nitrilase complex / タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) | 生物種:  | |||||||||||||||
| 由来(組換発現) | 生物種:  | |||||||||||||||
| 緩衝液 | pH: 8 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 0.15 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: Quantifoil, UltrAuFoil, R1.2/1.3 | |||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK I / 凍結剤: ETHANE / 湿度: 50 % / 凍結前の試料温度: 293 K 詳細: The sample (2.5 ul) was applied to the grid and incubated for 30 seconds at 50% humidity before blotting and plunging. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 750 nm / Cs: 2.7 mm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 7 sec. / 電子線照射量: 45.5 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 1266 |
| 画像スキャン | サンプリングサイズ: 5 µm / 横: 3838 / 縦: 3710 / 動画フレーム数/画像: 45 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| らせん対称 | 回転角度/サブユニット: -72.98 ° / 軸方向距離/サブユニット: 17.48 Å / らせん対称軸の対称性: C1 | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 392263 | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 133106 / クラス平均像の数: 67 / 対称性のタイプ: HELICAL | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: REAL / Target criteria: Cross-correlation coefficient 詳細: Ab initio fitting was performed using Buccaneer, cleaned up with Coot and Phenix Real-Space Refine. |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj