+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7065 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human insulin degrading enzyme in complex with insulin | |||||||||||||||
 マップデータ マップデータ | Insulin degrading enzyme in complex with insulin | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | IDE / insulin degrading enzyme / amyloid beta / BIOSYNTHETIC PROTEIN / HYDROLASE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報insulysin / ubiquitin recycling / insulin catabolic process / insulin metabolic process / amyloid-beta clearance by cellular catabolic process / hormone catabolic process / bradykinin catabolic process / insulin binding / regulation of aerobic respiration / peptide catabolic process ...insulysin / ubiquitin recycling / insulin catabolic process / insulin metabolic process / amyloid-beta clearance by cellular catabolic process / hormone catabolic process / bradykinin catabolic process / insulin binding / regulation of aerobic respiration / peptide catabolic process / amyloid-beta clearance / peroxisomal matrix / amyloid-beta metabolic process / Insulin receptor recycling / proteolysis involved in protein catabolic process / Peroxisomal protein import / peptide binding / protein catabolic process / antigen processing and presentation of endogenous peptide antigen via MHC class I / metalloendopeptidase activity / positive regulation of protein catabolic process / peroxisome / positive regulation of protein binding / insulin receptor signaling pathway / virus receptor activity / basolateral plasma membrane / endopeptidase activity / Ub-specific processing proteases / external side of plasma membrane / cell surface / protein homodimerization activity / mitochondrion / proteolysis / extracellular space / extracellular exosome / zinc ion binding / ATP binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
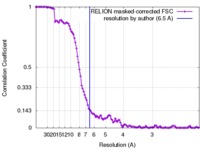
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Zhang Z / Liang WG / Bailey LJ / Tan YZ / Wei H / Kossiakoff AA / Carragher B / Potter SC / Tang WJ | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: Ensemble cryoEM elucidates the mechanism of insulin capture and degradation by human insulin degrading enzyme. 著者: Zhening Zhang / Wenguang G Liang / Lucas J Bailey / Yong Zi Tan / Hui Wei / Andrew Wang / Mara Farcasanu / Virgil A Woods / Lauren A McCord / David Lee / Weifeng Shang / Rebecca Deprez- ...著者: Zhening Zhang / Wenguang G Liang / Lucas J Bailey / Yong Zi Tan / Hui Wei / Andrew Wang / Mara Farcasanu / Virgil A Woods / Lauren A McCord / David Lee / Weifeng Shang / Rebecca Deprez-Poulain / Benoit Deprez / David R Liu / Akiko Koide / Shohei Koide / Anthony A Kossiakoff / Sheng Li / Bridget Carragher / Clinton S Potter / Wei-Jen Tang /   要旨: Insulin degrading enzyme (IDE) plays key roles in degrading peptides vital in type two diabetes, Alzheimer's, inflammation, and other human diseases. However, the process through which IDE recognizes ...Insulin degrading enzyme (IDE) plays key roles in degrading peptides vital in type two diabetes, Alzheimer's, inflammation, and other human diseases. However, the process through which IDE recognizes peptides that tend to form amyloid fibrils remained unsolved. We used cryoEM to understand both the apo- and insulin-bound dimeric IDE states, revealing that IDE displays a large opening between the homologous ~55 kDa N- and C-terminal halves to allow selective substrate capture based on size and charge complementarity. We also used cryoEM, X-ray crystallography, SAXS, and HDX-MS to elucidate the molecular basis of how amyloidogenic peptides stabilize the disordered IDE catalytic cleft, thereby inducing selective degradation by substrate-assisted catalysis. Furthermore, our insulin-bound IDE structures explain how IDE processively degrades insulin by stochastically cutting either chain without breaking disulfide bonds. Together, our studies provide a mechanism for how IDE selectively degrades amyloidogenic peptides and offers structural insights for developing IDE-based therapies. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7065.map.gz emd_7065.map.gz | 8.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7065-v30.xml emd-7065-v30.xml emd-7065.xml emd-7065.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7065_fsc.xml emd_7065_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7065.png emd_7065.png | 66.1 KB | ||
| マスクデータ |  emd_7065_msk_1.map emd_7065_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-7065.cif.gz emd-7065.cif.gz | 6.9 KB | ||
| その他 |  emd_7065_half_map_1.map.gz emd_7065_half_map_1.map.gz emd_7065_half_map_2.map.gz emd_7065_half_map_2.map.gz | 98.3 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7065 http://ftp.pdbj.org/pub/emdb/structures/EMD-7065 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7065 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7065 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7065_validation.pdf.gz emd_7065_validation.pdf.gz | 741.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7065_full_validation.pdf.gz emd_7065_full_validation.pdf.gz | 740.7 KB | 表示 | |
| XML形式データ |  emd_7065_validation.xml.gz emd_7065_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_7065_validation.cif.gz emd_7065_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7065 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7065 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7065 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7065 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6b7yMC  7041C  7062C  7066C  7090C  7091C  7092C  7093C  5wobC  6b3qC  6b70C  6b7zC  6bf6C  6bf7C  6bf8C  6bf9C  6bfcC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7065.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7065.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Insulin degrading enzyme in complex with insulin | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.073 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_7065_msk_1.map emd_7065_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Insulin degrading enzyme in complex with insulin
| ファイル | emd_7065_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Insulin degrading enzyme in complex with insulin | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Insulin degrading enzyme in complex with insulin
| ファイル | emd_7065_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Insulin degrading enzyme in complex with insulin | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Insulin degrading enzyme
| 全体 | 名称: Insulin degrading enzyme |
|---|---|
| 要素 |
|
-超分子 #1: Insulin degrading enzyme
| 超分子 | 名称: Insulin degrading enzyme / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Cryo-EM structure of human Apo insulin degrading enzyme |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 100 KDa |
-分子 #1: Insulin-degrading enzyme
| 分子 | 名称: Insulin-degrading enzyme / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: insulysin |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 111.866484 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AIKRIGNHIT KSPEDKREYR GLELANGIKV LLISDPTTDK SSAALDVHIG SLSDPPNIAG LSHFLEHMLF LGTKKYPKEN EYSQFLSEH AGSSNAFTSG EHTNYYFDVS HEHLEGALDR FAQFFLSPLF DESAKDREVN AVDSEHEKNV MNDAWRLFQL E KATGNPKH ...文字列: AIKRIGNHIT KSPEDKREYR GLELANGIKV LLISDPTTDK SSAALDVHIG SLSDPPNIAG LSHFLEHMLF LGTKKYPKEN EYSQFLSEH AGSSNAFTSG EHTNYYFDVS HEHLEGALDR FAQFFLSPLF DESAKDREVN AVDSEHEKNV MNDAWRLFQL E KATGNPKH PFSKFGTGNK YTLETRPNQE GIDVRQELLK FHSAYYSSNL MAVVVLGRES LDDLTNLVVK LFSEVENKNV PL PEFPEHP FQEEHLKQLY KIVPIKDIRN LYVTFPIPDL QKYYKSNPGH YLGHLIGHEG PGSLLSELKS KGWVNTLVGG QKE GARGFM FFIINVDLTE EGLLHVEDII LHMFQYIQKL RAEGPQEWVF QELKDLNAVA FRFKDKERPR GYTSKIAGIL HYYP LEEVL TAEYLLEEFR PDLIEMVLDK LRPENVRVAI VSKSFEGKTD RTEEWYGTQY KQEAIPDEVI KKWQNADLNG KFKLP TKNE FIPTNFEILP LEKEATPYPA LIKDTAMSKL WFKQDDKFFL PKANLNFEFF SPFAYVDPLH SNMAYLYLEL LKDSLN EYA YAAELAGLSY DLQNTIYGMY LSVKGYNDKQ PILLKKIIEK MATFEIDEKR FEIIKEAYMR SLNNFRAEQP HQHAMYY LR LLMTEVAWTK DELKEALDDV TLPRLKAFIP QLLSRLHIEA LLHGNITKQA ALGIMQMVED TLIEHAHTKP LLPSQLVR Y REVQLPDRGW FVYQQRNEVH NNSGIEIYYQ TDMQSTSENM FLELFAQIIS EPAFNTLRTK EQLGYIVFSG PRRANGIQG LRFIIQSEKP PHYLESRVEA FLITMEKSIE DMTEEAFQKH IQALAIRRLD KPKKLSAESA KYWGEIISQQ YNFDRDNTEV AYLKTLTKE DIIKFYKEML AVDAPRRHKV SVHVLAREMD SCPVVGEFPC QNDINLSQAP ALPQPEVIQN MTEFKRGLPL F PLVKPH UniProtKB: Insulin-degrading enzyme |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||
| グリッド | モデル: Homemade / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 10 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.001 kPa / 詳細: The grids are homemade lacey gold nanowire grids | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 298 K / 装置: HOMEMADE PLUNGER / 詳細: The cryo grids were made using Spotiton. | ||||||||||||
| 詳細 | The sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| アライメント法 | Coma free - Residual tilt: 10.0 mrad |
| 詳細 | The image was collected at 20-50 degree tilt |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: COUNTING / #0 - 撮影したグリッド数: 1 / #0 - 実像数: 509 / #0 - 平均露光時間: 10.0 sec. / #0 - 平均電子線量: 7.9 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 検出モード: COUNTING / #1 - デジタル化 - サイズ - 横: 3710 pixel / #1 - デジタル化 - サイズ - 縦: 3838 pixel / #1 - デジタル化 - 画像ごとのフレーム数: 1-50 / #1 - 撮影したグリッド数: 1 / #1 - 実像数: 620 / #1 - 平均露光時間: 10.0 sec. / #1 - 平均電子線量: 6.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 46598 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm 最小 デフォーカス(公称値): 0.9400000000000001 µm 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 92 |
|---|---|
| 得られたモデル |  PDB-6b7y: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)