+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6121 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of quasi-HPV16 complexed with H16.14J Fab | |||||||||
 マップデータ マップデータ | Reconstruction of quasi-HPV16 complexed with H16.14J Fab | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | quasi-HPV16 / L1 capsomer / H16.14J Fab | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=7 icosahedral viral capsid / endocytosis involved in viral entry into host cell / host cell nucleus / virion attachment to host cell / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human papillomavirus 16 (パピローマウイルス) Human papillomavirus 16 (パピローマウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 13.9 Å | |||||||||
 データ登録者 データ登録者 | Guan J / Bywaters SM / Brendle SA / Lee H / Ashley RE / Conway JF / Makhov AM / Christensen ND / Hafenstein S | |||||||||
 引用 引用 |  ジャーナル: Virology / 年: 2015 ジャーナル: Virology / 年: 2015タイトル: Structural comparison of four different antibodies interacting with human papillomavirus 16 and mechanisms of neutralization. 著者: Jian Guan / Stephanie M Bywaters / Sarah A Brendle / Hyunwook Lee / Robert E Ashley / Alexander M Makhov / James F Conway / Neil D Christensen / Susan Hafenstein /  要旨: Cryo-electron microscopy (cryo-EM) was used to solve the structures of human papillomavirus type 16 (HPV16) complexed with fragments of antibody (Fab) from three different neutralizing monoclonals ...Cryo-electron microscopy (cryo-EM) was used to solve the structures of human papillomavirus type 16 (HPV16) complexed with fragments of antibody (Fab) from three different neutralizing monoclonals (mAbs): H16.1A, H16.14J, and H263.A2. The structure-function analysis revealed predominantly monovalent binding of each Fab with capsid interactions that involved multiple loops from symmetry related copies of the major capsid protein. The residues identified in each Fab-virus interface map to a conformational groove on the surface of the capsomer. In addition to the known involvement of the FG and HI loops, the DE loop was also found to constitute the core of each epitope. Surprisingly, the epitope mapping also identified minor contributions by EF and BC loops. Complementary immunological assays included mAb and Fab neutralization. The specific binding characteristics of mAbs correlated with different neutralizing behaviors in pre- and post-attachment neutralization assays. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6121.map.gz emd_6121.map.gz | 127 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6121-v30.xml emd-6121-v30.xml emd-6121.xml emd-6121.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6121.tif emd_6121.tif | 1000.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6121 http://ftp.pdbj.org/pub/emdb/structures/EMD-6121 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6121 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6121 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6121_validation.pdf.gz emd_6121_validation.pdf.gz | 320.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6121_full_validation.pdf.gz emd_6121_full_validation.pdf.gz | 320 KB | 表示 | |
| XML形式データ |  emd_6121_validation.xml.gz emd_6121_validation.xml.gz | 8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6121 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6121 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6121 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6121 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6121.map.gz / 形式: CCP4 / 大きさ: 379.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6121.map.gz / 形式: CCP4 / 大きさ: 379.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of quasi-HPV16 complexed with H16.14J Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
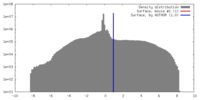
| ボクセルのサイズ | X=Y=Z: 2.33 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Quasi-human papillomavirus 16 complexed with H16.14J Fab
| 全体 | 名称: Quasi-human papillomavirus 16 complexed with H16.14J Fab |
|---|---|
| 要素 |
|
-超分子 #1000: Quasi-human papillomavirus 16 complexed with H16.14J Fab
| 超分子 | 名称: Quasi-human papillomavirus 16 complexed with H16.14J Fab タイプ: sample / ID: 1000 / 集合状態: 300 H16.14J Fabs bind to one HPV16 capsid / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 44.7 MDa |
-超分子 #1: Human papillomavirus 16
| 超分子 | 名称: Human papillomavirus 16 / タイプ: virus / ID: 1 / NCBI-ID: 337041 / 生物種: Human papillomavirus 16 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| Host system | 生物種:  Homo sapiens (ヒト) / 組換細胞: 293TT / 組換プラスミド: SV40 Homo sapiens (ヒト) / 組換細胞: 293TT / 組換プラスミド: SV40 |
| 分子量 | 理論値: 27.7 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: L1 L2 / 直径: 600 Å / T番号(三角分割数): 7 |
-分子 #1: H16.14J Fab
| 分子 | 名称: H16.14J Fab / タイプ: protein_or_peptide / ID: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 1 M NaCl, 200 mM Tris |
| グリッド | 詳細: glow-discharged holey carbon support grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 102 K / 装置: GATAN CRYOPLUNGE 3 / 手法: Blot for 0.7 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100 |
|---|---|
| 温度 | 平均: 95 K |
| 日付 | 2014年8月27日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 385 / 平均電子線量: 15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 4.69 µm / 最小 デフォーカス(公称値): 0.62 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | The particles were selected using semi-automatic program e2boxer.py (EMAN2). |
|---|---|
| CTF補正 | 詳細: Each particle |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 13.9 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Auto3dem 詳細: Semi-automatic particle selection was performed using e2boxer.py to obtain the particle coordinates, followed by particle boxing, linearization, normalization, and apodization of the images ...詳細: Semi-automatic particle selection was performed using e2boxer.py to obtain the particle coordinates, followed by particle boxing, linearization, normalization, and apodization of the images using Robem. Defocus and astigmatism values necessary to perform contrast transfer function (CTF) correction for the extracted particles were assessed using Robem. The icosahedrally averaged reconstruction was initiated using a random model generated with setup_rmc. For the last step of refinement, the final maps were CTF-corrected using a B factor of 200 A2. 使用した粒子像数: 5642 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera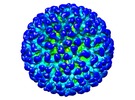

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)