+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31954 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Vaccinia virus scaffolding protein D13 trimer sextet | ||||||||||||
 マップデータ マップデータ | Cryo-EM single particle reconstruction on Vaccinia virus scaffold protein D13 trimer sextet. The map has been sharpened using Relion post-processing, and density-normalized (mean=0, s.d=1). | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Scaffold / capsid / double-jelly-roll / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 | Poxvirus rifampicin-resistance / Poxvirus rifampicin resistance protein / response to antibiotic / identical protein binding / membrane / Scaffold protein OPG125 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Vaccinia virus WR (ウイルス) / Vaccinia virus WR (ウイルス) /  Vaccinia virus (strain Western Reserve) (ウイルス) Vaccinia virus (strain Western Reserve) (ウイルス) | ||||||||||||
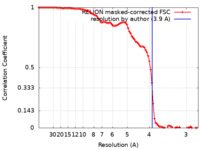
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||||||||
 データ登録者 データ登録者 | Wolf M / Hyun J | ||||||||||||
| 資金援助 |  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Assembly mechanism of the pleomorphic immature poxvirus scaffold. 著者: Jaekyung Hyun / Hideyuki Matsunami / Tae Gyun Kim / Matthias Wolf /    要旨: In Vaccinia virus (VACV), the prototype poxvirus, scaffold protein D13 forms a honeycomb-like lattice on the viral membrane that results in formation of the pleomorphic immature virion (IV). The ...In Vaccinia virus (VACV), the prototype poxvirus, scaffold protein D13 forms a honeycomb-like lattice on the viral membrane that results in formation of the pleomorphic immature virion (IV). The structure of D13 is similar to those of major capsid proteins that readily form icosahedral capsids in nucleocytoplasmic large DNA viruses (NCLDVs). However, the detailed assembly mechanism of the nonicosahedral poxvirus scaffold has never been understood. Here we show the cryo-EM structures of the D13 trimer and scaffold intermediates produced in vitro. The structures reveal that the displacement of the short N-terminal α-helix is critical for initiation of D13 self-assembly. The continuous curvature of the IV is mediated by electrostatic interactions that induce torsion between trimers. The assembly mechanism explains the semiordered capsid-like arrangement of D13 that is distinct from icosahedral NCLDVs. Our structures explain how a single protein can self-assemble into different capsid morphologies and represent a local exception to the universal Caspar-Klug theory of quasi-equivalence. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31954.map.gz emd_31954.map.gz | 9.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31954-v30.xml emd-31954-v30.xml emd-31954.xml emd-31954.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31954_fsc.xml emd_31954_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31954.png emd_31954.png | 165.9 KB | ||
| マスクデータ |  emd_31954_msk_1.map emd_31954_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-31954.cif.gz emd-31954.cif.gz | 7 KB | ||
| その他 |  emd_31954_additional_1.map.gz emd_31954_additional_1.map.gz emd_31954_additional_2.map.gz emd_31954_additional_2.map.gz emd_31954_half_map_1.map.gz emd_31954_half_map_1.map.gz emd_31954_half_map_2.map.gz emd_31954_half_map_2.map.gz | 59.2 MB 26 MB 28.3 MB 28.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31954 http://ftp.pdbj.org/pub/emdb/structures/EMD-31954 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31954 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31954 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31954_validation.pdf.gz emd_31954_validation.pdf.gz | 927.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31954_full_validation.pdf.gz emd_31954_full_validation.pdf.gz | 927.2 KB | 表示 | |
| XML形式データ |  emd_31954_validation.xml.gz emd_31954_validation.xml.gz | 17.1 KB | 表示 | |
| CIF形式データ |  emd_31954_validation.cif.gz emd_31954_validation.cif.gz | 21.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31954 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31954 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31954 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31954 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31954.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31954.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM single particle reconstruction on Vaccinia virus scaffold protein D13 trimer sextet. The map has been sharpened using Relion post-processing, and density-normalized (mean=0, s.d=1). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.39 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_31954_msk_1.map emd_31954_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM single particle reconstruction on Vaccinia virus scaffold...
| ファイル | emd_31954_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM single particle reconstruction on Vaccinia virus scaffold protein D13 trimer sextet. The map is unsharpened, and density-normalized (mean=0, s.d=1). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM single particle reconstruction on Vaccinia virus scaffold...
| ファイル | emd_31954_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM single particle reconstruction on Vaccinia virus scaffold protein D13 trimer sextet. The map has been density-modified using PHENIX ResolveCryoEM, and density-normalized (mean=0, s.d=1). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM single particle reconstruction on Vaccinia virus scaffold...
| ファイル | emd_31954_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM single particle reconstruction on Vaccinia virus scaffold protein D13 trimer sextet (half map 1). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM single particle reconstruction on Vaccinia virus scaffold...
| ファイル | emd_31954_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM single particle reconstruction on Vaccinia virus scaffold protein D13 trimer sextet (half map 2). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vaccinia virus scaffolding protein D13 with N-terminal 17 residue...
| 全体 | 名称: Vaccinia virus scaffolding protein D13 with N-terminal 17 residue truncation, in its trimer sextet oligomeric state |
|---|---|
| 要素 |
|
-超分子 #1: Vaccinia virus scaffolding protein D13 with N-terminal 17 residue...
| 超分子 | 名称: Vaccinia virus scaffolding protein D13 with N-terminal 17 residue truncation, in its trimer sextet oligomeric state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Recombinant D13 was expressed with N-terminal polyhistidine-tag (His-tag) using bacterial expression system. The protein was purified using metal affinity chromatography. His-tag was removed ...詳細: Recombinant D13 was expressed with N-terminal polyhistidine-tag (His-tag) using bacterial expression system. The protein was purified using metal affinity chromatography. His-tag was removed by proteolysis and the protein was further purified using size exclusion chromatography. The final purified protein was trimeric. D13 assembled in tubular objects in low salt buffer. |
|---|---|
| 由来(天然) | 生物種:  Vaccinia virus WR (ウイルス) Vaccinia virus WR (ウイルス) |
| 分子量 | 理論値: 1.080 MDa |
-分子 #1: Scaffold protein D13
| 分子 | 名称: Scaffold protein D13 / タイプ: protein_or_peptide / ID: 1 / コピー数: 18 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vaccinia virus (strain Western Reserve) (ウイルス) Vaccinia virus (strain Western Reserve) (ウイルス)株: Western Reserve |
| 分子量 | 理論値: 59.817219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RSNVFAVDSQ IPTLYMPQYI SLSGVMTNDG PDNQAIASFE IRDQYITALN HLVLSLELPE VKGMGRFGYV PYVGYKCINH VSISSCNGV IWEIEGEELY NNCINNTIAL KHSGYSSELN DISIGLTPND TIKEPSTVYV YIKTPFDVED TFSSLKLSDS K ITVTVTFN ...文字列: RSNVFAVDSQ IPTLYMPQYI SLSGVMTNDG PDNQAIASFE IRDQYITALN HLVLSLELPE VKGMGRFGYV PYVGYKCINH VSISSCNGV IWEIEGEELY NNCINNTIAL KHSGYSSELN DISIGLTPND TIKEPSTVYV YIKTPFDVED TFSSLKLSDS K ITVTVTFN PVSDIVIRDS SFDFETFNKE FVYVPELSFI GYMVKNVQIK PSFIEKPRRV IGQINQPTAT VTEVHAATSL SV YTKPYYG NTDNKFISYP GYSQDEKDYI DAYVSRLLDD LVIVSDGPPT GYPESAEIVE VPEDGIVSIQ DADVYVKIDN VPD NMSVYL HTNLLMFGTR KNSFIYNISK KFSAITGTYS DATKRTIFAH ISHSINIIDT SIPVSLWTSQ RNVYNGDNRS AESK AKDLF INDPFIKGID FKNKTDIISR LEVRFGNDVL YSENGPISRI YNELLTKSNN GTRTLTFNFT PKIFFRPTTI TANVS RGKD KLSVRVVYST MDVNHPIYYV QKQLVVVCND LYKVSYDQGV SITKIMG UniProtKB: Scaffold protein OPG125 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.12 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 3 microliter sample volume was loaded onto a holey grid with additional graphene oxide film. 10 sec waiting time, 5 sec blotting time and blot force 0, no delay time were applied before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 2 / 実像数: 7621 / 平均露光時間: 10.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)