[日本語] English
 万見
万見- EMDB-29406: Pseudomonas phage E217 5-fold vertex (capsid and decorating proteins) -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pseudomonas phage E217 5-fold vertex (capsid and decorating proteins) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Pseudomonas / phage / E217 / VIRUS / capsid / decorating proteins | |||||||||
| 機能・相同性 | : / Structural cement protein (E217 gp24/Pam3 gp6) / Capsid and scaffold protein / Virion protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas phage vB_PaeM_E217 (ファージ) Pseudomonas phage vB_PaeM_E217 (ファージ) | |||||||||
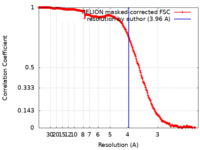
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.96 Å | |||||||||
 データ登録者 データ登録者 | Li F / Cingolani G / Hou C | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: High-resolution cryo-EM structure of the Pseudomonas bacteriophage E217. 著者: Fenglin Li / Chun-Feng David Hou / Ravi K Lokareddy / Ruoyu Yang / Francesca Forti / Federica Briani / Gino Cingolani /   要旨: E217 is a Pseudomonas phage used in an experimental cocktail to eradicate cystic fibrosis-associated Pseudomonas aeruginosa. Here, we describe the structure of the whole E217 virion before and after ...E217 is a Pseudomonas phage used in an experimental cocktail to eradicate cystic fibrosis-associated Pseudomonas aeruginosa. Here, we describe the structure of the whole E217 virion before and after DNA ejection at 3.1 Å and 4.5 Å resolution, respectively, determined using cryogenic electron microscopy (cryo-EM). We identify and build de novo structures for 19 unique E217 gene products, resolve the tail genome-ejection machine in both extended and contracted states, and decipher the complete architecture of the baseplate formed by 66 polypeptide chains. We also determine that E217 recognizes the host O-antigen as a receptor, and we resolve the N-terminal portion of the O-antigen-binding tail fiber. We propose that E217 design principles presented in this paper are conserved across PB1-like Myoviridae phages of the Pbunavirus genus that encode a ~1.4 MDa baseplate, dramatically smaller than the coliphage T4. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29406.map.gz emd_29406.map.gz | 407.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29406-v30.xml emd-29406-v30.xml emd-29406.xml emd-29406.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29406_fsc.xml emd_29406_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29406.png emd_29406.png | 956.2 KB | ||
| Filedesc metadata |  emd-29406.cif.gz emd-29406.cif.gz | 5.8 KB | ||
| その他 |  emd_29406_half_map_1.map.gz emd_29406_half_map_1.map.gz emd_29406_half_map_2.map.gz emd_29406_half_map_2.map.gz | 411.7 MB 411.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29406 http://ftp.pdbj.org/pub/emdb/structures/EMD-29406 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29406 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29406 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29406_validation.pdf.gz emd_29406_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29406_full_validation.pdf.gz emd_29406_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_29406_validation.xml.gz emd_29406_validation.xml.gz | 26.1 KB | 表示 | |
| CIF形式データ |  emd_29406_validation.cif.gz emd_29406_validation.cif.gz | 34.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29406 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29406 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29406 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29406 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29406.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29406.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_29406_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_29406_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pseudomonas phage vB_PaeM_E217
| 全体 | 名称:  Pseudomonas phage vB_PaeM_E217 (ファージ) Pseudomonas phage vB_PaeM_E217 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Pseudomonas phage vB_PaeM_E217
| 超分子 | 名称: Pseudomonas phage vB_PaeM_E217 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2034346 / 生物種: Pseudomonas phage vB_PaeM_E217 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|
-分子 #1: Major structural protein
| 分子 | 名称: Major structural protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage vB_PaeM_E217 (ファージ) Pseudomonas phage vB_PaeM_E217 (ファージ) |
| 分子量 | 理論値: 34.735047 KDa |
| 配列 | 文字列: NFTAPVTTPS IPTPIQFLQT WLPGFVKVMT AARKIDEIIG IDTVGSWEDQ EIVQGIVEPA GTAVEYGDHT NIPLTSWNAN FERRTIVRG ELGMMVGTLE EGRASAIRLN SAETKRQQAA IGLETFRNAI GFYGWQSGLG NRTYGFLNDP NLPAFQTPPS Q GWSTADWA ...文字列: NFTAPVTTPS IPTPIQFLQT WLPGFVKVMT AARKIDEIIG IDTVGSWEDQ EIVQGIVEPA GTAVEYGDHT NIPLTSWNAN FERRTIVRG ELGMMVGTLE EGRASAIRLN SAETKRQQAA IGLETFRNAI GFYGWQSGLG NRTYGFLNDP NLPAFQTPPS Q GWSTADWA GIIGDIREAV RQLRIQSQDQ IDPKAEKITL ALATSKVDYL SVTTPYGISV SDWIEQTYPK MRIVSAPELS GV QMKNQEP EDALVLFVED VNAAVDGSTD GGSVFSQLVQ SKFITLGVEK RAKSYVEDFS NGTAGALCKR PWAVVRYLGI UniProtKB: Capsid and scaffold protein |
-分子 #2: Structural protein gp24
| 分子 | 名称: Structural protein gp24 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage vB_PaeM_E217 (ファージ) Pseudomonas phage vB_PaeM_E217 (ファージ) |
| 分子量 | 理論値: 21.610312 KDa |
| 配列 | 文字列: MFQKQVYRQY TPGFPGDLIE DGPKRARPGR IMSLSAVNPA ATATGPNRAS RAFGYAGDVS ALGEGQPKTI AARASEVVIG GANFFGVLG HPKHYALFGS AGDSLAPSYD LPDGAEGEFF DMATGLVVEI FNGAAAALDL DYGDLVAYVP NNLATADDAL G LPAGALVG ...文字列: MFQKQVYRQY TPGFPGDLIE DGPKRARPGR IMSLSAVNPA ATATGPNRAS RAFGYAGDVS ALGEGQPKTI AARASEVVIG GANFFGVLG HPKHYALFGS AGDSLAPSYD LPDGAEGEFF DMATGLVVEI FNGAAAALDL DYGDLVAYVP NNLATADDAL G LPAGALVG FKTGSMPTGL VQIPNARIVN AISLPAQSAG NLVAGVTIVQ LTQ UniProtKB: Virion protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)