+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human liver glycogen phosphorylase | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Glycogen Phosphorylase / PLP / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報vitamin binding / purine nucleobase binding / 5-phosphoribose 1-diphosphate biosynthetic process / D-glucose binding / glycogen phosphorylase / glycogen phosphorylase activity / linear malto-oligosaccharide phosphorylase activity / SHG alpha-glucan phosphorylase activity / glycogen catabolic process / bile acid binding ...vitamin binding / purine nucleobase binding / 5-phosphoribose 1-diphosphate biosynthetic process / D-glucose binding / glycogen phosphorylase / glycogen phosphorylase activity / linear malto-oligosaccharide phosphorylase activity / SHG alpha-glucan phosphorylase activity / glycogen catabolic process / bile acid binding / Glycogen breakdown (glycogenolysis) / glycogen metabolic process / AMP binding / necroptotic process / response to bacterium / pyridoxal phosphate binding / glucose homeostasis / secretory granule lumen / ficolin-1-rich granule lumen / Neutrophil degranulation / extracellular exosome / extracellular region / ATP binding / identical protein binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.65 Å | |||||||||
 データ登録者 データ登録者 | Su C / Lyu M / Zhang Z / Yu EW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2023 ジャーナル: Cell Rep / 年: 2023タイトル: High-resolution structural-omics of human liver enzymes. 著者: Chih-Chia Su / Meinan Lyu / Zhemin Zhang / Masaru Miyagi / Wei Huang / Derek J Taylor / Edward W Yu /  要旨: We applied raw human liver microsome lysate to a holey carbon grid and used cryo-electron microscopy (cryo-EM) to define its composition. From this sample we identified and simultaneously determined ...We applied raw human liver microsome lysate to a holey carbon grid and used cryo-electron microscopy (cryo-EM) to define its composition. From this sample we identified and simultaneously determined high-resolution structural information for ten unique human liver enzymes involved in diverse cellular processes. Notably, we determined the structure of the endoplasmic bifunctional protein H6PD, where the N- and C-terminal domains independently possess glucose-6-phosphate dehydrogenase and 6-phosphogluconolactonase enzymatic activity, respectively. We also obtained the structure of heterodimeric human GANAB, an ER glycoprotein quality-control machinery that contains a catalytic α subunit and a noncatalytic β subunit. In addition, we observed a decameric peroxidase, PRDX4, which directly contacts a disulfide isomerase-related protein, ERp46. Structural data suggest that several glycosylations, bound endogenous compounds, and ions associate with these human liver enzymes. These results highlight the importance of cryo-EM in facilitating the elucidation of human organ proteomics at the atomic level. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
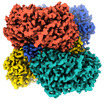
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28263.map.gz emd_28263.map.gz | 97.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28263-v30.xml emd-28263-v30.xml emd-28263.xml emd-28263.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28263_fsc.xml emd_28263_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28263.png emd_28263.png | 132.8 KB | ||
| マスクデータ |  emd_28263_msk_1.map emd_28263_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28263.cif.gz emd-28263.cif.gz | 5.5 KB | ||
| その他 |  emd_28263_half_map_1.map.gz emd_28263_half_map_1.map.gz emd_28263_half_map_2.map.gz emd_28263_half_map_2.map.gz | 95.4 MB 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28263 http://ftp.pdbj.org/pub/emdb/structures/EMD-28263 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28263 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28263 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28263_validation.pdf.gz emd_28263_validation.pdf.gz | 853.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28263_full_validation.pdf.gz emd_28263_full_validation.pdf.gz | 852.8 KB | 表示 | |
| XML形式データ |  emd_28263_validation.xml.gz emd_28263_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_28263_validation.cif.gz emd_28263_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28263 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28263 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28263 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28263 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8emsMC  7uzmC  8ekwC  8ekyC  8em2C  8emrC  8emtC  8eneC  8eojC  8eorC  23433 M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28263.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28263.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28263_msk_1.map emd_28263_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
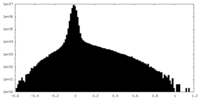
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28263_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
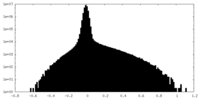
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28263_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Glycogen Phosphorylase
| 全体 | 名称: Glycogen Phosphorylase |
|---|---|
| 要素 |
|
-超分子 #1: Glycogen Phosphorylase
| 超分子 | 名称: Glycogen Phosphorylase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glycogen phosphorylase, liver form
| 分子 | 名称: Glycogen phosphorylase, liver form / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: glycogen phosphorylase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: liver Homo sapiens (ヒト) / 器官: liver |
| 分子量 | 理論値: 95.376406 KDa |
| 配列 | 文字列: MAKPLTDQEK RRQISIRGIV GVENVAELKK SFNRHLHFTL VKDRNVATTR DYYFALAHTV RDHLVGRWIR TQQHYYDKCP KRVYYLSLE FYMGRTLQNT MINLGLQNAC DEAIYQLGLD IEELEEIEED AGLGNGGLGR LAACFLDSMA TLGLAAYGYG I RYEYGIFN ...文字列: MAKPLTDQEK RRQISIRGIV GVENVAELKK SFNRHLHFTL VKDRNVATTR DYYFALAHTV RDHLVGRWIR TQQHYYDKCP KRVYYLSLE FYMGRTLQNT MINLGLQNAC DEAIYQLGLD IEELEEIEED AGLGNGGLGR LAACFLDSMA TLGLAAYGYG I RYEYGIFN QKIRDGWQVE EADDWLRYGN PWEKSRPEFM LPVHFYGKVE HTNTGTKWID TQVVLALPYD TPVPGYMNNT VN TMRLWSA RAPNDFNLRD FNVGDYIQAV LDRNLAENIS RVLYPNDNFF EGKELRLKQE YFVVAATLQD IIRRFKASKF GST RGAGTV FDAFPDQVAI QLNDTHPALA IPELMRIFVD IEKLPWSKAW ELTQKTFAYT NHTVLPEALE RWPVDLVEKL LPRH LEIIY EINQKHLDRI VALFPKDVDR LRRMSLIEEE GSKRINMAHL CIVGSHAVNG VAKIHSDIVK TKVFKDFSEL EPDKF QNKT NGITPRRWLL LCNPGLAELI AEKIGEDYVK DLSQLTKLHS FLGDDVFLRE LAKVKQENKL KFSQFLETEY KVKINP SSM FDVQVKRIHE YKRQLLNCLH VITMYNRIKK DPKKLFVPRT VIIGGKAAPG YHMAKMIIKL ITSVADVVNN DPMVGSK LK VIFLENYRVS LAEKVIPATD LSEQISTAGT EASGTGNMKF MLNGALTIGT MDGANVEMAE EAGEENLFIF GMRIDDVA A LDKKGYEAKE YYEALPELKL VIDQIDNGFF SPKQPDLFKD IINMLFYHDR FKVFADYEAY VKCQDKVSQL YMNPKAWNT MVLKNIAASG KFSSDRTIKE YAQNIWNVE UniProtKB: Glycogen phosphorylase, liver form |
-分子 #2: PYRIDOXAL-5'-PHOSPHATE
| 分子 | 名称: PYRIDOXAL-5'-PHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: PLP |
|---|---|
| 分子量 | 理論値: 247.142 Da |
| Chemical component information |  ChemComp-PLP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 41.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











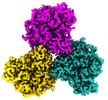




 Z
Z Y
Y X
X