+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23338 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
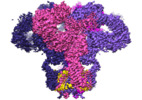
| タイトル | Structure of full-length IP3R1 channel solubilized in LNMG & lipid in the apo-state | ||||||||||||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of full-length type 1 IP3R in LMNG (apo-state) | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード |  Calcium channel (カルシウムチャネル) / Calcium channel (カルシウムチャネル) /  lipids (脂質) / lipids (脂質) /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / Elevation of cytosolic Ca2+ levels / cGMP effects / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane ...Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / Elevation of cytosolic Ca2+ levels / cGMP effects / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling / calcineurin complex / platelet dense granule membrane ...Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / Elevation of cytosolic Ca2+ levels / cGMP effects / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / regulation of postsynaptic cytosolic calcium ion concentration / voluntary musculoskeletal movement / calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / regulation of postsynaptic cytosolic calcium ion concentration / voluntary musculoskeletal movement /  inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / Ion homeostasis / dendrite development / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / GABA-ergic synapse / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol / inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / Ion homeostasis / dendrite development / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / GABA-ergic synapse / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol /  phosphatidylinositol binding / post-embryonic development / secretory granule membrane / phosphatidylinositol binding / post-embryonic development / secretory granule membrane /  筋小胞体 / 筋小胞体 /  synaptic membrane / calcium-mediated signaling / synaptic membrane / calcium-mediated signaling /  liver regeneration / calcium ion transmembrane transport / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of insulin secretion / positive regulation of neuron projection development / calcium ion transport / presynapse / liver regeneration / calcium ion transmembrane transport / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of insulin secretion / positive regulation of neuron projection development / calcium ion transport / presynapse /  核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / cellular response to hypoxia / postsynapse / 核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / cellular response to hypoxia / postsynapse /  protein phosphatase binding / transmembrane transporter binding / protein phosphatase binding / transmembrane transporter binding /  postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding / neuronal cell body / postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding / neuronal cell body /  シナプス / シナプス /  樹状突起 / 樹状突起 /  calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane / calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane /  核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm / 核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | ||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.96 Å クライオ電子顕微鏡法 / 解像度: 2.96 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Baker MR / Fan G | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2021 ジャーナル: Commun Biol / 年: 2021タイトル: Cryo-EM structure of type 1 IPR channel in a lipid bilayer. 著者: Mariah R Baker / Guizhen Fan / Alexander B Seryshev / Melina A Agosto / Matthew L Baker / Irina I Serysheva /  要旨: Type 1 inositol 1,4,5-trisphosphate receptor (IPR1) is the predominant Ca-release channel in neurons. IPR1 mediates Ca release from the endoplasmic reticulum into the cytosol and thereby is involved ...Type 1 inositol 1,4,5-trisphosphate receptor (IPR1) is the predominant Ca-release channel in neurons. IPR1 mediates Ca release from the endoplasmic reticulum into the cytosol and thereby is involved in many physiological processes. Here, we present the cryo-EM structures of full-length rat IPR1 reconstituted in lipid nanodisc and detergent solubilized in the presence of phosphatidylcholine determined in ligand-free, closed states by single-particle electron cryo-microscopy. Notably, both structures exhibit the well-established IPR1 protein fold and reveal a nearly complete representation of lipids with similar locations of ordered lipids bound to the transmembrane domains. The lipid-bound structures show improved features that enabled us to unambiguously build atomic models of IPR1 including two membrane associated helices that were not previously resolved in the TM region. Our findings suggest conserved locations of protein-bound lipids among homotetrameric ion channels that are critical for their structural and functional integrity despite the diversity of structural mechanisms for their gating. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23338.map.gz emd_23338.map.gz | 135.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23338-v30.xml emd-23338-v30.xml emd-23338.xml emd-23338.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23338.png emd_23338.png | 308 KB | ||
| Filedesc metadata |  emd-23338.cif.gz emd-23338.cif.gz | 7.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23338 http://ftp.pdbj.org/pub/emdb/structures/EMD-23338 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23338 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23338 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23338.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23338.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of full-length type 1 IP3R in LMNG (apo-state) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein c...
| 全体 | 名称: Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein complex |
|---|---|
| 要素 |
|
-超分子 #1: Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein c...
| 超分子 | 名称: Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) / 器官: cerebellum / Organelle: endoplasmic reticulum / 細胞中の位置: membrane Rattus norvegicus (ドブネズミ) / 器官: cerebellum / Organelle: endoplasmic reticulum / 細胞中の位置: membrane |
| 分子量 | 理論値: 1.3 MDa |
-分子 #1: Inositol 1,4,5-trisphosphate receptor type 1
| 分子 | 名称: Inositol 1,4,5-trisphosphate receptor type 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) / 器官: cerebellum Rattus norvegicus (ドブネズミ) / 器官: cerebellum |
| 分子量 | 理論値: 312.0835 KDa |
| 配列 | 文字列: SSFLHIGDIC SLYAEGSTNG FISTLGLVDD RCVVQPEAGD LNNPPKKFRD CLFKLCPMNR YSAQKQFWKA AKPGANSTTD AVLLNKLHH AADLEKKQNE TENRKLLGTV IQYGNVIQLL HLKSNKYLTV NKRLPALLEK NAMRVTLDEA GNEGSWFYIQ P FYKLRSIG ...文字列: SSFLHIGDIC SLYAEGSTNG FISTLGLVDD RCVVQPEAGD LNNPPKKFRD CLFKLCPMNR YSAQKQFWKA AKPGANSTTD AVLLNKLHH AADLEKKQNE TENRKLLGTV IQYGNVIQLL HLKSNKYLTV NKRLPALLEK NAMRVTLDEA GNEGSWFYIQ P FYKLRSIG DSVVIGDKVV LNPVNAGQPL HASSHQLVDN PGCNEVNSVN CNTSWKIVLF MKWSDNKDDI LKGGDVVRLF HA EQEKFLT CDEHRKKQHV FLRTTGRQSA TSATSSKALW EVEVVQHDPC RGGAGYWNSL FRFKHLATGH YLAAEVDPDF EEE CLEFQP SVDPDQDASR SRLRNAQEKM VYSLVSVPEG NDISSIFELD PTTLRGGDSL VPRNSYVRLR HLCTNTWVHS TNIP IDKEE EKPVMLKIGT SPLKEDKEAF AIVPVSPAEV RDLDFANDAS KVLGSIAGKL EKGTITQNER RSVTKLLEDL VYFVT GGTN SGQDVLEVVF SKPNRERQKL MREQNILKQI FKLLQAPFTD CGDGPMLRLE ELGDQRHAPF RHICRLCYRV LRHSQQ DYR KNQEYIAKQF GFMQKQIGYD VLAEDTITAL LHNNRKLLEK HITAAEIDTF VSLVRKNREP RFLDYLSDLC VSMNKSI PV TQELICKAVL NPTNADILIE TKLVLSRFEF EGVSTGENAL EAGEDEEEVW LFWRDSNKEI RSKSVRELAQ DAKEGQKE D RDVLSYYRYQ LNLFARMCLD RQYLAINEIS GQLDVDLILR CMSDENLPYD LRASFCRLML HMHVDRDPQE QVTPVKYAR LWSEIPSEIA IDDYDSSGAS KDEIKERFAQ TMEFVEEYLR DVVCQRFPFS DKEKNKLTFE VVNLARNLIY FGFYNFSDLL RLTKILLAI LDCVHVTTIF PISKMTKGEE NKGSNVMRSI HGVGELMTQV VLRGGGFLPM TPMAAAPEGN VKQAEPEKED I MVMDTKLK IIEILQFILN VRLDYRISCL LCIFKREFDE SNSQSSETSS GNSSQEGPSN VPGALDFEHI EEQAEGIFGG SE ENTPLDL DDHGGRTFLR VLLHLTMHDY PPLVSGALQL LFRHFSQRQE VLQAFKQVQL LVTSQDVDNY KQIKQDLDQL RSI VEKSEL WVYKGQGPDE PMDGASGENE HKKTEEGTSK PLKHESTSSY NYRVVKEILI RLSKLCVQES ASVRKSRKQQ QRLL RNMGA HAVVLELLQI PYEKAEDTKM QEIMRLAHEF LQNFCAGNQQ NQALLHKHIN LFLNPGILEA VTMQHIFMNN FQLCS EINE RVVQHFVHCI ETHGRNVQYI KFLQTIVKAE GKFIKKCQDM VMAELVNSGE DVLVFYNDRA SFQTLIQMMR SERDRM DEN SPLFMYHIHL VELLAVCTEG KNVYTEIKCN SLLPLDDIVR VVTHEDCIPE VKIAYINFLN HCYVDTEVEM KEIYTSN HM WKLFENFLVD ICRACNNTSD RKHADSVLEK YVTEIVMSIV TTFFSSPFSD QSTTLQTRQP VFVQLLQGVF RVYHCNWL M PSQKASVESC IRVLSDVAKS RAIAIPVDLD SQVNNLFLKS HNIVQKTAMN WRLSARNAAR RDSVLAASRD YRNIIERLQ DIVSALEDRL RPLVQAELSV LVDVLHRPEL LFPENTDARR KCESGGFICK LIKHTKQLLE ENEEKLCIKV LQTLREMMTK DRGYGEKQI SIDELENAEL PQPPEAENST EQELEPSPPL RQLEDHKRGE ALRQILVNRY YGNIRPSGRR ESLTSFGNGP L SPGGPSKP GGGGGGPGSG STSRGEMSLA EVQCHLDKEG ASNLVIDLIM NASSDRVFHE SILLAIALLE GGNTTIQHSF FC RLTEDKK SEKFFKVFYD RMKVAQQEIK ATVTVNTSDL GNKKKDDEVD RDAPSRKKAK EPTTQITEEV RDQLLEASAA TRK AFTTFR READPDDHYQ SGEGTQATTD KAKDDLEMSA VITIMQPILR FLQLLCENHN RDLQNFLRCQ NNKTNYNLVC ETLQ FLDCI CGSTTGGLGL LGLYINEKNV ALINQTLESL TEYCQGPCHE NQNCIATHES NGIDIITALI LNDINPLGKK RMDLV LELK NNASKLLLAI MESRHDSENA ERILYNMRPK ELVEVIKKAY MQGEVEFEDG ENGEDGAASP RNVGHNIYIL AHQLAR HNK ELQTMLKPGG QVDGDEALEF YAKHTAQIEI VRLDRTMEQI VFPVPSICEF LTKESKLRIY YTTERDEQGS KINDFFL RS EDLFNEMNWQ KKLRAQPVLY WCARNMSFWS SISFNLAVLM NLLVAFFYPF KGVRGGTLEP HWSGLLWTAM LISLAIVI A LPKPHGIRAL IASTILRLIF SVGLQPTLFL LGAFNVCNKI IFLMSFVGNC GTFTRGYRAM VLDVEFLYHL LYLLICAMG LFVHEFFYSL LLFDLVYREE TLLNVIKSVT RNGRPIILTA ALALILVYLF SIVGYLFFKD DFILEVDRLP NETAGPETGE SLANDFLYS DVCRVETGEN CTSPAPKEEL LPVEETEQDK EHTCETLLMC IVTVLSHGLR SGGGVGDVLR KPSKEEPLFA A RVIYDLLF FFMVIIIVLN LIFGVIIDTF ADLRSEKQKK EEILKTTCFI CGLERDKFDN KTVTFEEHIK EEHNMWHYLC FI VLVKVKD STEYTGPESY VAEMIRERNL DWFPRMRAMS LVSSDSEGEQ NELRNLQEKL ESTMKLVTNL SGQLSELKDQ MTE QRKQKQ RIGLLGHPPH UniProtKB: Inositol 1,4,5-trisphosphate receptor type 1 |
-分子 #2: (9R,11S)-9-({[(1S)-1-HYDROXYHEXADECYL]OXY}METHYL)-2,2-DIMETHYL-5,...
| 分子 | 名称: (9R,11S)-9-({[(1S)-1-HYDROXYHEXADECYL]OXY}METHYL)-2,2-DIMETHYL-5,7,10-TRIOXA-2LAMBDA~5~-AZA-6LAMBDA~5~-PHOSPHAOCTACOSANE-6,6,11-TRIOL タイプ: ligand / ID: 2 / コピー数: 28 / 式: PLX |
|---|---|
| 分子量 | 理論値: 767.132 Da |
| Chemical component information |  ChemComp-PLX: |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.5 µm / 倍率(補正後): 46943 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3840 pixel / デジタル化 - サイズ - 縦: 3712 pixel / デジタル化 - 画像ごとのフレーム数: 2-35 / 実像数: 19105 / 平均露光時間: 0.2 sec. / 平均電子線量: 42.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 1407714 |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: 詳細: filtered to 60 angstroms |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 3) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3) |
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.96 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3) / 使用した粒子像数: 303481 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.96 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3) / 使用した粒子像数: 303481 |
 ムービー
ムービー コントローラー
コントローラー