+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13054 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of nonameric EPEC SctV-C | |||||||||
 マップデータ マップデータ | Post-processed map_masked | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | T3SS / SctV nonamer / export gate / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   | |||||||||
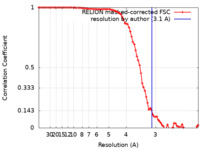
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Yuan B / Wald J / Fahrenkamp D / Marlovits TC | |||||||||
| 資金援助 |  オーストリア, 1件 オーストリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2021 ジャーナル: J Mol Biol / 年: 2021タイトル: Structural Dynamics of the Functional Nonameric Type III Translocase Export Gate. 著者: Biao Yuan / Athina G Portaliou / Rinky Parakra / Jochem H Smit / Jiri Wald / Yichen Li / Bindu Srinivasu / Maria S Loos / Harveer Singh Dhupar / Dirk Fahrenkamp / Charalampos G Kalodimos / ...著者: Biao Yuan / Athina G Portaliou / Rinky Parakra / Jochem H Smit / Jiri Wald / Yichen Li / Bindu Srinivasu / Maria S Loos / Harveer Singh Dhupar / Dirk Fahrenkamp / Charalampos G Kalodimos / Franck Duong van Hoa / Thorben Cordes / Spyridoula Karamanou / Thomas C Marlovits / Anastassios Economou /      要旨: Type III protein secretion is widespread in Gram-negative pathogens. It comprises the injectisome with a surface-exposed needle and an inner membrane translocase. The translocase contains the SctRSTU ...Type III protein secretion is widespread in Gram-negative pathogens. It comprises the injectisome with a surface-exposed needle and an inner membrane translocase. The translocase contains the SctRSTU export channel enveloped by the export gate subunit SctV that binds chaperone/exported clients and forms a putative ante-chamber. We probed the assembly, function, structure and dynamics of SctV from enteropathogenic E. coli (EPEC). In both EPEC and E. coli lab strains, SctV forms peripheral oligomeric clusters that are detergent-extracted as homo-nonamers. Membrane-embedded SctV is necessary and sufficient to act as a receptor for different chaperone/exported protein pairs with distinct C-domain binding sites that are essential for secretion. Negative staining electron microscopy revealed that peptidisc-reconstituted His-SctV forms a tripartite particle of ∼22 nm with a N-terminal domain connected by a short linker to a C-domain ring structure with a ∼5 nm-wide inner opening. The isolated C-domain ring was resolved with cryo-EM at 3.1 Å and structurally compared to other SctV homologues. Its four sub-domains undergo a three-stage "pinching" motion. Hydrogen-deuterium exchange mass spectrometry revealed this to involve dynamic and rigid hinges and a hyper-flexible sub-domain that flips out of the ring periphery and binds chaperones on and between adjacent protomers. These motions are coincident with local conformational changes at the pore surface and ring entry mouth that may also be modulated by the ATPase inner stalk. We propose that the intrinsic dynamics of the SctV protomer are modulated by chaperones and the ATPase and could affect allosterically the other subunits of the nonameric ring during secretion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13054.map.gz emd_13054.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13054-v30.xml emd-13054-v30.xml emd-13054.xml emd-13054.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13054_fsc.xml emd_13054_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13054.png emd_13054.png | 139.8 KB | ||
| マスクデータ |  emd_13054_msk_1.map emd_13054_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13054.cif.gz emd-13054.cif.gz | 5.7 KB | ||
| その他 |  emd_13054_half_map_1.map.gz emd_13054_half_map_1.map.gz emd_13054_half_map_2.map.gz emd_13054_half_map_2.map.gz | 40.7 MB 40.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13054 http://ftp.pdbj.org/pub/emdb/structures/EMD-13054 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13054 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13054 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13054_validation.pdf.gz emd_13054_validation.pdf.gz | 740.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13054_full_validation.pdf.gz emd_13054_full_validation.pdf.gz | 740 KB | 表示 | |
| XML形式データ |  emd_13054_validation.xml.gz emd_13054_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_13054_validation.cif.gz emd_13054_validation.cif.gz | 20 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13054 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13054 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13054 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13054 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13054.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13054.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map_masked | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13054_msk_1.map emd_13054_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
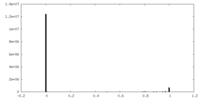
| 密度ヒストグラム |
-ハーフマップ: half-map1 from auto-refinement
| ファイル | emd_13054_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map1 from auto-refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
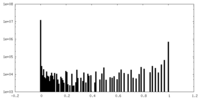
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 from autorefinement
| ファイル | emd_13054_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 from autorefinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
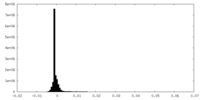
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EPEC SctV-C nonamer
| 全体 | 名称: EPEC SctV-C nonamer |
|---|---|
| 要素 |
|
-超分子 #1: EPEC SctV-C nonamer
| 超分子 | 名称: EPEC SctV-C nonamer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 379.2 kDa/nm |
-分子 #1: Translocator EscV
| 分子 | 名称: Translocator EscV / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: E2348/69 / EPEC |
| 分子量 | 理論値: 37.952457 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NISPGAEPLI LNLSSNIYSS DITQQIEVMR WNFFEESGIP LPKIIVNPVK NNDSAIEFLL YQESIYKDTL IDDTVYFEAG HAEISFEFV QEKLSTNSIV YKTNKTNQQL AHLTGMDVYA TTNDKITFLL KKLVLSNAKE FIGVQETRYL MDIMERKYNE L VKELQRQL ...文字列: NISPGAEPLI LNLSSNIYSS DITQQIEVMR WNFFEESGIP LPKIIVNPVK NNDSAIEFLL YQESIYKDTL IDDTVYFEAG HAEISFEFV QEKLSTNSIV YKTNKTNQQL AHLTGMDVYA TTNDKITFLL KKLVLSNAKE FIGVQETRYL MDIMERKYNE L VKELQRQL GLSKIVDILQ RLVEENVSIR DLRTIFETLI FWSTKEKDVV ILCEYVRIAL RRHILGRYSV SGTLLNVWLI GS DIENELR ESIRQTSSGS YLNISPERTE QIIGFLKNIM NPTGNGVILT ALDIRRYVKK MIEGSFPSVP VLSFQEVGNN IEL KVLGTV NDFRA UniProtKB: Translocator EscV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | The sample was mono-disperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 3739 / 平均電子線量: 41.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X