+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10124 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of ZEBOV GP in complex with 5T0180 antibody | |||||||||
 マップデータ マップデータ | Locally Filtered - Z flipped | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endoplasmic reticulum / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / membrane raft / fusion of virus membrane with host endosome membrane / viral envelope ...host cell endoplasmic reticulum / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / membrane raft / fusion of virus membrane with host endosome membrane / viral envelope / lipid binding / host cell plasma membrane / virion membrane / extracellular region / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
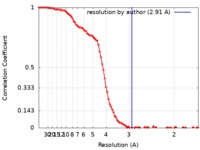
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.91 Å | |||||||||
 データ登録者 データ登録者 | Diskin R / Cohen-Dvashi H | |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2020 ジャーナル: Cell Host Microbe / 年: 2020タイトル: Structural Basis for a Convergent Immune Response against Ebola Virus. 著者: Hadas Cohen-Dvashi / Matthias Zehner / Stefanie Ehrhardt / Michael Katz / Nadav Elad / Florian Klein / Ron Diskin /   要旨: Ebola virus disease is a severe health problem in Africa. Vaccines that display the Zaire ebolavirus glycoprotein spike complex are a prime component for the effort to combat it. The V3-15/V1-40- ...Ebola virus disease is a severe health problem in Africa. Vaccines that display the Zaire ebolavirus glycoprotein spike complex are a prime component for the effort to combat it. The V3-15/V1-40-based class of antibodies was recently discovered to be a common response in individuals who received the Ebola virus vaccines. These antibodies display attractive properties, and thus likely contribute to the efficacy of the vaccines. Here, we use cryo-EM to elucidate how three V3-15/V1-40 antibodies from different individuals target the virus and found a convergent mechanism against a partially conserved site on the spike complex. Our study rationalizes the selection of the V3-15/V1-40 germline genes for specifically targeting this site and highlights Ebolavirus species-specific sequence divergences that may restrict breadth of V3-15/V1-40-based humoral response. The results from this study could help develop improved immunization schemes and further enable the design of immunogens that would be efficacious against a broader set of Ebolavirus species. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10124.map.gz emd_10124.map.gz | 4.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10124-v30.xml emd-10124-v30.xml emd-10124.xml emd-10124.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10124_fsc.xml emd_10124_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10124.png emd_10124.png | 149.1 KB | ||
| その他 |  emd_10124_half_map_1.map.gz emd_10124_half_map_1.map.gz emd_10124_half_map_2.map.gz emd_10124_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10124 http://ftp.pdbj.org/pub/emdb/structures/EMD-10124 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10124 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10124 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10124_validation.pdf.gz emd_10124_validation.pdf.gz | 433.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10124_full_validation.pdf.gz emd_10124_full_validation.pdf.gz | 432.2 KB | 表示 | |
| XML形式データ |  emd_10124_validation.xml.gz emd_10124_validation.xml.gz | 15.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10124 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10124 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10124 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10124 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10124.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10124.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally Filtered - Z flipped | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.849 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map B
| ファイル | emd_10124_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
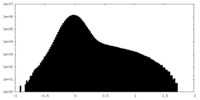
| 投影像・断面図 |
| ||||||||||||
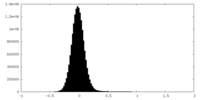
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_10124_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
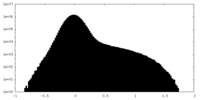
| 投影像・断面図 |
| ||||||||||||
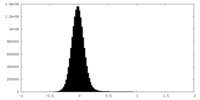
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EBOV-GP in complex with 3T0265 antibody
| 全体 | 名称: EBOV-GP in complex with 3T0265 antibody |
|---|---|
| 要素 |
|
-超分子 #1: EBOV-GP in complex with 3T0265 antibody
| 超分子 | 名称: EBOV-GP in complex with 3T0265 antibody / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: EBOV-GP
| 超分子 | 名称: EBOV-GP / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-超分子 #3: 5T0180 antibody
| 超分子 | 名称: 5T0180 antibody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1-#2 / 詳細: A fab portion generated by cleavage |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-分子 #1: Light Chain
| 分子 | 名称: Light Chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.130488 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSVLTQPPSV SGAPGQRVTI SCTGSSSNIG AGYDVQWYQQ LPGTAPKVLI YGNNNRPSGV PDRFSGSKSG SSASLAITGL QAEDEADYY CQTYDSRLRD QWVFGGGTKL TVLGQPKAAP SVTLFPPSSE ELQANKATLV CLISDFYPGA VTVAWKADSS P VKAGVETT ...文字列: QSVLTQPPSV SGAPGQRVTI SCTGSSSNIG AGYDVQWYQQ LPGTAPKVLI YGNNNRPSGV PDRFSGSKSG SSASLAITGL QAEDEADYY CQTYDSRLRD QWVFGGGTKL TVLGQPKAAP SVTLFPPSSE ELQANKATLV CLISDFYPGA VTVAWKADSS P VKAGVETT TPSKQSNNKY AASSYLSLTP EQWKSHRSYS CQVTHEGSTV EKTVAPTECS |
-分子 #2: Heavy Chain
| 分子 | 名称: Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.636709 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVESGGG LVKPGGSLRL SCAASGFSFG NAWMNWVRQA PGKGLEWVGR IKSKSDGGTT DYAAPVKDRF IISRDDSKKT LYLQMNSLR REDTAVYYCV RGPFYCDTCG PNDYWGQGTL VTVSSGSTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE P VTVSWNSG ...文字列: EVQLVESGGG LVKPGGSLRL SCAASGFSFG NAWMNWVRQA PGKGLEWVGR IKSKSDGGTT DYAAPVKDRF IISRDDSKKT LYLQMNSLR REDTAVYYCV RGPFYCDTCG PNDYWGQGTL VTVSSGSTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE P VTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYICN VNHKPSNTKV DKRVEPKSCD KTH |
-分子 #3: Envelope Glycoprotein 1
| 分子 | 名称: Envelope Glycoprotein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 35.706977 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ETGRSIPLGV IHNSALQVSD VDKLVCRDKL SSTNQLRSVG LNLEGNGVAT DVPSATKRWG FRSGVPPKVV NYEAGEWAEN CYNLEIKKP DGSECLPAAP DGIRGFPRCR YVHKVSGTGP CAGDFAFHKE GAFFLYDRLA STVIYRGTTF AEGVVAFLIL P QAKKDFFS ...文字列: ETGRSIPLGV IHNSALQVSD VDKLVCRDKL SSTNQLRSVG LNLEGNGVAT DVPSATKRWG FRSGVPPKVV NYEAGEWAEN CYNLEIKKP DGSECLPAAP DGIRGFPRCR YVHKVSGTGP CAGDFAFHKE GAFFLYDRLA STVIYRGTTF AEGVVAFLIL P QAKKDFFS SHPLREPVNA TEDPSSGYYS TTIRYQATGF GTNETEYLFE VDNLTYVQLE SRFTPQFLLQ LNETIYTSGK RS NTTGKLI WKVNPEIDTT IGEWAFWETK KNLTRKIRSE ELSFTVVSTH HQDTGEESAS SGKLGLITNT IAGVAGLITG GRR TRR |
-分子 #4: Envelope glycoprotein
| 分子 | 名称: Envelope glycoprotein / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 18.989391 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EAIVNAQPKC NPNLHYWTTQ DEGAAIGLAW IPYFGPAAEG IYIEGLMHNQ DGLICGLRQL ANETTQALQL FLRATTELRT FSILNRKAI DFLLQRWGGT CHILGPDCCI EPHDWTKNIT DKIDQIIHDF VDGSGYIPEA PRDGQAYVRK DGEWVLLSTF L GTHHHHHH |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 163 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 27.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X