+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21951 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of Human Apoferritin Light Chain Vitrified Using Back-it-up | ||||||||||||
 マップデータ マップデータ | Sharpened map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Human / apoferritin / ferritin / light chain / back-it-up / through-grid wicking / METAL BINDING PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / autolysosome / Scavenging by Class A Receptors / Golgi Associated Vesicle Biogenesis / intracellular sequestering of iron ion / ferric iron binding / Iron uptake and transport / ferrous iron binding / azurophil granule lumen / iron ion transport ...: / autolysosome / Scavenging by Class A Receptors / Golgi Associated Vesicle Biogenesis / intracellular sequestering of iron ion / ferric iron binding / Iron uptake and transport / ferrous iron binding / azurophil granule lumen / iron ion transport / intracellular iron ion homeostasis / iron ion binding / Neutrophil degranulation / extracellular exosome / extracellular region / identical protein binding / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.0 Å | ||||||||||||
 データ登録者 データ登録者 | Tan YZ / Rubinstein JL | ||||||||||||
| 資金援助 |  カナダ, 3件 カナダ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2020 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2020タイトル: Through-grid wicking enables high-speed cryoEM specimen preparation. 著者: Yong Zi Tan / John L Rubinstein /  要旨: Blotting times for conventional cryoEM specimen preparation complicate time-resolved studies and lead to some specimens adopting preferred orientations or denaturing at the air-water interface. Here, ...Blotting times for conventional cryoEM specimen preparation complicate time-resolved studies and lead to some specimens adopting preferred orientations or denaturing at the air-water interface. Here, it is shown that solution sprayed onto one side of a holey cryoEM grid can be wicked through the grid by a glass-fiber filter held against the opposite side, often called the `back', of the grid, producing a film suitable for vitrification. This process can be completed in tens of milliseconds. Ultrasonic specimen application and through-grid wicking were combined in a high-speed specimen-preparation device that was named `Back-it-up' or BIU. The high liquid-absorption capacity of the glass fiber compared with self-wicking grids makes the method relatively insensitive to the amount of sample applied. Consequently, through-grid wicking produces large areas of ice that are suitable for cryoEM for both soluble and detergent-solubilized protein complexes. The speed of the device increases the number of views for a specimen that suffers from preferred orientations. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21951.map.gz emd_21951.map.gz | 85.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21951-v30.xml emd-21951-v30.xml emd-21951.xml emd-21951.xml | 28 KB 28 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21951_fsc.xml emd_21951_fsc.xml | 11 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21951.png emd_21951.png | 198.6 KB | ||
| マスクデータ |  emd_21951_msk_1.map emd_21951_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21951.cif.gz emd-21951.cif.gz | 6.3 KB | ||
| その他 |  emd_21951_additional_1.map.gz emd_21951_additional_1.map.gz emd_21951_additional_2.map.gz emd_21951_additional_2.map.gz emd_21951_additional_3.map.gz emd_21951_additional_3.map.gz emd_21951_additional_4.map.gz emd_21951_additional_4.map.gz emd_21951_additional_5.map.gz emd_21951_additional_5.map.gz emd_21951_half_map_1.map.gz emd_21951_half_map_1.map.gz emd_21951_half_map_2.map.gz emd_21951_half_map_2.map.gz | 43.9 MB 7.9 MB 194.9 KB 20.5 MB 23.7 MB 84.1 MB 84.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21951 http://ftp.pdbj.org/pub/emdb/structures/EMD-21951 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21951 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21951 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21951_validation.pdf.gz emd_21951_validation.pdf.gz | 990.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21951_full_validation.pdf.gz emd_21951_full_validation.pdf.gz | 990.2 KB | 表示 | |
| XML形式データ |  emd_21951_validation.xml.gz emd_21951_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_21951_validation.cif.gz emd_21951_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21951 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21951 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21951 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21951 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6wx6MC  6wxbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10421 (タイトル: Single-Particle CryoEM of Human Apoferritin Light Chain Vitrified Using Back-it-up EMPIAR-10421 (タイトル: Single-Particle CryoEM of Human Apoferritin Light Chain Vitrified Using Back-it-upData size: 2.1 TB Data #1: Unaligned Falcon IV movie frames [micrographs - multiframe] Data #2: Aligned Dose-Weighted Micrographs [micrographs - single frame] Data #3: Final Particle Stack with Final Euler Angles and Shifts [picked particles - multiframe - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21951.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21951.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.816 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21951_msk_1.map emd_21951_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
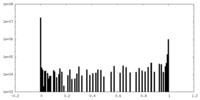
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Raw map
| ファイル | emd_21951_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map | ||||||||||||
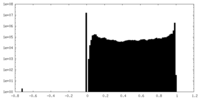
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local resolution map
| ファイル | emd_21951_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 3DFSC - Thresholded and Binarized
| ファイル | emd_21951_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3DFSC - Thresholded and Binarized | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 3DFSC - Thresholded
| ファイル | emd_21951_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3DFSC - Thresholded | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 3DFSC - Raw
| ファイル | emd_21951_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3DFSC - Raw | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_21951_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_21951_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human apoferritin light chain
| 全体 | 名称: Human apoferritin light chain |
|---|---|
| 要素 |
|
-超分子 #1: Human apoferritin light chain
| 超分子 | 名称: Human apoferritin light chain / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 580 KDa |
-分子 #1: Ferritin light chain
| 分子 | 名称: Ferritin light chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.275898 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TGWSHPQFEK LKGGSSRGGG GGSGGSGGSG GSMSSQIRQN YSTDVEAAVN SLVNLYLQAS YTYLSLGFYF DRDDVALEGV SHFFRELAE EKREGYERLL KMQNQRGGRA LFQDIKKPAE DEWGKTPDAM KAAMALEKKL NQALLDLHAL GSARTDPHLC D FLETHFLD ...文字列: TGWSHPQFEK LKGGSSRGGG GGSGGSGGSG GSMSSQIRQN YSTDVEAAVN SLVNLYLQAS YTYLSLGFYF DRDDVALEGV SHFFRELAE EKREGYERLL KMQNQRGGRA LFQDIKKPAE DEWGKTPDAM KAAMALEKKL NQALLDLHAL GSARTDPHLC D FLETHFLD EEVKLIKKMG DHLTNLHRLG GPEAGLGEYL FERLTLRHDG GSGGSGGSGG SGGGASGGS UniProtKB: Ferritin light chain |
-分子 #2: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 2 / コピー数: 8 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 2420 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Homemade / 材質: COPPER/RHODIUM / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR 詳細: Both sides of the grid were glow discharged for 120 seconds. |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 50 % / チャンバー内温度: 298 K / 装置: HOMEMADE PLUNGER 詳細: Back-it-up (ultrasonic specimen application and through-grid wicking in a high-speed specimen preparation device) was used. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 2 / 実像数: 3168 / 平均露光時間: 9.0 sec. / 平均電子線量: 27.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





























 Z
Z Y
Y X
X