+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12875 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The archaellum of Methanocaldococcus villosus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Archaellum / archaellin / Methanocaldococcus villosus / cryoEM / helical reconstruction / PROTEIN FIBRIL | |||||||||
| 生物種 |  Methanocaldococcus villosus (メタン生成菌) Methanocaldococcus villosus (メタン生成菌) | |||||||||
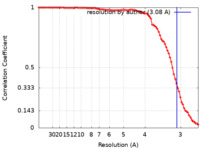
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||
 データ登録者 データ登録者 | Isupov M / Gambelli L | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: An archaellum filament composed of two alternating subunits. 著者: Lavinia Gambelli / Michail N Isupov / Rebecca Conners / Mathew McLaren / Annett Bellack / Vicki Gold / Reinhard Rachel / Bertram Daum /   要旨: Archaea use a molecular machine, called the archaellum, to swim. The archaellum consists of an ATP-powered intracellular motor that drives the rotation of an extracellular filament composed of ...Archaea use a molecular machine, called the archaellum, to swim. The archaellum consists of an ATP-powered intracellular motor that drives the rotation of an extracellular filament composed of multiple copies of proteins named archaellins. In many species, several archaellin homologs are encoded in the same operon; however, previous structural studies indicated that archaellum filaments mainly consist of only one protein species. Here, we use electron cryo-microscopy to elucidate the structure of the archaellum from Methanocaldococcus villosus at 3.08 Å resolution. The filament is composed of two alternating archaellins, suggesting that the architecture and assembly of archaella is more complex than previously thought. Moreover, we identify structural elements that may contribute to the filament's flexibility. #1:  ジャーナル: Faraday Disc.Chem.Soc / 年: 2022 ジャーナル: Faraday Disc.Chem.Soc / 年: 2022タイトル: Escaping the symmetry trap in helical reconstruction 著者: Gambelli L / Isupov MN / Daum B | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12875.map.gz emd_12875.map.gz | 57.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12875-v30.xml emd-12875-v30.xml emd-12875.xml emd-12875.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12875_fsc.xml emd_12875_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12875.png emd_12875.png | 163.6 KB | ||
| Filedesc metadata |  emd-12875.cif.gz emd-12875.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12875 http://ftp.pdbj.org/pub/emdb/structures/EMD-12875 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12875 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12875 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12875_validation.pdf.gz emd_12875_validation.pdf.gz | 502.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12875_full_validation.pdf.gz emd_12875_full_validation.pdf.gz | 502.4 KB | 表示 | |
| XML形式データ |  emd_12875_validation.xml.gz emd_12875_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_12875_validation.cif.gz emd_12875_validation.cif.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12875 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12875 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12875 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12875 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ofqMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10884 (タイトル: New insights into the architecture and dynamics of archaella EMPIAR-10884 (タイトル: New insights into the architecture and dynamics of archaellaData size: 3.3 TB Data #1: Unaligned 39-frames movies of Methanocaldococcus villosus archaella [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12875.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12875.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.39 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Archaellum
| 全体 | 名称: Archaellum |
|---|---|
| 要素 |
|
-超分子 #1: Archaellum
| 超分子 | 名称: Archaellum / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Methanocaldococcus villosus (メタン生成菌) Methanocaldococcus villosus (メタン生成菌) |
-分子 #1: Archaellin
| 分子 | 名称: Archaellin / タイプ: protein_or_peptide / ID: 1 / コピー数: 23 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Methanocaldococcus villosus (メタン生成菌) Methanocaldococcus villosus (メタン生成菌) |
| 分子量 | 理論値: 22.084279 KDa |
| 組換発現 | 生物種:  Methanocaldococcus villosus (メタン生成菌) Methanocaldococcus villosus (メタン生成菌) |
| 配列 | 文字列: AIGIGTLIIF IAMVLVAAVA AAVLINTSGF LQQKAMATGK ESTEQVASGL LCSGVTGHYV KNKGIDRIVI YITPNAGSAP IDLKQCKLF LMYDGKAVSL NFSKYDTNTV GDFTNGIKDI FNTTVVKWNN ADATSFVVVA LQDDDKSLLT NAVINKGDLA G VLVNVSAA ...文字列: AIGIGTLIIF IAMVLVAAVA AAVLINTSGF LQQKAMATGK ESTEQVASGL LCSGVTGHYV KNKGIDRIVI YITPNAGSAP IDLKQCKLF LMYDGKAVSL NFSKYDTNTV GDFTNGIKDI FNTTVVKWNN ADATSFVVVA LQDDDKSLLT NAVINKGDLA G VLVNVSAA FGKHVGTRER VSGYLQPEFG APAVIEFTTP AAFTSDVIEL Q |
-分子 #2: Archaellin
| 分子 | 名称: Archaellin / タイプ: protein_or_peptide / ID: 2 / コピー数: 22 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Methanocaldococcus villosus (メタン生成菌) Methanocaldococcus villosus (メタン生成菌) |
| 分子量 | 理論値: 22.273643 KDa |
| 組換発現 | 生物種:  Methanocaldococcus villosus (メタン生成菌) Methanocaldococcus villosus (メタン生成菌) |
| 配列 | 文字列: AIGIGTLIIF IAMVLVAAVA AAVLINTSGF LQQKAMATGK ESTEQVASGL QVIRVLGNHS GGKINWLAVL ISPNAGSAPI DLSQATVMI TDGTHKVIAK YNSTFFNGTL KNGGSIFEAK YNNTTALKPL FDDLPATAFG IVVLQDADTS CSKDTPVINK G DIVAICLN ...文字列: AIGIGTLIIF IAMVLVAAVA AAVLINTSGF LQQKAMATGK ESTEQVASGL QVIRVLGNHS GGKINWLAVL ISPNAGSAPI DLSQATVMI TDGTHKVIAK YNSTFFNGTL KNGGSIFEAK YNNTTALKPL FDDLPATAFG IVVLQDADTS CSKDTPVINK G DIVAICLN VSNTLNLKPR TKVTGAVIPE FGAPAVISFT TPATYLDTQH IIELQ |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 45 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #4: UNKNOWN LIGAND
| 分子 | 名称: UNKNOWN LIGAND / タイプ: ligand / ID: 4 / コピー数: 804 / 式: UNL |
|---|---|
| Chemical component information | 
ChemComp-UNL: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 構成要素 - 濃度: 5.0 mM / 構成要素 - 式: C8H18N2O4S / 構成要素 - 名称: HEPES |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 294 K / 装置: FEI VITROBOT MARK III / 詳細: blotting time of 4 sec, -1 blot force. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2795 / 平均露光時間: 1.0 sec. / 平均電子線量: 37.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: SPOT SCAN / 撮影モード: OTHER / Cs: 2.7 mm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)