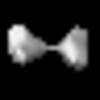+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8250 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram-averaged map of a single rubella virus capsid unit | |||||||||
 マップデータ マップデータ | Subtomogram-averaged map of single capsid unit | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | rubella virus capsid protein / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=4 icosahedral viral capsid / host cell Golgi membrane / host cell mitochondrion / viral nucleocapsid / clathrin-dependent endocytosis of virus by host cell / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / virion membrane / RNA binding ...T=4 icosahedral viral capsid / host cell Golgi membrane / host cell mitochondrion / viral nucleocapsid / clathrin-dependent endocytosis of virus by host cell / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / virion membrane / RNA binding / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rubella virus strain M33 (ウイルス) / Rubella virus strain M33 (ウイルス) /  Rubella virus (風疹ウイルス) Rubella virus (風疹ウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 35.0 Å | |||||||||
 データ登録者 データ登録者 | Mangala Prasad V / Klose T | |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2017 ジャーナル: PLoS Pathog / 年: 2017タイトル: Assembly, maturation and three-dimensional helical structure of the teratogenic rubella virus. 著者: Vidya Mangala Prasad / Thomas Klose / Michael G Rossmann /  要旨: Viral infections during pregnancy are a significant cause of infant morbidity and mortality. Of these, rubella virus infection is a well-substantiated example that leads to miscarriages or severe ...Viral infections during pregnancy are a significant cause of infant morbidity and mortality. Of these, rubella virus infection is a well-substantiated example that leads to miscarriages or severe fetal defects. However, structural information about the rubella virus has been lacking due to the pleomorphic nature of the virions. Here we report a helical structure of rubella virions using cryo-electron tomography. Sub-tomogram averaging of the surface spikes established the relative positions of the viral glycoproteins, which differed from the earlier icosahedral models of the virus. Tomographic analyses of in vitro assembled nucleocapsids and virions provide a template for viral assembly. Comparisons of immature and mature virions show large rearrangements in the glycoproteins that may be essential for forming the infectious virions. These results present the first known example of a helical membrane-enveloped virus, while also providing a structural basis for its assembly and maturation pathway. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8250.map.gz emd_8250.map.gz | 14.4 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8250-v30.xml emd-8250-v30.xml emd-8250.xml emd-8250.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8250.png emd_8250.png | 60.2 KB | ||
| Filedesc metadata |  emd-8250.cif.gz emd-8250.cif.gz | 5.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8250 http://ftp.pdbj.org/pub/emdb/structures/EMD-8250 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8250 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8250 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8250.map.gz / 形式: CCP4 / 大きさ: 54.7 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8250.map.gz / 形式: CCP4 / 大きさ: 54.7 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram-averaged map of single capsid unit | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.28 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rubella virus strain M33
| 全体 | 名称:  Rubella virus strain M33 (ウイルス) Rubella virus strain M33 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Rubella virus strain M33
| 超分子 | 名称: Rubella virus strain M33 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Rubella virus strain M33 (ウイルス) Rubella virus strain M33 (ウイルス) |
| 分子量 | 理論値: 64 KDa |
-分子 #1: capsid protein
| 分子 | 名称: capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rubella virus (風疹ウイルス) Rubella virus (風疹ウイルス) |
| 分子量 | 理論値: 30.016332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEDLQKALEA QSRALRAELA AGASQSRRPR PPRQRDSSTS GDDSGRDSGG PRRRRGNRGR GQRRDWSRAP PPPEERQETR SQTPAPKPS RAPPQQPQPP RMQTGRGGSA PRPELGPPTN PFQAAVARGL RPPLHDPDTE APTEACVTSW LWSEGEGAVF Y RVDLHFTN ...文字列: MEDLQKALEA QSRALRAELA AGASQSRRPR PPRQRDSSTS GDDSGRDSGG PRRRRGNRGR GQRRDWSRAP PPPEERQETR SQTPAPKPS RAPPQQPQPP RMQTGRGGSA PRPELGPPTN PFQAAVARGL RPPLHDPDTE APTEACVTSW LWSEGEGAVF Y RVDLHFTN LGTPPLDEDG RWDPALMYNP CGPEPPAHVV RAYNQPAGDV RGVWGKGERT YAEQDFRVGG TRWHRLLRMP VR GLDGDSA PLPPHTTERI ETRSARHPWR IR UniProtKB: Structural polyprotein |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 35 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON | |||||||||
| 凍結 | 凍結剤: ETHANE / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 11000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 35.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: PEET (ver. 1.11.0) / 使用したサブトモグラム数: 18 |
|---|---|
| 抽出 | トモグラム数: 15 / 使用した粒子像数: 20 / 参照モデル: none / 手法: manual picking / ソフトウェア - 名称: IMOD/PEET (ver. 4.8.40/1.11.0) |
| 最終 角度割当 | タイプ: OTHER |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: sumf |
|---|---|
| 得られたモデル |  PDB-5khf: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)