+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7khr | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of bafilomycin A1-bound intact V-ATPase from bovine brain | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PROTON TRANSPORT / bafilomycin A1 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ROS and RNS production in phagocytes / Insulin receptor recycling / Transferrin endocytosis and recycling / Amino acids regulate mTORC1 / Ion channel transport / Metabolism of Angiotensinogen to Angiotensins / pH reduction / RHOA GTPase cycle / plasma membrane proton-transporting V-type ATPase complex / cellular response to increased oxygen levels ...ROS and RNS production in phagocytes / Insulin receptor recycling / Transferrin endocytosis and recycling / Amino acids regulate mTORC1 / Ion channel transport / Metabolism of Angiotensinogen to Angiotensins / pH reduction / RHOA GTPase cycle / plasma membrane proton-transporting V-type ATPase complex / cellular response to increased oxygen levels / synaptic vesicle lumen acidification / lysosomal lumen acidification / clathrin-coated vesicle membrane / endosomal lumen acidification / vacuolar proton-transporting V-type ATPase, V0 domain / vacuolar proton-transporting V-type ATPase, V1 domain / vacuolar transport / proton-transporting V-type ATPase complex / cell projection organization / vacuolar proton-transporting V-type ATPase complex / vacuolar acidification / regulation of cellular pH / dendritic spine membrane / Neutrophil degranulation / ATPase activator activity / autophagosome membrane / cilium assembly / positive regulation of Wnt signaling pathway / regulation of macroautophagy / endomembrane system / H+-transporting two-sector ATPase / ATP metabolic process / transport vesicle / RNA endonuclease activity / endoplasmic reticulum-Golgi intermediate compartment membrane / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / proton transmembrane transport / receptor-mediated endocytosis / cilium / transmembrane transport / synaptic vesicle membrane / positive regulation of canonical Wnt signaling pathway / melanosome / signaling receptor activity / ATPase binding / postsynaptic membrane / intracellular iron ion homeostasis / receptor-mediated endocytosis of virus by host cell / 加水分解酵素; エステル加水分解酵素 / early endosome / lysosome / endosome membrane / endosome / nuclear speck / apical plasma membrane / lysosomal membrane / external side of plasma membrane / axon / endoplasmic reticulum membrane / perinuclear region of cytoplasm / Golgi apparatus / ATP hydrolysis activity / ATP binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.62 Å | ||||||
 データ登録者 データ登録者 | Wang, R. / Li, X. | ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Molecular basis of V-ATPase inhibition by bafilomycin A1. 著者: Rong Wang / Jin Wang / Abdirahman Hassan / Chia-Hsueh Lee / Xiao-Song Xie / Xiaochun Li /  要旨: Pharmacological inhibition of vacuolar-type H-ATPase (V-ATPase) by its specific inhibitor can abrogate tumor metastasis, prevent autophagy, and reduce cellular signaling responses. Bafilomycin A1, a ...Pharmacological inhibition of vacuolar-type H-ATPase (V-ATPase) by its specific inhibitor can abrogate tumor metastasis, prevent autophagy, and reduce cellular signaling responses. Bafilomycin A1, a member of macrolide antibiotics and an autophagy inhibitor, serves as a specific and potent V-ATPases inhibitor. Although there are many V-ATPase structures reported, the molecular basis of specific inhibitors on V-ATPase remains unknown. Here, we report the cryo-EM structure of bafilomycin A1 bound intact bovine V-ATPase at an overall resolution of 3.6-Å. The structure reveals six bafilomycin A1 molecules bound to the c-ring. One bafilomycin A1 molecule engages with two c subunits and disrupts the interactions between the c-ring and subunit a, thereby preventing proton translocation. Structural and sequence analyses demonstrate that the bafilomycin A1-binding residues are conserved in yeast and mammalian species and the 7'-hydroxyl group of bafilomycin A1 acts as a unique feature recognized by subunit c. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7khr.cif.gz 7khr.cif.gz | 1.3 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7khr.ent.gz pdb7khr.ent.gz | 1.1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7khr.json.gz 7khr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7khr_validation.pdf.gz 7khr_validation.pdf.gz | 1.9 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7khr_full_validation.pdf.gz 7khr_full_validation.pdf.gz | 2 MB | 表示 | |
| XML形式データ |  7khr_validation.xml.gz 7khr_validation.xml.gz | 203.3 KB | 表示 | |
| CIF形式データ |  7khr_validation.cif.gz 7khr_validation.cif.gz | 311.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kh/7khr https://data.pdbj.org/pub/pdb/validation_reports/kh/7khr ftp://data.pdbj.org/pub/pdb/validation_reports/kh/7khr ftp://data.pdbj.org/pub/pdb/validation_reports/kh/7khr | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-V-type proton ATPase ... , 14種, 30分子 ABCDEFGHIJKLMNOPabdescgklmnopq
| #1: タンパク質 | 分子量: 68420.914 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  #2: タンパク質 | 分子量: 56637.555 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  #3: タンパク質 | | 分子量: 44042.566 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #4: タンパク質 | | 分子量: 28297.893 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #5: タンパク質 | 分子量: 26178.371 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  #6: タンパク質 | | 分子量: 13417.275 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #7: タンパク質 | 分子量: 13588.344 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  #8: タンパク質 | | 分子量: 54155.875 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #9: タンパク質 | | 分子量: 96431.398 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #10: タンパク質 | | 分子量: 21530.426 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #11: タンパク質 | | 分子量: 40369.949 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #12: タンパク質 | | 分子量: 9188.992 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #13: タンパク質 | | 分子量: 51818.754 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #15: タンパク質 | 分子量: 15727.726 Da / 分子数: 9 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-タンパク質 , 2種, 2分子 rf
| #14: タンパク質 | 分子量: 39529.266 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #16: タンパク質 | 分子量: 11030.029 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  参照: UniProt: Q3ZC23, 加水分解酵素; エステル加水分解酵素 |
-糖 , 3種, 9分子 
| #17: 多糖 | alpha-D-glucopyranose-(1-2)-alpha-D-glucopyranose-(1-3)-alpha-D-glucopyranose-(1-3)-alpha-D- ...alpha-D-glucopyranose-(1-2)-alpha-D-glucopyranose-(1-3)-alpha-D-glucopyranose-(1-3)-alpha-D-mannopyranose-(1-2)-alpha-D-mannopyranose-(1-2)-alpha-D-mannopyranose-(1-3)-beta-D-mannopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |
|---|---|
| #18: 多糖 | alpha-D-mannopyranose-(1-6)-alpha-D-mannopyranose |
| #23: 糖 | ChemComp-NAG / |
-非ポリマー , 6種, 18分子 

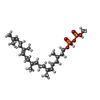


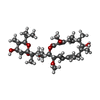





| #19: 化合物 | ChemComp-MG / | ||||
|---|---|---|---|---|---|
| #20: 化合物 | ChemComp-ADP / | ||||
| #21: 化合物 | ChemComp-WJP / | ||||
| #22: 化合物 | ChemComp-POV / ( #24: 化合物 | ChemComp-OLA / | #25: 化合物 | ChemComp-WEV / ( |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Cryo-EM structure of bafilomycin A1-bound intact V-ATPase from bovine brain タイプ: COMPLEX / Entity ID: #1-#16 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 60 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING ONLY |
|---|---|
| 3次元再構成 | 解像度: 3.62 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 26530 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj













