| 登録情報 | データベース: PDB / ID: 5zol
|
|---|
| タイトル | Crystal structure of a three sites mutantion of FSAA complexed with HA and product |
|---|
 要素 要素 | Fructose-6-phosphate aldolase 1 |
|---|
 キーワード キーワード | LYASE / Fructose-6-phosphate aldolase / donor / acceptor / mutant / flexible |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
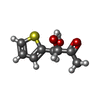
ketone catabolic process / fructose 6-phosphate aldolase activity / 付加脱離酵素(リアーゼ); C-Cリアーゼ; アルデヒド基の付加脱離 / fructose metabolic process / identical protein binding / cytoplasm類似検索 - 分子機能 Fructose-6-phosphate aldolase / Transaldolase type 3B/Fructose-6-phosphate aldolase / Transaldolase/Fructose-6-phosphate aldolase, archaeal/bacterial / Transaldolase active site. / Transaldolase, active site / Transaldolase signature 1. / Transaldolase/Fructose-6-phosphate aldolase / Transaldolase/Fructose-6-phosphate aldolase / Aldolase class I / Aldolase-type TIM barrel ...Fructose-6-phosphate aldolase / Transaldolase type 3B/Fructose-6-phosphate aldolase / Transaldolase/Fructose-6-phosphate aldolase, archaeal/bacterial / Transaldolase active site. / Transaldolase, active site / Transaldolase signature 1. / Transaldolase/Fructose-6-phosphate aldolase / Transaldolase/Fructose-6-phosphate aldolase / Aldolase class I / Aldolase-type TIM barrel / TIM Barrel / Alpha-Beta Barrel / Alpha Beta類似検索 - ドメイン・相同性 1-hydroxypropan-2-one / thiophene-2-carbaldehyde / Chem-LW2 / Fructose-6-phosphate aldolase 1類似検索 - 構成要素 |
|---|
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.172 Å シンクロトロン / 解像度: 2.172 Å |
|---|
 データ登録者 データ登録者 | Wu, L. / Yang, X.H. / Yu, H.W. / Zhou, J.H. |
|---|
 引用 引用 |  ジャーナル: Chem.Commun.(Camb.) / 年: 2020 ジャーナル: Chem.Commun.(Camb.) / 年: 2020
タイトル: The engineering of decameric d-fructose-6-phosphate aldolase A by combinatorial modulation of inter- and intra-subunit interactions.
著者: Yang, X. / Wu, L. / Li, A. / Ye, L. / Zhou, J. / Yu, H. |
|---|
| 履歴 | | 登録 | 2018年4月13日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年6月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年7月22日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2024年10月23日 | Group: Advisory / Data collection ...Advisory / Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_unobs_or_zero_occ_atoms
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.172 Å
シンクロトロン / 解像度: 2.172 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chem.Commun.(Camb.) / 年: 2020
ジャーナル: Chem.Commun.(Camb.) / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5zol.cif.gz
5zol.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5zol.ent.gz
pdb5zol.ent.gz PDB形式
PDB形式 5zol.json.gz
5zol.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/zo/5zol
https://data.pdbj.org/pub/pdb/validation_reports/zo/5zol ftp://data.pdbj.org/pub/pdb/validation_reports/zo/5zol
ftp://data.pdbj.org/pub/pdb/validation_reports/zo/5zol リンク
リンク 集合体
集合体
 要素
要素











 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL19U1 / 波長: 0.97853 Å
/ ビームライン: BL19U1 / 波長: 0.97853 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj




