+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5yd6 | ||||||
|---|---|---|---|---|---|---|---|
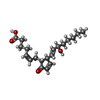
| タイトル | Crystal structure of PG-bound Nurr1-LBD | ||||||
 要素 要素 | Nuclear receptor subfamily 4 group A member 2 | ||||||
 キーワード キーワード | TRANSCRIPTION / NURR1 / LBD / PROSTAGLANDIN / MICHAEL ADDITION | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報general adaptation syndrome / habenula development / central nervous system neuron differentiation / cellular response to corticotropin-releasing hormone stimulus / central nervous system projection neuron axonogenesis / regulation of dopamine metabolic process / : / midbrain dopaminergic neuron differentiation / nuclear glucocorticoid receptor binding / regulation of respiratory gaseous exchange ...general adaptation syndrome / habenula development / central nervous system neuron differentiation / cellular response to corticotropin-releasing hormone stimulus / central nervous system projection neuron axonogenesis / regulation of dopamine metabolic process / : / midbrain dopaminergic neuron differentiation / nuclear glucocorticoid receptor binding / regulation of respiratory gaseous exchange / dopaminergic neuron differentiation / neuron maturation / dopamine biosynthetic process / fat cell differentiation / negative regulation of apoptotic signaling pathway / canonical Wnt signaling pathway / nuclear retinoid X receptor binding / response to amphetamine / adult locomotory behavior / post-embryonic development / neuron migration / SUMOylation of intracellular receptors / beta-catenin binding / Nuclear Receptor transcription pathway / nuclear receptor activity / sequence-specific double-stranded DNA binding / cellular response to oxidative stress / DNA-binding transcription activator activity, RNA polymerase II-specific / neuron apoptotic process / negative regulation of neuron apoptotic process / transcription regulator complex / transcription by RNA polymerase II / response to hypoxia / DNA-binding transcription factor activity, RNA polymerase II-specific / nuclear speck / protein heterodimerization activity / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-templated transcription / chromatin / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / signal transduction / positive regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / zinc ion binding / nucleoplasm / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.34 Å 分子置換 / 解像度: 2.34 Å | ||||||
 データ登録者 データ登録者 | Sreekanth, R. / Yoon, H.S. | ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of Nurr1 bound to cyclopentenone prostaglandin A2 and its mechanism of action in ameliorating dopaminergic neurodegeneration in Drosophila 著者: Rajan, S. / Toh, H.T. / Lim, K.H. / Yoon, H.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5yd6.cif.gz 5yd6.cif.gz | 381.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5yd6.ent.gz pdb5yd6.ent.gz | 312.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5yd6.json.gz 5yd6.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5yd6_validation.pdf.gz 5yd6_validation.pdf.gz | 1.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5yd6_full_validation.pdf.gz 5yd6_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  5yd6_validation.xml.gz 5yd6_validation.xml.gz | 38 KB | 表示 | |
| CIF形式データ |  5yd6_validation.cif.gz 5yd6_validation.cif.gz | 52.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yd/5yd6 https://data.pdbj.org/pub/pdb/validation_reports/yd/5yd6 ftp://data.pdbj.org/pub/pdb/validation_reports/yd/5yd6 ftp://data.pdbj.org/pub/pdb/validation_reports/yd/5yd6 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1ovlS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 30576.256 Da / 分子数: 4 / 断片: UNP residues 328-598 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: NR4A2, NOT, NURR1, TINUR / プラスミド: PET-SUMO / 発現宿主: Homo sapiens (ヒト) / 遺伝子: NR4A2, NOT, NURR1, TINUR / プラスミド: PET-SUMO / 発現宿主:  #2: 化合物 | ChemComp-8SU / (~{ #3: 化合物 | #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.24 Å3/Da / 溶媒含有率: 45.14 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.5 / 詳細: PEG 3350, MES PH 5.5 AND MGCL2 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSRRC NSRRC  / ビームライン: BL13B1 / 波長: 1 Å / ビームライン: BL13B1 / 波長: 1 Å |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2016年4月14日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.34→94.08 Å / Num. obs: 46996 / % possible obs: 99.7 % / 冗長度: 5.7 % / Rmerge(I) obs: 0.072 / Net I/σ(I): 12.8 |
| 反射 シェル | 解像度: 2.34→2.47 Å / 冗長度: 5.9 % / Rmerge(I) obs: 0.746 / Mean I/σ(I) obs: 2.5 / Num. unique obs: 6778 / % possible all: 99.9 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1OVL 解像度: 2.34→20 Å / Cor.coef. Fo:Fc: 0.953 / Cor.coef. Fo:Fc free: 0.933 / SU B: 19.847 / SU ML: 0.233 / 交差検証法: THROUGHOUT / ESU R: 0.396 / ESU R Free: 0.265
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 55.042 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.34→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj