+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5127 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional EM structure of an intact activator-dependent transcription initiation complex | |||||||||
 マップデータ マップデータ | E. coli Class I transcription activation complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transcription / initiation / Class I / activator / RNA polymerase / holoenzyme / sigma70 / open complex / CAP / CRP / cAMP-dependent / DNA / prokaryotic | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報carbon catabolite repression of transcription / sigma factor antagonist complex / DNA binding, bending / RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / minor groove of adenine-thymine-rich DNA binding / regulation of DNA-templated transcription initiation / sigma factor activity / bacterial-type flagellum assembly ...carbon catabolite repression of transcription / sigma factor antagonist complex / DNA binding, bending / RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / minor groove of adenine-thymine-rich DNA binding / regulation of DNA-templated transcription initiation / sigma factor activity / bacterial-type flagellum assembly / bacterial-type RNA polymerase core enzyme binding / cytosolic DNA-directed RNA polymerase complex / bacterial-type flagellum-dependent cell motility / nitrate assimilation / cAMP binding / regulation of DNA-templated transcription elongation / transcription elongation factor complex / transcription antitermination / DNA-directed RNA polymerase complex / cell motility / DNA-templated transcription initiation / protein-DNA complex / ribonucleoside binding / DNA-directed RNA polymerase / DNA-directed RNA polymerase activity / response to heat / protein-containing complex assembly / sequence-specific DNA binding / intracellular iron ion homeostasis / protein dimerization activity / transcription cis-regulatory region binding / DNA-binding transcription factor activity / response to antibiotic / negative regulation of DNA-templated transcription / DNA-templated transcription / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / magnesium ion binding / DNA binding / zinc ion binding / identical protein binding / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 19.8 Å | |||||||||
 データ登録者 データ登録者 | Hudson BP / Quispe J / Lara S / Kim Y / Berman HM / Arnold E / Ebright RH / Lawson CL | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2009 ジャーナル: Proc Natl Acad Sci U S A / 年: 2009タイトル: Three-dimensional EM structure of an intact activator-dependent transcription initiation complex. 著者: Brian P Hudson / Joel Quispe / Samuel Lara-González / Younggyu Kim / Helen M Berman / Eddy Arnold / Richard H Ebright / Catherine L Lawson /  要旨: We present the experimentally determined 3D structure of an intact activator-dependent transcription initiation complex comprising the Escherichia coli catabolite activator protein (CAP), RNA ...We present the experimentally determined 3D structure of an intact activator-dependent transcription initiation complex comprising the Escherichia coli catabolite activator protein (CAP), RNA polymerase holoenzyme (RNAP), and a DNA fragment containing positions -78 to +20 of a Class I CAP-dependent promoter with a CAP site at position -61.5 and a premelted transcription bubble. A 20-A electron microscopy reconstruction was obtained by iterative projection-based matching of single particles visualized in carbon-sandwich negative stain and was fitted using atomic coordinate sets for CAP, RNAP, and DNA. The structure defines the organization of a Class I CAP-RNAP-promoter complex and supports previously proposed interactions of CAP with RNAP alpha subunit C-terminal domain (alphaCTD), interactions of alphaCTD with sigma(70) region 4, interactions of CAP and RNAP with promoter DNA, and phased-DNA-bend-dependent partial wrapping of DNA around the complex. The structure also reveals the positions and shapes of species-specific domains within the RNAP beta', beta, and sigma(70) subunits. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5127.map.gz emd_5127.map.gz | 1.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5127-v30.xml emd-5127-v30.xml emd-5127.xml emd-5127.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5127_1.tif emd_5127_1.tif | 764.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5127 http://ftp.pdbj.org/pub/emdb/structures/EMD-5127 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5127 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5127 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5127.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5127.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli Class I transcription activation complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.64 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
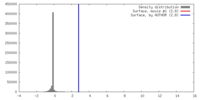
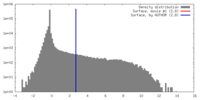
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli RNA polymerase holoenzyme (sigma70) and E. coli catabolit...
| 全体 | 名称: E. coli RNA polymerase holoenzyme (sigma70) and E. coli catabolite activator protein (CAP) bound to 98-mer DNA containing the lac promoter and engineered open transcription bubble |
|---|---|
| 要素 |
|
-超分子 #1000: E. coli RNA polymerase holoenzyme (sigma70) and E. coli catabolit...
| 超分子 | 名称: E. coli RNA polymerase holoenzyme (sigma70) and E. coli catabolite activator protein (CAP) bound to 98-mer DNA containing the lac promoter and engineered open transcription bubble タイプ: sample / ID: 1000 / 詳細: Complex formation was verified by gel shift 集合状態: One molecule of RNAP (containing six subunits) and one CAP homodimer bound to a DNA duplex) Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 570 KDa |
-分子 #1: Catabolite Activator Protein
| 分子 | 名称: Catabolite Activator Protein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: CAP / コピー数: 1 / 集合状態: homodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 50 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: regulation of DNA-templated transcription / InterPro: INTERPRO: IPR001808 |
-分子 #3: RNA polymerase holoenzyme (sigma70)
| 分子 | 名称: RNA polymerase holoenzyme (sigma70) / タイプ: protein_or_peptide / ID: 3 / Name.synonym: RNAP 詳細: Six subunits include alphaI (RpoA), alphaII (RpoA), beta (RpoB), beta prime (RpoC) with C-terminal 6His-tag, omega (RpoZ), and sigma70. コピー数: 1 / 集合状態: heterohexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 460 KDa |
| 組換発現 | 生物種:  組換プラスミド: pEcABC-H6, pRSFduet-sigma, pCDF-omega |
| 配列 | GO: DNA-templated transcription |
-分子 #2: lac(ICAP)UP-UV5-bubble
| 分子 | 名称: lac(ICAP)UP-UV5-bubble / タイプ: dna / ID: 2 / Name.synonym: lac(ICAP)UP-UV5-bubble 詳細: An engineered 98mer duplex based on positions -78 to 20 of the Class I CAP-dependent promoter lac but containing consensus -10 sequence and consensus binding sites for CAP and RNAP alpha-CTD. ...詳細: An engineered 98mer duplex based on positions -78 to 20 of the Class I CAP-dependent promoter lac but containing consensus -10 sequence and consensus binding sites for CAP and RNAP alpha-CTD. Positions -11 to 2 are non-complementary to create an artificial transcription bubble. Top strand 5'-CGCAATAAATGTGATCTAGATCACATTTTAGGCAAAAAAGGCTTTACACTTTATGCTTCCGGCTCGTATAATCGCACCTTATGTGAGCGGATAACAAG-3' Bottom strand 5'-CTTGTTATCCGCTCACAATTCCACACTAATAACGAGCCGGAAGCATAAAGTGTAAAGCCTTTTTTGCCTAAAATGTGATCTAGATCACATTTATTGCG-3' 分類: DNA / Structure: OTHER / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 実験値: 60 KDa / 理論値: 60 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.18 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 25mM HEPES, 100mM KCl, 10mM MgCl2, 1mM DTT, 0.2mM cAMP |
| 染色 | タイプ: NEGATIVE 詳細: Sample and 2% uranyl formate stain were applied to the grid in rapid succession, with the last drop of stain remaining on the sample for 1 minute. The grid was then submerged in stain and ...詳細: Sample and 2% uranyl formate stain were applied to the grid in rapid succession, with the last drop of stain remaining on the sample for 1 minute. The grid was then submerged in stain and brought up under thin carbon to form an upper sandwich layer. The grid was then blotted and dried for 10 minutes. |
| グリッド | 詳細: 400-mesh copper 2.0x0.5 hole pattern C-flat grid covered with thin layer of continuous carbon |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 293 K |
| 詳細 | 15 um pixel size on detector |
| 日付 | 2008年11月4日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F415 (4k x 4k) 実像数: 349 / 平均電子線量: 16 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: standard side-entry room-temperature stage 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | 32816 particles were automatically selected by the Appion DoGpicker initially |
|---|---|
| CTF補正 | 詳細: ACE |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 19.8 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN, SPIDER 詳細: EMAN interleaved with SPIDER correspondence analysis 使用した粒子像数: 14097 |
| 最終 2次元分類 | クラス数: 280 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Chimera, Yup.scx |
| 詳細 | Protocol: rigid body, Yup.scx simulated annealing. A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The model was regularized with PHENIX and the refined against the EM map with Yup.scx using default parameters. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: map-derived potential energy |
| 得られたモデル |  PDB-3iyd: |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Chimera, Yup.scx |
| 詳細 | Protocol: rigid body fit followed by Yup.scx simulated annealing. A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The model was regularized with PHENIX and the refined against the EM map with Yup.scx using default parameters. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: map-derived potential energy |
| 得られたモデル |  PDB-3iyd: |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: Chimera, Yup.scx |
| 詳細 | Protocol: manual fit followed by Yup.scx simulated annealing. A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The model was regularized with PHENIX and the refined against the EM map with Yup.scx using default parameters. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: map-derived potential energy |
| 得られたモデル |  PDB-3iyd: |
-原子モデル構築 4
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: Chimera, Yup.scx |
| 詳細 | Protocol: rigid body fit followed by Yup.scx simulated annealing. A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The model was regularized with PHENIX and the refined against the EM map with Yup.scx using default parameters. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: map-derived potential energy |
| 得られたモデル |  PDB-3iyd: |
-原子モデル構築 5
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Chimera, Modeller, Yup.scx |
| 詳細 | Protocol: rigid body fit followed by Yup.scx simulated annealing. A complete ternary complex model was generated using multiple PDB entries, with a homology modelling step for RNAP. The model was regularized with PHENIX and the refined against the EM map with Yup.scx using default parameters. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: map-derived potential energy |
| 得られたモデル |  PDB-3iyd: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)