+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4432 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MloK1 consensus map from single particle analysis of 2D crystals | |||||||||
 マップデータ マップデータ | MloK1 consensus map obtained from single particle analysis of 2D crystals. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Voltage-gated potassium channel / Cyclic nucleotide-binding domain / ion channel / ion transport / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellularly cyclic nucleotide-activated monoatomic cation channel activity / potassium channel activity / cAMP binding / protein-containing complex binding / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mesorhizobium loti MAFF303099 (根粒菌) / Mesorhizobium loti MAFF303099 (根粒菌) /  Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (根粒菌) Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (根粒菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Righetto R / Biyani N | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Retrieving high-resolution information from disordered 2D crystals by single-particle cryo-EM. 著者: Ricardo D Righetto / Nikhil Biyani / Julia Kowal / Mohamed Chami / Henning Stahlberg /  要旨: Electron crystallography can reveal the structure of membrane proteins within 2D crystals under close-to-native conditions. High-resolution structural information can only be reached if crystals are ...Electron crystallography can reveal the structure of membrane proteins within 2D crystals under close-to-native conditions. High-resolution structural information can only be reached if crystals are perfectly flat and highly ordered. In practice, such crystals are difficult to obtain. Available image unbending algorithms correct for disorder, but only perform well on images of non-tilted, flat crystals, while out-of-plane distortions are not addressed. Here, we present an approach that employs single-particle refinement procedures to locally unbend crystals in 3D. With this method, density maps of the MloK1 potassium channel with a resolution of 4 Å were obtained from images of 2D crystals that do not diffract beyond 10 Å. Furthermore, 3D classification allowed multiple structures to be resolved, revealing a series of MloK1 conformations within a single 2D crystal. This conformational heterogeneity explains the poor diffraction observed and is related to channel function. The approach is implemented in the FOCUS package. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4432.map.gz emd_4432.map.gz | 4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4432-v30.xml emd-4432-v30.xml emd-4432.xml emd-4432.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4432_fsc.xml emd_4432_fsc.xml | 3.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4432.png emd_4432.png | 99 KB | ||
| マスクデータ |  emd_4432_msk_1.map emd_4432_msk_1.map | 4.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4432.cif.gz emd-4432.cif.gz | 6.6 KB | ||
| その他 |  emd_4432_half_map_1.map.gz emd_4432_half_map_1.map.gz emd_4432_half_map_2.map.gz emd_4432_half_map_2.map.gz | 4 MB 4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4432 http://ftp.pdbj.org/pub/emdb/structures/EMD-4432 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4432 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4432 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4432_validation.pdf.gz emd_4432_validation.pdf.gz | 498.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4432_full_validation.pdf.gz emd_4432_full_validation.pdf.gz | 497.3 KB | 表示 | |
| XML形式データ |  emd_4432_validation.xml.gz emd_4432_validation.xml.gz | 5.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4432 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4432 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4432 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4432 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6i9dMC  4439C  4441C  4513C  4514C  4515C  4516C  4517C  4518C  4519C  6iaxC  6qcyC  6qczC  6qd0C  6qd1C  6qd2C  6qd3C  6qd4C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10233 (タイトル: Retrieving High-Resolution Information from Disordered 2D Crystals by Single Particle Cryo-EM EMPIAR-10233 (タイトル: Retrieving High-Resolution Information from Disordered 2D Crystals by Single Particle Cryo-EMData size: 103.0 Data #1: Aligned averages of MloK1 2D crystals in the presence of cAMP [micrographs - single frame] Data #2: Particle stack from MloK1 2D crystals in the presence of cAMP [picked particles - single frame - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4432.map.gz / 形式: CCP4 / 大きさ: 4.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4432.map.gz / 形式: CCP4 / 大きさ: 4.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MloK1 consensus map obtained from single particle analysis of 2D crystals. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4432_msk_1.map emd_4432_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
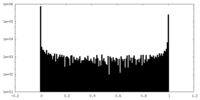
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_4432_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
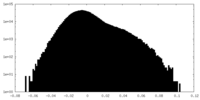
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_4432_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MloK1 potassium channel in lipidic bilayer of 2D crystals
| 全体 | 名称: MloK1 potassium channel in lipidic bilayer of 2D crystals |
|---|---|
| 要素 |
|
-超分子 #1: MloK1 potassium channel in lipidic bilayer of 2D crystals
| 超分子 | 名称: MloK1 potassium channel in lipidic bilayer of 2D crystals タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mesorhizobium loti MAFF303099 (根粒菌) Mesorhizobium loti MAFF303099 (根粒菌) |
| 分子量 | 理論値: 160 KDa |
-分子 #1: Cyclic nucleotide-gated potassium channel mll3241
| 分子 | 名称: Cyclic nucleotide-gated potassium channel mll3241 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (根粒菌) Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (根粒菌)株: LMG 29417 / CECT 9101 / MAFF 303099 |
| 分子量 | 理論値: 37.766297 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVLPFLRIY APLNAVLAAP GLLAVAALTI PDMSGRSRLA LAALLAVIWG AYLLQLAATL LKRRAGVVRD RTPKIAIDVL AVLVPLAAF LLDGSPDWSL YCAVWLLKPL RDSTFFPVLG RVLANEARNL IGVTTLFGVV LFAVALAAYV IERDIQPEKF G SIPQAMWW ...文字列: MSVLPFLRIY APLNAVLAAP GLLAVAALTI PDMSGRSRLA LAALLAVIWG AYLLQLAATL LKRRAGVVRD RTPKIAIDVL AVLVPLAAF LLDGSPDWSL YCAVWLLKPL RDSTFFPVLG RVLANEARNL IGVTTLFGVV LFAVALAAYV IERDIQPEKF G SIPQAMWW AVVTLSTTGY GDTIPQSFAG RVLAGAVMMS GIGIFGLWAG ILATGFYQEV RRGDFVRNWQ LVAAVPLFQK LG PAVLVEI VRALRARTVP AGAVICRIGE PGDRMFFVVE GSVSVATPNP VELGPGAFFG EMALISGEPR SATVSAATTV SLL SLHSAD FQMLCSSSPE IAEIFRKTAL ERRGAAASA UniProtKB: Cyclic nucleotide-gated potassium channel mll3241 |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 詳細: 20 mM KCl, 20 mM Tris-HCl pH 7.6, 1 mM BaCl2, 1 mM EDTA, 0.2 mM cAMP |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 270 / 平均露光時間: 16.0 sec. / 平均電子線量: 45.0 e/Å2 詳細: The dataset of 346 movies recorded and processed for Kowal et al. (2018) 2 was employed. As reported, the data were collected on an FEI Titan Krios TEM equipped with a Gatan K2 DED. Total ...詳細: The dataset of 346 movies recorded and processed for Kowal et al. (2018) 2 was employed. As reported, the data were collected on an FEI Titan Krios TEM equipped with a Gatan K2 DED. Total dose: 40 e-/A^2 distributed over 40 movie frames. Pixel size: 1.3 A on the sample level (counting mode). Nominal tilt range: -55 to +55 degrees. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.3 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | プロトコル: FLEXIBLE FIT | ||||||||||
| 得られたモデル |  PDB-6i9d: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)