+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3zfs | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the F420-reducing NiFe-hydrogenase from a methanogenic archaeon with bound substrate | ||||||
 要素 要素 | (F420-REDUCING HYDROGENASE, SUBUNIT ...) x 3 | ||||||
 キーワード キーワード | OXIDOREDUCTASE / METHANOGENESIS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報coenzyme F420 hydrogenase / coenzyme F420 hydrogenase activity / oxidoreductase activity, acting on CH or CH2 groups, with an iron-sulfur protein as acceptor / ferredoxin hydrogenase activity / iron-sulfur cluster binding / nickel cation binding / flavin adenine dinucleotide binding / 4 iron, 4 sulfur cluster binding 類似検索 - 分子機能 | ||||||
| 生物種 |   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4 Å | ||||||
| Model type details | CA ATOMS ONLY, CHAIN A, B, C | ||||||
 データ登録者 データ登録者 | Mills, D.J. / Vitt, S. / Strauss, M. / Shima, S. / Vonck, J. | ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2013 ジャーナル: Elife / 年: 2013タイトル: De novo modeling of the F(420)-reducing [NiFe]-hydrogenase from a methanogenic archaeon by cryo-electron microscopy. 著者: Deryck J Mills / Stella Vitt / Mike Strauss / Seigo Shima / Janet Vonck /  要旨: Methanogenic archaea use a [NiFe]-hydrogenase, Frh, for oxidation/reduction of F420, an important hydride carrier in the methanogenesis pathway from H2 and CO2. Frh accounts for about 1% of the ...Methanogenic archaea use a [NiFe]-hydrogenase, Frh, for oxidation/reduction of F420, an important hydride carrier in the methanogenesis pathway from H2 and CO2. Frh accounts for about 1% of the cytoplasmic protein and forms a huge complex consisting of FrhABG heterotrimers with each a [NiFe] center, four Fe-S clusters and an FAD. Here, we report the structure determined by near-atomic resolution cryo-EM of Frh with and without bound substrate F420. The polypeptide chains of FrhB, for which there was no homolog, was traced de novo from the EM map. The 1.2-MDa complex contains 12 copies of the heterotrimer, which unexpectedly form a spherical protein shell with a hollow core. The cryo-EM map reveals strong electron density of the chains of metal clusters running parallel to the protein shell, and the F420-binding site is located at the end of the chain near the outside of the spherical structure. DOI:http://dx.doi.org/10.7554/eLife.00218.001. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR PROVIDED. | ||||||
| Remark 700 | SHEET DETERMINATION METHOD: AUTHOR PROVIDED. |
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3zfs.cif.gz 3zfs.cif.gz | 52.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3zfs.ent.gz pdb3zfs.ent.gz | 26.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3zfs.json.gz 3zfs.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3zfs_validation.pdf.gz 3zfs_validation.pdf.gz | 1.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3zfs_full_validation.pdf.gz 3zfs_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  3zfs_validation.xml.gz 3zfs_validation.xml.gz | 21.7 KB | 表示 | |
| CIF形式データ |  3zfs_validation.cif.gz 3zfs_validation.cif.gz | 29.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zf/3zfs https://data.pdbj.org/pub/pdb/validation_reports/zf/3zfs ftp://data.pdbj.org/pub/pdb/validation_reports/zf/3zfs ftp://data.pdbj.org/pub/pdb/validation_reports/zf/3zfs | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-F420-REDUCING HYDROGENASE, SUBUNIT ... , 3種, 3分子 ABC
| #1: タンパク質 | 分子量: 44873.332 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: FRHA CONTAINS A NIFE CENTER 由来: (天然)   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌)株: DSM 2133 / 参照: UniProt: D9PYF9, coenzyme F420 hydrogenase |
|---|---|
| #2: タンパク質 | 分子量: 30267.762 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: FRHG CONTAINS THREE 4FE4S CLUSTERS 由来: (天然)   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌)株: DSM 2133 / 参照: UniProt: D9PYF7, coenzyme F420 hydrogenase |
| #3: タンパク質 | 分子量: 30778.752 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: FRHB CONTAINS FAD AND A 4FE4S CLUSTER 由来: (天然)   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌)株: DSM 2133 / 参照: UniProt: D9PYF6, coenzyme F420 hydrogenase |
-非ポリマー , 6種, 9分子 



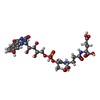






| #4: 化合物 | ChemComp-FCO / | ||||
|---|---|---|---|---|---|
| #5: 化合物 | ChemComp-NI / | ||||
| #6: 化合物 | ChemComp-FE2 / | ||||
| #7: 化合物 | ChemComp-SF4 / #8: 化合物 | ChemComp-F42 / | #9: 化合物 | ChemComp-FAD / | |
-詳細
| 非ポリマーの詳細 | COENZYME F420 (F42): ONLY ISOALLOXAZ |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: F420-REDUCING HYDROGENASE / タイプ: COMPLEX |
|---|---|
| 緩衝液 | 名称: 50 MM TRIS/HCL, 2 MM DTT, 0.025 MM FAD / pH: 7.6 / 詳細: 50 MM TRIS/HCL, 2 MM DTT, 0.025 MM FAD |
| 試料 | 濃度: 0.7 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: HOLEY CARBON |
| 急速凍結 | 装置: FEI VITROBOT MARK I / 凍結剤: ETHANE / 詳細: LIQUID ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI POLARA 300 / 日付: 2011年2月11日 |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 59000 X / 倍率(補正後): 61400 X / 最大 デフォーカス(公称値): 3820 nm / 最小 デフォーカス(公称値): 1650 nm / Cs: 2 mm |
| 試料ホルダ | 温度: 77 K |
| 撮影 | 電子線照射量: 15 e/Å2 / フィルム・検出器のモデル: KODAK SO-163 FILM |
| 画像スキャン | デジタル画像の数: 80 |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: PER MICROGRAPH | ||||||||||||||||
| 対称性 | 点対称性: T (正4面体型対称) | ||||||||||||||||
| 3次元再構成 | 手法: REFINEMENT IN EMAN2 / 解像度: 4 Å / 粒子像の数: 97290 / ピクセルサイズ(公称値): 1.19 Å / ピクセルサイズ(実測値): 1.14 Å / 倍率補正: FIT OF X-RAY MODEL 詳細: SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-2097.(DEPOSITION ID: 10788). 対称性のタイプ: POINT | ||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL / 詳細: REFINEMENT PROTOCOL--RIGID BODY | ||||||||||||||||
| 原子モデル構築 | PDB-ID: 2WPN Accession code: 2WPN / Source name: PDB / タイプ: experimental model | ||||||||||||||||
| 精密化 | 最高解像度: 4 Å | ||||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 4 Å
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj

















