+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human C-terminally bound ATG9A-ATG2A-WIPI4 complex | |||||||||
 マップデータ マップデータ | Cryo-EM map of C-terminally bound ATG9A-ATG2A-WIPI4 complex. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Lipid transfer / ATG9A-ATG2A-WIPI4 complex / single particle cryo-EM / autophagy / LIPID TRANSPORT-MEMBRANE PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phagophore / lipid transfer activity / organelle membrane contact site / phosphatidylinositol phosphate binding / glycophagy / autophagy of peroxisome / phospholipid scramblase activity / nucleophagy / programmed necrotic cell death / positive regulation of autophagosome assembly ...phagophore / lipid transfer activity / organelle membrane contact site / phosphatidylinositol phosphate binding / glycophagy / autophagy of peroxisome / phospholipid scramblase activity / nucleophagy / programmed necrotic cell death / positive regulation of autophagosome assembly / protein localization to phagophore assembly site / phagophore assembly site membrane / piecemeal microautophagy of the nucleus / phosphatidylinositol-3-phosphate binding / phagophore assembly site / bone morphogenesis / phosphatidylinositol-3,5-bisphosphate binding / reticulophagy / extrinsic component of membrane / autophagy of mitochondrion / Macroautophagy / autophagosome assembly / protein-membrane adaptor activity / autophagosome / cellular response to starvation / lipid droplet / PINK1-PRKN Mediated Mitophagy / mitochondrial membrane / trans-Golgi network / recycling endosome / autophagy / recycling endosome membrane / late endosome / late endosome membrane / endosome / Golgi membrane / intracellular membrane-bounded organelle / endoplasmic reticulum membrane / protein kinase binding / Golgi apparatus / mitochondrion / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
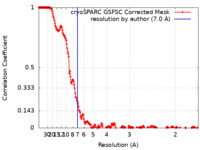
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.0 Å | |||||||||
 データ登録者 データ登録者 | Wang Y / Stjepanovic G | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural basis for lipid transfer by the ATG2A-ATG9A complex. 著者: Yang Wang / Selma Dahmane / Rujuan Ti / Xinyi Mai / Lizhe Zhu / Lars-Anders Carlson / Goran Stjepanovic /   要旨: Autophagy is characterized by the formation of double-membrane vesicles called autophagosomes. Autophagy-related proteins (ATGs) 2A and 9A have an essential role in autophagy by mediating lipid ...Autophagy is characterized by the formation of double-membrane vesicles called autophagosomes. Autophagy-related proteins (ATGs) 2A and 9A have an essential role in autophagy by mediating lipid transfer and re-equilibration between membranes for autophagosome formation. Here we report the cryo-electron microscopy structures of human ATG2A in complex with WD-repeat protein interacting with phosphoinositides 4 (WIPI4) at 3.2 Å and the ATG2A-WIPI4-ATG9A complex at 7 Å global resolution. On the basis of molecular dynamics simulations, we propose a mechanism of lipid extraction from the donor membranes. Our analysis revealed 3:1 stoichiometry of the ATG9A-ATG2A complex, directly aligning the ATG9A lateral pore with ATG2A lipid transfer cavity, and an interaction of the ATG9A trimer with both the N-terminal and the C-terminal tip of rod-shaped ATG2A. Cryo-electron tomography of ATG2A liposome-binding states showed that ATG2A tethers lipid vesicles at different orientations. In summary, this study provides a molecular basis for the growth of the phagophore membrane and lends structural insights into spatially coupled lipid transport and re-equilibration during autophagosome formation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37091.map.gz emd_37091.map.gz | 776.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37091-v30.xml emd-37091-v30.xml emd-37091.xml emd-37091.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37091_fsc.xml emd_37091_fsc.xml | 20.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37091.png emd_37091.png | 53.2 KB | ||
| Filedesc metadata |  emd-37091.cif.gz emd-37091.cif.gz | 7.5 KB | ||
| その他 |  emd_37091_half_map_1.map.gz emd_37091_half_map_1.map.gz emd_37091_half_map_2.map.gz emd_37091_half_map_2.map.gz | 765 MB 765 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37091 http://ftp.pdbj.org/pub/emdb/structures/EMD-37091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37091 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37091 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37091_validation.pdf.gz emd_37091_validation.pdf.gz | 711.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37091_full_validation.pdf.gz emd_37091_full_validation.pdf.gz | 710.9 KB | 表示 | |
| XML形式データ |  emd_37091_validation.xml.gz emd_37091_validation.xml.gz | 28.2 KB | 表示 | |
| CIF形式データ |  emd_37091_validation.cif.gz emd_37091_validation.cif.gz | 37.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37091 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37091 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37091 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37091.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37091.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
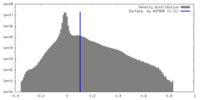
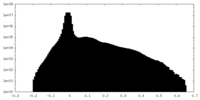
| 注釈 | Cryo-EM map of C-terminally bound ATG9A-ATG2A-WIPI4 complex. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ |
| ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map B of C-terminally bound ATG9A-ATG2A-WIPI4 complex.
| ファイル | emd_37091_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
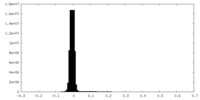
| 注釈 | Half map B of C-terminally bound ATG9A-ATG2A-WIPI4 complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C-terminally bound ATG9A-ATG2A-WIPI4 complex
| 全体 | 名称: C-terminally bound ATG9A-ATG2A-WIPI4 complex |
|---|---|
| 要素 |
|
-超分子 #1: C-terminally bound ATG9A-ATG2A-WIPI4 complex
| 超分子 | 名称: C-terminally bound ATG9A-ATG2A-WIPI4 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 536 KDa |
-分子 #1: Autophagy-related protein 9A
| 分子 | 名称: Autophagy-related protein 9A / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 94.551031 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAQFDTEYQR LEASYSDSPP GEEDLLVHVA EGSKSPWHHI ENLDLFFSRV YNLHQKNGFT CMLIGEIFEL MQFLFVVAFT TFLVSCVDY DILFANKMVN HSLHPTEPVK VTLPDAFLPA QVCSARIQEN GSLITILVIA GVFWIHRLIK FIYNICCYWE I HSFYLHAL ...文字列: MAQFDTEYQR LEASYSDSPP GEEDLLVHVA EGSKSPWHHI ENLDLFFSRV YNLHQKNGFT CMLIGEIFEL MQFLFVVAFT TFLVSCVDY DILFANKMVN HSLHPTEPVK VTLPDAFLPA QVCSARIQEN GSLITILVIA GVFWIHRLIK FIYNICCYWE I HSFYLHAL RIPMSALPYC TWQEVQARIV QTQKEHQICI HKRELTELDI YHRILRFQNY MVALVNKSLL PLRFRLPGLG EA VFFTRGL KYNFELILFW GPGSLFLNEW SLKAEYKRGG QRLELAQRLS NRILWIGIAN FLLCPLILIW QILYAFFSYA EVL KREPGA LGARCWSLYG RCYLRHFNEL EHELQSRLNR GYKPASKYMN CFLSPLLTLL AKNGAFFAGS ILAVLIALTI YDED VLAVE HVLTTVTLLG VTVTVCRSFI PDQHMVFCPE QLLRVILAHI HYMPDHWQGN AHRSQTRDEF AQLFQYKAVF ILEEL LSPI VTPLILIFCL RPRALEIIDF FRNFTVEVVG VGDTCSFAQM DVRQHGHPQW LSAGQTEASV YQQAEDGKTE LSLMHF AIT NPGWQPPRES TAFLGFLKEQ VQRDGAAASL AQGGLLPENA LFTSIQSLQS ESEPLSLIAN VVAGSSCRGP PLPRDLQ GS RHRAEVASAL RSFSPLQPGQ APTGRAHSTM TGSGVDARTA SSGSSVWEGQ LQSLVLSEYA STEMSLHALY MHQLHKQQ A QAEPERHVWH RRESDESGES APDEGGEGAR APQSIPRSAS YPCAAPRPGA PETTALHGGF QRRYGGITDP GTVPRVPSH FSRLPLGGWA EDGQSASRHP EPVPEEGSED ELPPQVHKV UniProtKB: Autophagy-related protein 9A |
-分子 #2: WD repeat domain phosphoinositide-interacting protein 4
| 分子 | 名称: WD repeat domain phosphoinositide-interacting protein 4 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 39.916539 KDa |
| 組換発現 | 生物種:  Baculovirus expression vector pFastBac1-HM (ウイルス) Baculovirus expression vector pFastBac1-HM (ウイルス) |
| 配列 | 文字列: MTQQPLRGVT SLRFNQDQSC FCCAMETGVR IYNVEPLMEK GHLDHEQVGS MGLVEMLHRS NLLALVGGGS SPKFSEISVL IWDDAREGK DSKEKLVLEF TFTKPVLSVR MRHDKIVIVL KNRIYVYSFP DNPRKLFEFD TRDNPKGLCD LCPSLEKQLL V FPGHKCGS ...文字列: MTQQPLRGVT SLRFNQDQSC FCCAMETGVR IYNVEPLMEK GHLDHEQVGS MGLVEMLHRS NLLALVGGGS SPKFSEISVL IWDDAREGK DSKEKLVLEF TFTKPVLSVR MRHDKIVIVL KNRIYVYSFP DNPRKLFEFD TRDNPKGLCD LCPSLEKQLL V FPGHKCGS LQLVDLASTK PGTSSAPFTI NAHQSDIACV SLNQPGTVVA SASQKGTLIR LFDTQSKEKL VELRRGTDPA TL YCINFSH DSSFLCASSD KGTVHIFALK DTRLNRRSAL ARVGKVGPMI GQYVDSQWSL ASFTVPAESA CICAFGRNTS KNV NSVIAI CVDGTFHKYV FTPDGNCNRE AFDVYLDICD DDDF UniProtKB: WD repeat domain phosphoinositide-interacting protein 4 |
-分子 #3: Autophagy-related protein 2 homolog A
| 分子 | 名称: Autophagy-related protein 2 homolog A / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 213.100281 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSRWLWPWSN CVKERVCRYL LHHYLGHFFQ EHLSLDQLSL DLYKGSVALR DIHLEIWSVN EVLESMESPL ELVEGFVGSI EVAVPWAAL LTDHCTVRVS GLQLTLQPRR GPAPGAADSQ SWASCMTTSL QLAQECLRDG LPEPSEPPQP LEGLEMFAQT I ETVLRRIK ...文字列: MSRWLWPWSN CVKERVCRYL LHHYLGHFFQ EHLSLDQLSL DLYKGSVALR DIHLEIWSVN EVLESMESPL ELVEGFVGSI EVAVPWAAL LTDHCTVRVS GLQLTLQPRR GPAPGAADSQ SWASCMTTSL QLAQECLRDG LPEPSEPPQP LEGLEMFAQT I ETVLRRIK VTFLDTVVRV EHSPGDGERG VAVEVRVQRL EYCDEAVRDP SQAPPVDVHQ PPAFLHKLLQ LAGVRLHYEE LP AQEEPPE PPLQIGSCSG YMELMVKLKQ NEAFPGPKLE VAGQLGSLHL LLTPRQLQQL QELLSAVSLT DHEGLADKLN KSR PLGAED LWLIEQDLNQ QLQAGAVAEP LSPDPLTNPL LNLDNTDLFF SMAGLTSSVA SALSELSLSD VDLASSVRSD MASR RLSAQ AHPAGKMAPN PLLDTMRPDS LLKMTLGGVT LTLLQTSAPS SGPPDLATHF FTEFDATKDG PFGSRDFHHL RPRFQ RACP CSHVRLTGTA VQLSWELRTG SRGRRTTSME VHFGQLEVLE CLWPRGTSEP EYTEILTFPG TLGSQASARP CAHLRH TQI LRRVPKSRPR RSVACHCHSE LALDLANFQA DVELGALDRL AALLRLATVP AEPPAGLLTE PLPAMEQQTV FRLSAPR AT LRLRFPIADL RPEPDPWAGQ AVRAEQLRLE LSEPQFRSEL SSGPGPPVPT HLELTCSDLH GIYEDGGKPP VPCLRVSK A LDPKSTGRKY FLPQVVVTVN PQSSSTQWEV APEKGEELEL SVESPCELRE PEPSPFSSKR TMYETEEMVI PGDPEEMRT FQSRTLALSR CSLEVILPSV HIFLPSKEVY ESIYNRINND LLMWEPADLL PTPDPAAQPS GFPGPSGFWH DSFKMCKSAF KLANCFDLT PDSDSDDEDA HFFSVGASGG PQAAAPEAPS LHLQSTFSTL VTVLKGRITA LCETKDEGGK RLEAVHGELV L DMEHGTLF SVSQYCGQPG LGYFCLEAEK ATLYHRAAVD DYPLPSHLDL PSFAPPAQLA PTIYPSEEGV TERGASGRKG QG RGPHMLS TAVRIHLDPH KNVKEFLVTL RLHKATLRHY MALPEQSWHS QLLEFLDVLD DPVLGYLPPT VITILHTHLF SCS VDYRPL YLPVRVLITA ETFTLSSNII MDTSTFLLRF ILDDSALYLS DKCEVETLDL RRDYVCVLDV DLLELVIKTW KGST EGKLS QPLFELRCSN NVVHVHSCAD SCALLVNLLQ YVMSTGDLHP PPRPPSPTEI AGQKLSESPA SLPSCPPVET ALINQ RDLA DALLDTERSL RELAQPSGGH LPQASPISVY LFPGERSGAP PPSPPVGGPA GSLGSCSEEK EDEREEEGDG DTLDSD EFC ILDAPGLGIP PRDGEPVVTQ LHPGPIVVRD GYFSRPIGST DLLRAPAHFP VPSTRVVLRE VSLVWHLYGG RDFGPHP GH RARTGLSGPR SSPSRCSGPN RPQNSWRTQG GSGRQHHVLM EIQLSKVSFQ HEVYPAEPAT GPAAPSQELE ERPLSRQV F IVQELEVRDR LASSQINKFL YLHTSERMPR RAHSNMLTIK ALHVAPTTNL GGPECCLRVS LMPLRLNVDQ DALFFLKDF FTSLVAGINP VVPGETSAEA RPETRAQPSS PLEGQAEGVE TTGSQEAPGG GHSPSPPDQQ PIYFREFRFT SEVPIWLDYH GKHVTMDQV GTFAGLLIGL AQLNCSELKL KRLCCRHGLL GVDKVLGYAL NEWLQDIRKN QLPGLLGGVG PMHSVVQLFQ G FRDLLWLP IEQYRKDGRL MRGLQRGAAS FGSSTASAAL ELSNRLVQAI QATAETVYDI LSPAAPVSRS LQDKRSARRL RR GQQPADL REGVAKAYDT VREGILDTAQ TICDVASRGH EQKGLTGAVG GVIRQLPPTV VKPLILATEA TSSLLGGMRN QIV PDAHKD HALKWRSDSA QD UniProtKB: Autophagy-related protein 2 homolog A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 65.17 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)