+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31595 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the hedgehog release protein Disp from water bear (Hypsibius dujardini) | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | cryo-EM / Dispatched / Hedgehog release / Hedgehog signaling / membrane protein / setrol sensing domain / water bear | |||||||||||||||||||||
| 機能・相同性 | Membrane transport protein MMPL domain / MMPL family / Sterol-sensing domain (SSD) profile. / Sterol-sensing domain / membrane => GO:0016020 / plasma membrane / Protein dispatched-like protein 1 機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  Hypsibius dujardini (無脊椎動物) Hypsibius dujardini (無脊椎動物) | |||||||||||||||||||||
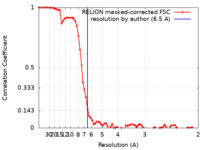
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.5 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Luo Y / Wan G | |||||||||||||||||||||
| 資金援助 |  中国, 6件 中国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Front Mol Biosci / 年: 2021 ジャーナル: Front Mol Biosci / 年: 2021タイトル: Architecture of Dispatched, a Transmembrane Protein Responsible for Hedgehog Release. 著者: Yitian Luo / Guoyue Wan / Xuan Zhou / Qiuwen Wang / Yunbin Zhang / Juan Bao / Yao Cong / Yun Zhao / Dianfan Li /  要旨: The evolutionarily conserved Hedgehog (Hh) signaling pathway is crucial for programmed cell differentiation and proliferation. Dispatched (Disp) is a 12-transmembrane protein that plays a critical ...The evolutionarily conserved Hedgehog (Hh) signaling pathway is crucial for programmed cell differentiation and proliferation. Dispatched (Disp) is a 12-transmembrane protein that plays a critical role in the Hedgehog (Hh) signaling pathway by releasing the dually lipidated ligand HhN from the membrane, a prerequisite step to the downstream signaling cascade. In this study, we focus on the Disp from water bear, a primitive animal known as the most indestructible on Earth. Using a zebrafish model, we show that the water bear homolog possesses the function of Disp. We have solved its structure to a 6.5-Å resolution using single-particle cryogenic electron microscopy. Consistent with the evolutional conservation of the pathway, the water bear Disp structure is overall similar to the previously reported structures of the fruit fly and human homologs. Although not revealing much detail at this resolution, the water bear Disp shows a different conformation compared to published structures, suggesting that they represent different functional snapshots. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31595.map.gz emd_31595.map.gz | 3.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31595-v30.xml emd-31595-v30.xml emd-31595.xml emd-31595.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31595_fsc.xml emd_31595_fsc.xml | 7.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31595.png emd_31595.png | 61.4 KB | ||
| Filedesc metadata |  emd-31595.cif.gz emd-31595.cif.gz | 6.5 KB | ||
| その他 |  emd_31595_half_map_1.map.gz emd_31595_half_map_1.map.gz emd_31595_half_map_2.map.gz emd_31595_half_map_2.map.gz | 26.4 MB 26.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31595 http://ftp.pdbj.org/pub/emdb/structures/EMD-31595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31595 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31595_validation.pdf.gz emd_31595_validation.pdf.gz | 830.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31595_full_validation.pdf.gz emd_31595_full_validation.pdf.gz | 830.5 KB | 表示 | |
| XML形式データ |  emd_31595_validation.xml.gz emd_31595_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_31595_validation.cif.gz emd_31595_validation.cif.gz | 17.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31595 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31595 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31595.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31595.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_31595_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_31595_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dispatched
| 全体 | 名称: Dispatched |
|---|---|
| 要素 |
|
-超分子 #1: Dispatched
| 超分子 | 名称: Dispatched / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Hypsibius dujardini (無脊椎動物) Hypsibius dujardini (無脊椎動物) |
| 分子量 | 理論値: 130 kDa/nm |
-分子 #1: Protein dispatched-like protein 1
| 分子 | 名称: Protein dispatched-like protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hypsibius dujardini (無脊椎動物) Hypsibius dujardini (無脊椎動物) |
| 分子量 | 理論値: 124.943414 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGSHHHHHHH HHHHHGSPRQ LVRRRKKTVM QRYAHFLAHH PFLILTLTFC ANIVLLLVIV CTDSLPAFDD PQAGFEPRGS LINQRAQAW RNFLERDGVV QPSTRRKATT TKPVAARQNA PVQEIGTGLR NSTVKENVFS SSGMASLGPI TQPRGFFCSR P LLGHAKLI ...文字列: MGSHHHHHHH HHHHHGSPRQ LVRRRKKTVM QRYAHFLAHH PFLILTLTFC ANIVLLLVIV CTDSLPAFDD PQAGFEPRGS LINQRAQAW RNFLERDGVV QPSTRRKATT TKPVAARQNA PVQEIGTGLR NSTVKENVFS SSGMASLGPI TQPRGFFCSR P LLGHAKLI LRRKDAGNLL NLEGIRELCQ LDADIRRLDE FQSCSEESAD TGRLCRTWNL PNYVAVLAGK TSCHQLEEHD IR NVLNLTA TCLPFYRSGQ LKADCSTSED DGKTMQEDTI TCPGVPTDCI KDSSIYHLLY FISDKESRET GYSSMKHTLA VLP VWSSPA ALPLYRALQW GNFTRPDDDG PIQVIGMEMG LGDRLFNTAL LEDTLYVGVA AGVVVVVLWL YTGSLFVTLM NLMA IFFSL VVSYFLYVFV FRLTFFPFMN LLTCVIIVAI GADALVIFAR LWHLAKTEKD DGRFEKVVHA TFRHAAQAIL VSSLT ASAA LFSDIVNPII AIRCFSIFAG LTVLVHLFFA VTWMPACFVV ADKWGSSVYV CTKSPVCRLL YRCRCLHSGL RQYCDY FRI FFEKLLPCLV IRLKWMWMAS FVLLTVGACV VVFVFPGLQL ASGRTFSLWN SGHPSEQYRL LKDRFAFEEN RNLNNNE KV SLHFVWGVLA LNQAALLDPT DTGITTMDGR FNMSDPLSQI WMLKFCADLR QQTFFDNSTQ SDNACYFDSF MLWMETGA C SGHSQFPYPP ATFIRCVHTF SNSFPQAKHF GPNFNSLHQI DSFVLRLQTS QLFSHSYTAM QQLHQHVDQW FTAALRTAP PSLQGAWFTG DFAFFDLQQS LISGTALSLI VSLFVAFLVL FFTTLNVGVS LIAITVIAGI MLATTAALVL MEWQLSVFES TIIGLAIGL SVDFTLHYAV SYCCAESYEE RELKTNIVIS EMASCVTMSA VTTFLAGALM IPSDILFYRQ LGLFIITVTA I SLLYATIF LPACLAVLGP QGAFLQFHYP SCRPLCCRPD PSKLVEKSMH SSAEFDTTYT TGGTYDHHRE TKQCFHQSIN QP YQGKALR SPSAAANNHH VVVISATPEQ PPKTHPALLL DVDPDLADEE TDSVGELGSV PTSRGPSRGT SLIGTLEVLF Q UniProtKB: Protein dispatched-like protein 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 3 / 実像数: 5380 / 平均露光時間: 7.6 sec. / 平均電子線量: 60.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X