+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bulky DNA lesion recognition complex 3 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | ||||||||||||
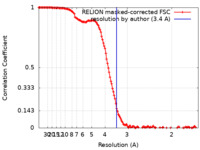
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Kim J / Yang W | ||||||||||||
| 資金援助 |  米国, 米国,  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Lesion recognition by XPC, TFIIH and XPA in DNA excision repair. 著者: Jinseok Kim / Chia-Lung Li / Xuemin Chen / Yanxiang Cui / Filip M Golebiowski / Huaibin Wang / Fumio Hanaoka / Kaoru Sugasawa / Wei Yang /     要旨: Nucleotide excision repair removes DNA lesions caused by ultraviolet light, cisplatin-like compounds and bulky adducts. After initial recognition by XPC in global genome repair or a stalled RNA ...Nucleotide excision repair removes DNA lesions caused by ultraviolet light, cisplatin-like compounds and bulky adducts. After initial recognition by XPC in global genome repair or a stalled RNA polymerase in transcription-coupled repair, damaged DNA is transferred to the seven-subunit TFIIH core complex (Core7) for verification and dual incisions by the XPF and XPG nucleases. Structures capturing lesion recognition by the yeast XPC homologue Rad4 and TFIIH in transcription initiation or DNA repair have been separately reported. How two different lesion recognition pathways converge and how the XPB and XPD helicases of Core7 move the DNA lesion for verification are unclear. Here we report on structures revealing DNA lesion recognition by human XPC and DNA lesion hand-off from XPC to Core7 and XPA. XPA, which binds between XPB and XPD, kinks the DNA duplex and shifts XPC and the DNA lesion by nearly a helical turn relative to Core7. The DNA lesion is thus positioned outside of Core7, as would occur with RNA polymerase. XPB and XPD, which track the lesion-containing strand but translocate DNA in opposite directions, push and pull the lesion-containing strand into XPD for verification. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29673.map.gz emd_29673.map.gz | 10.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29673-v30.xml emd-29673-v30.xml emd-29673.xml emd-29673.xml | 31.9 KB 31.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29673_fsc.xml emd_29673_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29673.png emd_29673.png | 103.7 KB | ||
| その他 |  emd_29673_half_map_1.map.gz emd_29673_half_map_1.map.gz emd_29673_half_map_2.map.gz emd_29673_half_map_2.map.gz | 155.4 MB 155.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29673 http://ftp.pdbj.org/pub/emdb/structures/EMD-29673 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29673 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29673 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29673_validation.pdf.gz emd_29673_validation.pdf.gz | 679 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29673_full_validation.pdf.gz emd_29673_full_validation.pdf.gz | 678.6 KB | 表示 | |
| XML形式データ |  emd_29673_validation.xml.gz emd_29673_validation.xml.gz | 20.9 KB | 表示 | |
| CIF形式データ |  emd_29673_validation.cif.gz emd_29673_validation.cif.gz | 27.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29673 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29673 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29673 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29673 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29673.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29673.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.833 Å | ||||||||||||||||||||||||||||||||||||
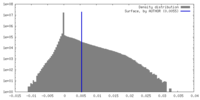
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_29673_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
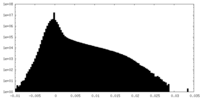
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_29673_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : bulky DNA lesion recognition complex 3
+超分子 #1: bulky DNA lesion recognition complex 3
+分子 #1: General transcription and DNA repair factor IIH helicase subunit XPB
+分子 #2: TFIIH basal transcription factor complex helicase XPD subunit
+分子 #3: General transcription factor IIH subunit 1
+分子 #4: General transcription factor IIH subunit 4
+分子 #5: General transcription factor IIH subunit 2
+分子 #6: General transcription factor IIH subunit 3
+分子 #7: General transcription factor IIH subunit 5
+分子 #8: DNA repair protein complementing XP-C cells
+分子 #9: DNA repair protein complementing XP-A cells
+分子 #10: DNA1
+分子 #11: DNA2
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.9 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 6243 / 平均露光時間: 2.5 sec. / 平均電子線量: 54.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
|---|
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)