+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Methylococcus capsulatus (Bath) pMMO treated with potassium cyanide in a native lipid nanodisc at 3.65 Angstrom resolution | |||||||||
 マップデータ マップデータ | CryoEM structure of Methylococcus capsulatus (Bath) pMMO treated with potassium cyanide in a native lipid nanodisc at 3.65 angstrom resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | complex / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methane monooxygenase (particulate) / methane monooxygenase (soluble) / : / : / monooxygenase activity / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.65 Å | |||||||||
 データ登録者 データ登録者 | Koo CW / Rosenzweig AC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Recovery of particulate methane monooxygenase structure and activity in a lipid bilayer. 著者: Christopher W Koo / Frank J Tucci / Yuan He / Amy C Rosenzweig /  要旨: Bacterial methane oxidation using the enzyme particulate methane monooxygenase (pMMO) contributes to the removal of environmental methane, a potent greenhouse gas. Crystal structures determined using ...Bacterial methane oxidation using the enzyme particulate methane monooxygenase (pMMO) contributes to the removal of environmental methane, a potent greenhouse gas. Crystal structures determined using inactive, detergent-solubilized pMMO lack several conserved regions neighboring the proposed active site. We show that reconstituting pMMO in nanodiscs with lipids extracted from the native organism restores methane oxidation activity. Multiple nanodisc-embedded pMMO structures determined by cryo-electron microscopy to 2.14- to 2.46-angstrom resolution reveal the structure of pMMO in a lipid environment. The resulting model includes stabilizing lipids, regions of the PmoA and PmoC subunits not observed in prior structures, and a previously undetected copper-binding site in the PmoC subunit with an adjacent hydrophobic cavity. These structures provide a revised framework for understanding and engineering pMMO function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25683.map.gz emd_25683.map.gz | 96.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25683-v30.xml emd-25683-v30.xml emd-25683.xml emd-25683.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
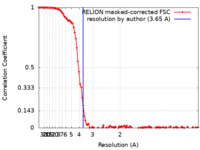
| FSC (解像度算出) |  emd_25683_fsc.xml emd_25683_fsc.xml | 10.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25683.png emd_25683.png | 126.9 KB | ||
| Filedesc metadata |  emd-25683.cif.gz emd-25683.cif.gz | 5.8 KB | ||
| その他 |  emd_25683_additional_1.map.gz emd_25683_additional_1.map.gz | 90.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25683 http://ftp.pdbj.org/pub/emdb/structures/EMD-25683 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25683 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25683 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25683_validation.pdf.gz emd_25683_validation.pdf.gz | 642.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25683_full_validation.pdf.gz emd_25683_full_validation.pdf.gz | 641.6 KB | 表示 | |
| XML形式データ |  emd_25683_validation.xml.gz emd_25683_validation.xml.gz | 11.7 KB | 表示 | |
| CIF形式データ |  emd_25683_validation.cif.gz emd_25683_validation.cif.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25683 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25683 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25683 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25683 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7t4oMC  7s4hC  7s4iC  7s4jC  7s4kC  7s4lC  7s4mC  7t4pC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25683.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25683.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of Methylococcus capsulatus (Bath) pMMO treated with potassium cyanide in a native lipid nanodisc at 3.65 angstrom resolution | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.511 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: CryoEM structure of Methylococcus capsulatus (Bath) pMMO treated...
| ファイル | emd_25683_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of Methylococcus capsulatus (Bath) pMMO treated with potassium cyanide in a native lipid nanodisc at 3.65 angstrom resolution, unsharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
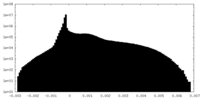
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : pMMO complex in a native lipid nanodisc
| 全体 | 名称: pMMO complex in a native lipid nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: pMMO complex in a native lipid nanodisc
| 超分子 | 名称: pMMO complex in a native lipid nanodisc / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア) |
-分子 #1: Particulate methane monooxygenase alpha subunit
| 分子 | 名称: Particulate methane monooxygenase alpha subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: methane monooxygenase (particulate) |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア)株: ATCC 33009 / NCIMB 11132 / Bath |
| 分子量 | 理論値: 46.129746 KDa |
| 配列 | 文字列: MKTIKDRIAK WSAIGLLSAV AATAFYAPSA SAHGEKSQAA FMRMRTIHWY DLSWSKEKVK INETVEIKGK FHVFEGWPET VDEPDVAFL NVGMPGPVFI RKESYIGGQL VPRSVRLEIG KTYDFRVVLK ARRPGDWHVH TMMNVQGGGP IIGPGKWITV E GSMSEFRN ...文字列: MKTIKDRIAK WSAIGLLSAV AATAFYAPSA SAHGEKSQAA FMRMRTIHWY DLSWSKEKVK INETVEIKGK FHVFEGWPET VDEPDVAFL NVGMPGPVFI RKESYIGGQL VPRSVRLEIG KTYDFRVVLK ARRPGDWHVH TMMNVQGGGP IIGPGKWITV E GSMSEFRN PVTTLTGQTV DLENYNEGNT YFWHAFWFAI GVAWIGYWSR RPIFIPRLLM VDAGRADELV SATDRKVAMG FL AATILIV VMAMSSANSK YPITIPLQAG TMRGMKPLEL PAPTVSVKVE DATYRVPGRA MRMKLTITNH GNSPIRLGEF YTA SVRFLD SDVYKDTTGY PEDLLAEDGL SVSDNSPLAP GETRTVDVTA SDAAWEVYRL SDIIYDPDSR FAGLLFFFDA TGNR QVVQI DAPLIPSFM UniProtKB: Particulate methane monooxygenase alpha subunit |
-分子 #2: Ammonia monooxygenase/methane monooxygenase, subunit C family protein
| 分子 | 名称: Ammonia monooxygenase/methane monooxygenase, subunit C family protein タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号: methane monooxygenase (soluble) |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア)株: ATCC 33009 / NCIMB 11132 / Bath |
| 分子量 | 理論値: 29.839309 KDa |
| 配列 | 文字列: MAATTIGGAA AAEAPLLDKK WLTFALAIYT VFYLWVRWYE GVYGWSAGLD SFAPEFETYW MNFLYTEIVL EIVTASILWG YLWKTRDRN LAALTPREEL RRNFTHLVWL VAYAWAIYWG ASYFTEQDGT WHQTIVRDTD FTPSHIIEFY LSYPIYIITG F AAFIYAKT ...文字列: MAATTIGGAA AAEAPLLDKK WLTFALAIYT VFYLWVRWYE GVYGWSAGLD SFAPEFETYW MNFLYTEIVL EIVTASILWG YLWKTRDRN LAALTPREEL RRNFTHLVWL VAYAWAIYWG ASYFTEQDGT WHQTIVRDTD FTPSHIIEFY LSYPIYIITG F AAFIYAKT RLPFFAKGIS LPYLVLVVGP FMILPNVGLN EWGHTFWFME ELFVAPLHYG FVIFGWLALA VMGTLTQTFY SF AQGGLGQ SLCEAVDEGL IAK UniProtKB: Ammonia monooxygenase/methane monooxygenase, subunit C family protein |
-分子 #3: Particulate methane monooxygenase beta subunit
| 分子 | 名称: Particulate methane monooxygenase beta subunit / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO / EC番号: methane monooxygenase (particulate) |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア)株: ATCC 33009 / NCIMB 11132 / Bath |
| 分子量 | 理論値: 28.445098 KDa |
| 配列 | 文字列: MSAAQSAVRS HAEAVQVSRT IDWMALFVVF FVIVGSYHIH AMLTMGDWDF WSDWKDRRLW VTVTPIVLVT FPAAVQSYLW ERYRLPWGA TVCVLGLLLG EWINRYFNFW GWTYFPINFV FPASLVPGAI ILDTVLMLSG SYLFTAIVGA MGWGLIFYPG N WPIIAPLH ...文字列: MSAAQSAVRS HAEAVQVSRT IDWMALFVVF FVIVGSYHIH AMLTMGDWDF WSDWKDRRLW VTVTPIVLVT FPAAVQSYLW ERYRLPWGA TVCVLGLLLG EWINRYFNFW GWTYFPINFV FPASLVPGAI ILDTVLMLSG SYLFTAIVGA MGWGLIFYPG N WPIIAPLH VPVEYNGMLM SIADIQGYNY VRTGTPEYIR MVEKGTLRTF GKDVAPVSAF FSAFMSILIY FMWHFIGRWF SN ERFLQST UniProtKB: Particulate methane monooxygenase beta subunit |
-分子 #4: COPPER (II) ION
| 分子 | 名称: COPPER (II) ION / タイプ: ligand / ID: 4 / コピー数: 5 / 式: CU |
|---|---|
| 分子量 | 理論値: 63.546 Da |
| Chemical component information |  ChemComp-CU: |
-分子 #5: 1,2-DIDECANOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIDECANOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 5 / コピー数: 9 / 式: P1O |
|---|---|
| 分子量 | 理論値: 566.728 Da |
| Chemical component information | 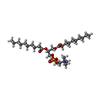 ChemComp-P1O: |
-分子 #6: DIUNDECYL PHOSPHATIDYL CHOLINE
| 分子 | 名称: DIUNDECYL PHOSPHATIDYL CHOLINE / タイプ: ligand / ID: 6 / コピー数: 3 / 式: PLC |
|---|---|
| 分子量 | 理論値: 622.834 Da |
| Chemical component information |  ChemComp-PLC: |
-分子 #7: DECANE
| 分子 | 名称: DECANE / タイプ: ligand / ID: 7 / コピー数: 9 / 式: D10 |
|---|---|
| 分子量 | 理論値: 142.282 Da |
| Chemical component information |  ChemComp-D10: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.3 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)