+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23637 | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
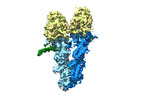
| タイトル | Closed conformation of the Yeast wild-type gamma-TuRC | ||||||||||||||||||||||||||||||||||||||||||
 マップデータ マップデータ | Closed WT gamma-TuRC unmasked unsharpened main map | ||||||||||||||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | microtubule nucleation / CELL CYCLE | ||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報gamma-tubulin complex localization to nuclear side of mitotic spindle pole body / protein localization to mitotic spindle pole body / inner plaque of spindle pole body / microtubule nucleation by spindle pole body / outer plaque of spindle pole body / central plaque of spindle pole body / gamma-tubulin small complex / regulation of microtubule nucleation / karyogamy involved in conjugation with cellular fusion / mitotic spindle elongation ...gamma-tubulin complex localization to nuclear side of mitotic spindle pole body / protein localization to mitotic spindle pole body / inner plaque of spindle pole body / microtubule nucleation by spindle pole body / outer plaque of spindle pole body / central plaque of spindle pole body / gamma-tubulin small complex / regulation of microtubule nucleation / karyogamy involved in conjugation with cellular fusion / mitotic spindle elongation / mitotic spindle pole body / equatorial microtubule organizing center / microtubule minus-end binding / gamma-tubulin complex / positive regulation of microtubule nucleation / meiotic spindle organization / microtubule nucleation / positive regulation of cytoplasmic translation / spindle pole body / gamma-tubulin binding / mitotic sister chromatid segregation / spindle assembly / cytoplasmic microtubule organization / mitotic spindle organization / meiotic cell cycle / structural constituent of cytoskeleton / spindle pole / spindle / mitotic cell cycle / protein-containing complex assembly / microtubule / cytoskeleton / calmodulin binding / protein-containing complex binding / GTP binding / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||||||||||||||
| 生物種 |   | ||||||||||||||||||||||||||||||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.03 Å | ||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Brilot AF / Lyon AS | ||||||||||||||||||||||||||||||||||||||||||
| 資金援助 |  米国, 13件 米国, 13件
| ||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: CM1-driven assembly and activation of yeast γ-tubulin small complex underlies microtubule nucleation. 著者: Axel F Brilot / Andrew S Lyon / Alex Zelter / Shruthi Viswanath / Alison Maxwell / Michael J MacCoss / Eric G Muller / Andrej Sali / Trisha N Davis / David A Agard /  要旨: Microtubule (MT) nucleation is regulated by the γ-tubulin ring complex (γTuRC), conserved from yeast to humans. In , γTuRC is composed of seven identical γ-tubulin small complex (γTuSC) sub- ...Microtubule (MT) nucleation is regulated by the γ-tubulin ring complex (γTuRC), conserved from yeast to humans. In , γTuRC is composed of seven identical γ-tubulin small complex (γTuSC) sub-assemblies, which associate helically to template MT growth. γTuRC assembly provides a key point of regulation for the MT cytoskeleton. Here, we combine crosslinking mass spectrometry, X-ray crystallography, and cryo-EM structures of both monomeric and dimeric γTuSCs, and open and closed helical γTuRC assemblies in complex with Spc110p to elucidate the mechanisms of γTuRC assembly. γTuRC assembly is substantially aided by the evolutionarily conserved CM1 motif in Spc110p spanning a pair of adjacent γTuSCs. By providing the highest resolution and most complete views of any γTuSC assembly, our structures allow phosphorylation sites to be mapped, surprisingly suggesting that they are mostly inhibitory. A comparison of our structures with the CM1 binding site in the human γTuRC structure at the interface between GCP2 and GCP6 allows for the interpretation of significant structural changes arising from CM1 helix binding to metazoan γTuRC. | ||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23637.map.gz emd_23637.map.gz | 318.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23637-v30.xml emd-23637-v30.xml emd-23637.xml emd-23637.xml | 27.8 KB 27.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23637.png emd_23637.png | 118.1 KB | ||
| Filedesc metadata |  emd-23637.cif.gz emd-23637.cif.gz | 8.3 KB | ||
| その他 |  emd_23637_half_map_1.map.gz emd_23637_half_map_1.map.gz emd_23637_half_map_2.map.gz emd_23637_half_map_2.map.gz | 66.8 MB 66.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23637 http://ftp.pdbj.org/pub/emdb/structures/EMD-23637 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23637 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23637 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23637_validation.pdf.gz emd_23637_validation.pdf.gz | 978.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23637_full_validation.pdf.gz emd_23637_full_validation.pdf.gz | 978.3 KB | 表示 | |
| XML形式データ |  emd_23637_validation.xml.gz emd_23637_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_23637_validation.cif.gz emd_23637_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23637 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23637 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23637 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23637 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23637.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23637.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Closed WT gamma-TuRC unmasked unsharpened main map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4183 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
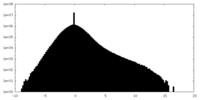
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Closed WT gamma-TuRC half map 1
| ファイル | emd_23637_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Closed WT gamma-TuRC half map 1 | ||||||||||||
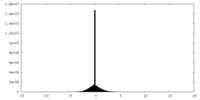
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Closed WT gamma-TuRC half map 2
| ファイル | emd_23637_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Closed WT gamma-TuRC half map 2 | ||||||||||||
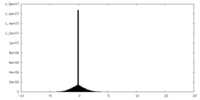
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Closed conformation of the Yeast wild-type gamma-TuRC
| 全体 | 名称: Closed conformation of the Yeast wild-type gamma-TuRC |
|---|---|
| 要素 |
|
-超分子 #1: Closed conformation of the Yeast wild-type gamma-TuRC
| 超分子 | 名称: Closed conformation of the Yeast wild-type gamma-TuRC タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Tubulin gamma chain
| 分子 | 名称: Tubulin gamma chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 52.671188 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGGEIITLQA GQCGNHVGKF LWSQLAKEHA IGTDGLSQLP DSSTERDDDT KPFFRENSRN KFTPRAIMMD SEPSVIADVE NTFRGFFDP RNTWVASDGA SAGNSWANGY DIGTRNQDDI LNKIDKEIDS TDNFEGFQLL HSVAGGTGSG LGSNLLEALC D RYPKKILT ...文字列: MGGEIITLQA GQCGNHVGKF LWSQLAKEHA IGTDGLSQLP DSSTERDDDT KPFFRENSRN KFTPRAIMMD SEPSVIADVE NTFRGFFDP RNTWVASDGA SAGNSWANGY DIGTRNQDDI LNKIDKEIDS TDNFEGFQLL HSVAGGTGSG LGSNLLEALC D RYPKKILT TYSVFPARSS EVVVQSYNTI LALRRLIEDS DATVVFDNAS LLNISGKVFR NPNIDLQHTN QLISTIISSV TN SIRFPSY MYSSMSSIYS TLIPSPELHF LSPSFTPFTS DYIHDDIAHK GHSSYDVMLD LLDPSNSLVS TAMNNPTYFN VYN TIIGNV EPRQISRAMT KLQQRIKFPS WSSSAMHVNI GRRSPYLPLQ PNENEVSGMM LSNMSTVVNV FENACNTFDK VFAK GAFLN NYNVGDLFQS MQNVQDEFAE SREVVQSLME DYVAAEQDSY LDDVLVDDEN MVGELEEDLD ADGDHKLV UniProtKB: Tubulin gamma chain |
-分子 #2: Spindle pole body component SPC98
| 分子 | 名称: Spindle pole body component SPC98 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 98.336211 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MELEPTLFGI IEALAPQLLS QSHLQTFVSD VVNLLRSSTK SATQLGPLID FYKLQSLDSP ETTIMWHKIE KFLDALFGIQ NTDDMVKYL SVFQSLLPSN YRAKIVQKSS GLNMENLANH EHLLSPVRAP SIYTEASFEN MDRFSERRSM VSSPNRYVPS S TYSSVTLR ...文字列: MELEPTLFGI IEALAPQLLS QSHLQTFVSD VVNLLRSSTK SATQLGPLID FYKLQSLDSP ETTIMWHKIE KFLDALFGIQ NTDDMVKYL SVFQSLLPSN YRAKIVQKSS GLNMENLANH EHLLSPVRAP SIYTEASFEN MDRFSERRSM VSSPNRYVPS S TYSSVTLR QLSNPYYVNT IPEEDILKYV SYTLLATTSA LFPFDHEQIQ IPSKIPNFES GLLHLIFEAG LLYQSLGYKV EK FRMLNIS PMKKALIIEI SEELQNYTAF VNNLVSSGTV VSLKSLYREI YENIIRLRIY CRFTEHLEEL SGDTFLIELN IFK SHGDLT IRKIATNLFN SMISLYYEYL MNWLTKGLLR ATYGEFFIAE NTDTNGTDDD FIYHIPIEFN QERVPAFIPK ELAY KIFMI GKSYIFLEKY CKEVQWTNEF SKKYHVLYQS NSYRGISTNF FEIINDQYSE IVNHTNQILN QKFHYRDVVF ALKNI LLMG KSDFMDALIE KANDILATPS DSLPNYKLTR VLQEAVQLSS LRHLMNSPRN SSVINGLDAR VLDLGHGSVG WDVFTL DYI LYPPLSLVLN VNRPFGRKEY LRIFNFLWRF KKNNYFYQKE MLKSNDIIRS FKKIRGYNPL IRDIINKLSR ISILRTQ FQ QFNSKMESYY LNCIIEENFK EMTRKLQRTE NKSQNQFDLI RLNNGTIELN GILTPKAEVL TKSSSSKPQK HAIEKTLN I DELESVHNTF LTNILSHKLF ATNTSEISVG DYSGQPYPTS LVLLLNSVYE FVKVYCNLND IGYEIFIKMN LNDHEASNG LLGKFNTNLK EIVSQYKNFK DRLYIFRADL KNDGDEELFL LSKSLR UniProtKB: Spindle pole body component SPC98 |
-分子 #3: Spindle pole body component SPC97
| 分子 | 名称: Spindle pole body component SPC97 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 96.940594 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEIKEVDDRA ELLRYTNNIP LLGKLVNHQP LWSTNPKLKS FSLEKISAPD QRRVQEALVV KDLLNVLIGL EGTYIRYFND YEPSDPETP IEFKIAKKMD PSFKTFSRRI VRYGKQYMIL TRAYEKWSDT SFGMVLQRFA YEIRRFLEDV YLKTLVERLE R DFNKVPNF ...文字列: MEIKEVDDRA ELLRYTNNIP LLGKLVNHQP LWSTNPKLKS FSLEKISAPD QRRVQEALVV KDLLNVLIGL EGTYIRYFND YEPSDPETP IEFKIAKKMD PSFKTFSRRI VRYGKQYMIL TRAYEKWSDT SFGMVLQRFA YEIRRFLEDV YLKTLVERLE R DFNKVPNF SIRELEQIIN ETEVNKQMEL LYNIYEEIFR EIEERRTNQS SQEDFNNFMD SMKNESSLHL RLMVAFDTTV YP VPKGGAI LKIFQQKILE NLGDRSSVMF LKKLLNNISQ DYCTMLYEWL TQGILNDPYQ EFMTYDDLEG KTDNIFDTRD RAW DTQYFI RKDVLLRDCD SEEDKNLLFK MLRTGILLKV VRASLQIPTI PSNSSDITIQ EINDFADLME GSNLELYVDK CYSR ANEIF LKLFFQGYDL INVLKHLQQI FLGYQSGHNV LKFLTKNMGE LTKHYRNDNN ANYDKLLQNF ELERQSENPN NLMRQ LLMI QFDTETLPQV LSHYLQIYPE VPENNSANDD SDPLMHANNF KNMNAILFDE LSKERTGAYH GSNLELYTPK SAIYHL KFD INIPYPLNII ISRTCMIKYQ IILRYQLVLQ YHSRLLDETW MDLNKTPSWK YRGYSHTVKR RIVRATRVLH AKMNHFI KT IMEYFNQNVI DKEVYSLEKC YRNPTLAVAI QNELEGGLTN IMTNRCLSDL IPLQLQIFDI VYKFCKFIKS MRAKLCQL D PVLYEKHKSG MMKTLNEGYR TNNGGQEDVG YQEDAALELI QKLIEYISNA SSIFRKCLIN FTQELSTEKF DFYDSSSVD AAGIERVLYS IVPPRSASAS SQR UniProtKB: Spindle pole body component SPC97 |
-分子 #4: Spindle pole body component 110
| 分子 | 名称: Spindle pole body component 110 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 25.449555 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDEASHLPNG SLKNMEFTPV GFIKSKRNTT QTQVVSPTKV PNANNGDENE GPVKKRQRRS IDDTIDSTRL FSEASQFDDS FPEIKANIP PSPRSGNVDK SRKRNLIDDL KKDVPMSQPL KEQEVREHQM KKERFDRALE SKLLGKRHIT YANSDISNKE L YINEIKSL ...文字列: MDEASHLPNG SLKNMEFTPV GFIKSKRNTT QTQVVSPTKV PNANNGDENE GPVKKRQRRS IDDTIDSTRL FSEASQFDDS FPEIKANIP PSPRSGNVDK SRKRNLIDDL KKDVPMSQPL KEQEVREHQM KKERFDRALE SKLLGKRHIT YANSDISNKE L YINEIKSL KHEIKELRKE KNDTLNNYDT LEEETDDLKN RLQALEKELD AKNKIVNSRK VD UniProtKB: Spindle pole body component 110 |
-分子 #5: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: Whatman #1 Filter papers used.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 平均露光時間: 15.0 sec. / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 47214 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 0.01 Å 想定した対称性 - らせんパラメータ - ΔΦ: 0 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 4.03 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM (ver. 1.0 Beta) / 使用した粒子像数: 20420 |
|---|---|
| Segment selection | 選択した数: 28753 詳細: Boxed approximately every 3 asymmetric units using a rise of 21 Angstroms, with a box size of 600 physical pixels (635.4A). |
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: cisTEM (ver. 1.0 Beta) / 詳細: Projection matching in cisTEM |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 130 |
|---|---|
| 得られたモデル |  PDB-7m2y: |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X