+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21901 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Burkholderia pseudomallei hopanoid biosynthesis-associated RND transporter HpnN | |||||||||
 マップデータ マップデータ | density modification in Phenix | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CryoEM structure / hopanoid transporter HpnN / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 | Hopanoid biosynthesis associated RND transporter-like protein HpnN / Membrane transport protein MMPL domain / MMPL family / Sterol-sensing domain (SSD) profile. / Sterol-sensing domain / membrane => GO:0016020 / plasma membrane / Hopanoid biosynthesis associated RND transporter like protein HpnN 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Burkholderia pseudomallei MSHR346 (類鼻疽菌) Burkholderia pseudomallei MSHR346 (類鼻疽菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.59 Å | |||||||||
 データ登録者 データ登録者 | Lyu M / Yu EW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2021 ジャーナル: Nat Methods / 年: 2021タイトル: A 'Build and Retrieve' methodology to simultaneously solve cryo-EM structures of membrane proteins. 著者: Chih-Chia Su / Meinan Lyu / Christopher E Morgan / Jani Reddy Bolla / Carol V Robinson / Edward W Yu /   要旨: Single-particle cryo-electron microscopy (cryo-EM) has become a powerful technique in the field of structural biology. However, the inability to reliably produce pure, homogeneous membrane protein ...Single-particle cryo-electron microscopy (cryo-EM) has become a powerful technique in the field of structural biology. However, the inability to reliably produce pure, homogeneous membrane protein samples hampers the progress of their structural determination. Here, we develop a bottom-up iterative method, Build and Retrieve (BaR), that enables the identification and determination of cryo-EM structures of a variety of inner and outer membrane proteins, including membrane protein complexes of different sizes and dimensions, from a heterogeneous, impure protein sample. We also use the BaR methodology to elucidate structural information from Escherichia coli K12 crude membrane and raw lysate. The findings demonstrate that it is possible to solve high-resolution structures of a number of relatively small (<100 kDa) and less abundant (<10%) unidentified membrane proteins within a single, heterogeneous sample. Importantly, these results highlight the potential of cryo-EM for systems structural proteomics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21901.map.gz emd_21901.map.gz | 2.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21901-v30.xml emd-21901-v30.xml emd-21901.xml emd-21901.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21901.png emd_21901.png | 106.3 KB | ||
| Filedesc metadata |  emd-21901.cif.gz emd-21901.cif.gz | 5.6 KB | ||
| その他 |  emd_21901_additional_1.map.gz emd_21901_additional_1.map.gz | 97.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21901 http://ftp.pdbj.org/pub/emdb/structures/EMD-21901 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21901 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21901 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21901_validation.pdf.gz emd_21901_validation.pdf.gz | 404.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21901_full_validation.pdf.gz emd_21901_full_validation.pdf.gz | 404.5 KB | 表示 | |
| XML形式データ |  emd_21901_validation.xml.gz emd_21901_validation.xml.gz | 4.2 KB | 表示 | |
| CIF形式データ |  emd_21901_validation.cif.gz emd_21901_validation.cif.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21901 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21901 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21901 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21901 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21901.map.gz / 形式: CCP4 / 大きさ: 2.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21901.map.gz / 形式: CCP4 / 大きさ: 2.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density modification in Phenix | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: orignal in cryosparc
| ファイル | emd_21901_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | orignal in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
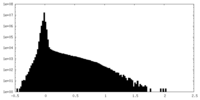
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : hopanoid transporter HpnN
| 全体 | 名称: hopanoid transporter HpnN |
|---|---|
| 要素 |
|
-超分子 #1: hopanoid transporter HpnN
| 超分子 | 名称: hopanoid transporter HpnN / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Hopanoid biosynthesis associated RND transporter like protein HpnN
| 分子 | 名称: Hopanoid biosynthesis associated RND transporter like protein HpnN タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Burkholderia pseudomallei MSHR346 (類鼻疽菌) Burkholderia pseudomallei MSHR346 (類鼻疽菌) |
| 分子量 | 理論値: 92.60093 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLTSVLVRLV AWSVRRPIWV VVLSLVIAAL SSVYVAHHFK INTDISKLVE NDPKWAALGR AIDDAFPQRN QTILAVVEAP APEFAGAAA DALAEGLRRE TEAGRIGQVS EPAGGPLFEH DGLLFLPEQD VATTTAQLAS ARPLINVLAK DPSIAGLATT L STTLGVPL ...文字列: MLTSVLVRLV AWSVRRPIWV VVLSLVIAAL SSVYVAHHFK INTDISKLVE NDPKWAALGR AIDDAFPQRN QTILAVVEAP APEFAGAAA DALAEGLRRE TEAGRIGQVS EPAGGPLFEH DGLLFLPEQD VATTTAQLAS ARPLINVLAK DPSIAGLATT L STTLGVPL QSGQVKLSGM AKLLSRSAAT VDDVLAGKPA AFSWRALVDA DAAREPARAF VTVQPVVNYG ALKAGEQASR TI RATAQAL KLDERFGAAV RLTGEQPLAD EEFASVQDGA LVNGIATLAI VLVILWIALR SKRMIASVFV TLFVGLVVTA ALG LMMVGS LNMISVAFMV LFVGLGVDFA IQYGVKYREE RHRDPNLDHA LVGAAHAMGM PLTLATAAVA ASFFSFLPTA YRGV SELGL IAGVGMFVAL FTTLTLLPAL LKLLAPPGER KPPGFPRLAP VDDYLDHHRK PILIGTLAVV IGALPLLAHL RFDFN PLHL KDPRSESMAT LLALKDSPEA SVNDVSLLAP SLVAANAAAQ RLGALPEVGR TTTLSTFIPD AQPQKLATIA AAARGL LPA LTQPAAAPVP DAQRVAALKR ASNLLEYASE DYPGPGAAAA KHLSESLAKL AAADAATRER AEHAFSVPLK IALNQLA ML LQPLEITREN LPPQIVRDWI APDGRALVQI SPKVVKGADP GDDAMLRRFA KAVKAAEPGA IGGPISILHS ADTIIRAF L QAAALSVVSI TVLLWITLRR FGDVLRTLVP LLVSGVVTLE LCVLLGMPLN FANIIALPLM LGVGVAFKVY FVMAWRAGQ TGLLQSSLTH AVLFSAATTA TAFGSLWLSH HPGTASMGRL LALALSCTLI GAVVFQPVLM GKPRTKRVTN QSQGIDE UniProtKB: Hopanoid biosynthesis associated RND transporter like protein HpnN |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.59 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 63910 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー





























 Z
Z Y
Y X
X