+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21702 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Empty phage G cryoEM capsid structure at 9 Angstrom resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Bacillus virus G (ウイルス) Bacillus virus G (ウイルス) | |||||||||
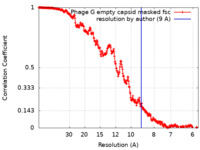
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.0 Å | |||||||||
 データ登録者 データ登録者 | Gonzalez B / Monroe L / Kunpeng L / Yan R / Wright E / Walter T / Kihara D / Weintraub SE / Julie AT / Philip S / Jiang W | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2020 ジャーナル: J Mol Biol / 年: 2020タイトル: Phage G Structure at 6.1 Å Resolution, Condensed DNA, and Host Identity Revision to a Lysinibacillus. 著者: Brenda González / Lyman Monroe / Kunpeng Li / Rui Yan / Elena Wright / Thomas Walter / Daisuke Kihara / Susan T Weintraub / Julie A Thomas / Philip Serwer / Wen Jiang /  要旨: Phage G has the largest capsid and genome of any known propagated phage. Many aspects of its structure, assembly, and replication have not been elucidated. Herein, we present the dsDNA-packed and ...Phage G has the largest capsid and genome of any known propagated phage. Many aspects of its structure, assembly, and replication have not been elucidated. Herein, we present the dsDNA-packed and empty phage G capsid at 6.1 and 9 Å resolution, respectively, using cryo-EM for structure determination and mass spectrometry for protein identification. The major capsid protein, gp27, is identified and found to share the HK97-fold universally conserved in all previously solved dsDNA phages. Trimers of the decoration protein, gp26, sit on the 3-fold axes and are thought to enhance the interactions of the hexameric capsomeres of gp27, for other phages encoding decoration proteins. Phage G's decoration protein is longer than what has been reported in other phages, and we suspect the extra interaction surface area helps stabilize the capsid. We identified several additional capsid proteins, including a candidate for the prohead protease responsible for processing gp27. Furthermore, cryo-EM reveals a range of partially full, condensed DNA densities that appear to have no contact with capsid shell. Three analyses confirm that the phage G host is a Lysinibacillus, and not Bacillus megaterium: identity of host proteins in our mass spectrometry analyses, genome sequence of the phage G host, and host range of phage G. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21702.map.gz emd_21702.map.gz | 1.5 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21702-v30.xml emd-21702-v30.xml emd-21702.xml emd-21702.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21702_fsc.xml emd_21702_fsc.xml | 27.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21702.png emd_21702.png | 243.1 KB | ||
| その他 |  emd_21702_half_map_1.map.gz emd_21702_half_map_1.map.gz emd_21702_half_map_2.map.gz emd_21702_half_map_2.map.gz | 618.8 MB 618.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21702 http://ftp.pdbj.org/pub/emdb/structures/EMD-21702 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21702 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21702 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21702_validation.pdf.gz emd_21702_validation.pdf.gz | 78.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21702_full_validation.pdf.gz emd_21702_full_validation.pdf.gz | 78 KB | 表示 | |
| XML形式データ |  emd_21702_validation.xml.gz emd_21702_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21702 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21702 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21702 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21702 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21702.map.gz / 形式: CCP4 / 大きさ: 1.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21702.map.gz / 形式: CCP4 / 大きさ: 1.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.9 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
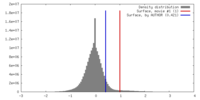
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_21702_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
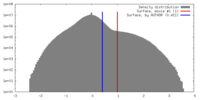
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_21702_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacillus virus G
| 全体 | 名称:  Bacillus virus G (ウイルス) Bacillus virus G (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Bacillus virus G
| 超分子 | 名称: Bacillus virus G / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 1084719 / 生物種: Bacillus virus G / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| ウイルス殻 | Shell ID: 1 / 直径: 1600.0 Å / T番号(三角分割数): 52 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: 0.01 M Tris-Cl (pH 7.4), 0.01 M MgSO4, 6% polyethylene glycol MW 3350 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 65 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 詳細: Three microliters of phage G sample was deposited on a 400 mesh Ted Pella ultrathin lacey carbon grid and incubated for 30 minutes in a humid chamber on ice. The grid was then washed with 10 ...詳細: Three microliters of phage G sample was deposited on a 400 mesh Ted Pella ultrathin lacey carbon grid and incubated for 30 minutes in a humid chamber on ice. The grid was then washed with 10 microliters of 0.2 TM buffer. Using a Gatan CP3 plunger, the grid was then blotted for 9 seconds with Whatman 1 filter paper at 65% humidity, then plunge-frozen in liquid ethane. The plunge-frozen, phage G grid was then imaged using a Titan Krios equipped with a K2 detector in super-resolution mode at the Purdue Cryo-EM Facility with a nominal magnification of 8,700 resulting in 1.742 Angstroms per pixel. A total of 375 movies was collected.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 375 / 平均電子線量: 14.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 8700 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)