+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20693 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Adeno-associated virus strain AAVhu.37 capsid icosahedral structure | |||||||||
 マップデータ マップデータ | icosahedrally-averaged map of AAVhu.37 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | capsid / blood-brain barrier penetration / VIRUS | |||||||||
| 機能・相同性 | Phospholipase A2-like domain / Phospholipase A2-like domain / Parvovirus coat protein VP2 / Parvovirus coat protein VP1/VP2 / Parvovirus coat protein VP2 / Capsid/spike protein, ssDNA virus / T=1 icosahedral viral capsid / structural molecule activity / Capsid protein VP1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) | |||||||||
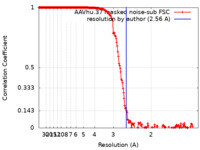
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.56 Å | |||||||||
 データ登録者 データ登録者 | Kaelber JT / Yost SA | |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr F Struct Biol Commun / 年: 2020 ジャーナル: Acta Crystallogr F Struct Biol Commun / 年: 2020タイトル: Structure of the AAVhu.37 capsid by cryoelectron microscopy. 著者: Jason T Kaelber / Samantha A Yost / Keith A Webber / Emre Firlar / Ye Liu / Olivier Danos / Andrew C Mercer /  要旨: Adeno-associated viruses (AAVs) are used as in vivo gene-delivery vectors in gene-therapy products and have been heavily investigated for numerous indications. Over 100 naturally occurring AAV ...Adeno-associated viruses (AAVs) are used as in vivo gene-delivery vectors in gene-therapy products and have been heavily investigated for numerous indications. Over 100 naturally occurring AAV serotypes and variants have been isolated from primate samples. Many reports have described unique properties of these variants (for instance, differences in potency, target cell or evasion of the immune response), despite high amino-acid sequence conservation. AAVhu.37 is of interest for clinical applications owing to its proficient transduction of the liver and central nervous system. The sequence identity of the AAVhu.37 VP1 to the well characterized AAVrh.10 serotype, for which no structure is available, is greater than 98%. Here, the structure of the AAVhu.37 capsid at 2.56 Å resolution obtained via single-particle cryo-electron microscopy is presented. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20693.map.gz emd_20693.map.gz | 609.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20693-v30.xml emd-20693-v30.xml emd-20693.xml emd-20693.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20693_fsc.xml emd_20693_fsc.xml | 21.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20693.png emd_20693.png | 229 KB | ||
| Filedesc metadata |  emd-20693.cif.gz emd-20693.cif.gz | 6.6 KB | ||
| その他 |  emd_20693_half_map_1.map.gz emd_20693_half_map_1.map.gz emd_20693_half_map_2.map.gz emd_20693_half_map_2.map.gz | 615.8 MB 615.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20693 http://ftp.pdbj.org/pub/emdb/structures/EMD-20693 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20693 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20693 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20693_validation.pdf.gz emd_20693_validation.pdf.gz | 984.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20693_full_validation.pdf.gz emd_20693_full_validation.pdf.gz | 983.8 KB | 表示 | |
| XML形式データ |  emd_20693_validation.xml.gz emd_20693_validation.xml.gz | 28.3 KB | 表示 | |
| CIF形式データ |  emd_20693_validation.cif.gz emd_20693_validation.cif.gz | 37.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20693 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20693 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20693 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20693 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20693.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20693.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | icosahedrally-averaged map of AAVhu.37 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.70065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half A
| ファイル | emd_20693_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
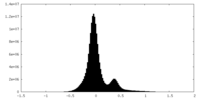
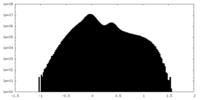
| 注釈 | half A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half B
| ファイル | emd_20693_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
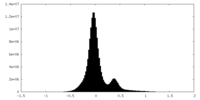
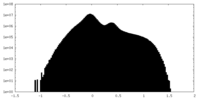
| 注釈 | half B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus
| 全体 | 名称:   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Adeno-associated virus
| 超分子 | 名称: Adeno-associated virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Virions containing GFP-encoding ssDNA were assembled in transfected HEK293T helper cells. NCBI-ID: 272636 / 生物種: Adeno-associated virus / Sci species strain: hu.37 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 250.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) |
| 分子量 | 理論値: 81.528852 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AADGYLPDWL EDNLSEGIRE WWDLKPGAPK PKANQQKQDD GRGLVLPGYK YLGPFNGLDK GEPVNAADAA ALEHDKAYDQ QLKAGDNPY LRYNHADAEF QERLQEDTSF GGNLGRAVFQ AKKRVLEPLG LVEEAAKTAP GKKRPVEPSP QRSPDSSTGI G KKGQQPAK ...文字列: AADGYLPDWL EDNLSEGIRE WWDLKPGAPK PKANQQKQDD GRGLVLPGYK YLGPFNGLDK GEPVNAADAA ALEHDKAYDQ QLKAGDNPY LRYNHADAEF QERLQEDTSF GGNLGRAVFQ AKKRVLEPLG LVEEAAKTAP GKKRPVEPSP QRSPDSSTGI G KKGQQPAK KRLNFGQTGD SESVPDPQPI GEPPAGPSGL GSGTMAAGGG APMADNNEGA DGVGSSSGNW HCDSTWLGDR VI TTSTRTW ALPTYNNHLY KQISNGTSGG STNDNTYFGY STPWGYFDFN RFHCHFSPRD WQRLINNNWG FRPKRLSFKL FNI QVKEVT QNEGTKTIAN NLTSTIQVFT DSEYQLPYVL GSAHQGCLPP FPADVFMIPQ YGYLTLNNGS QAVGRSSFYC LEYF PSQML RTGNNFEFSY TFEDVPFHSS YAHSQSLDRL MNPLIDQYLY YLSRTQSTGG TQGTQQLLFS QAGPANMSAQ AKNWL PGPC YRQQRVSTTL SQNNNSNFAW TGATKYHLNG RDSLVNPGVA MATHKDDEER FFPSSGVLMF GKQGAGRDNV DYSSVM LTS EEEIKTTNPV ATEQYGVVAD NLQQTNTGPI VGNVNSQGAL PGMVWQNRDV YLQGPIWAKI PHTDGNFHPS PLMGGFG LK HPPPQILIKN TPVPADPPTT FSQAKLASFI TQYSTGQVSV EIEWELQKEN SKRWNPEIQY TSNYYKSTNV DFAVNTEG T YSEPRPIGTR YLTRNL UniProtKB: Capsid protein VP1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
詳細: PBS | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2.86 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.037 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: LEICA EM GP 詳細: Whatman #1 filter paper, 4.5s blot. 3.5 microliters specimen applied.. | ||||||||||||
| 詳細 | approx. 7*10^13 genome copies per mL |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | 色収差補正装置: none / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-25 / 撮影したグリッド数: 1 / 平均露光時間: 5.0 sec. / 平均電子線量: 1.31 e/Å2 / 詳細: collected in superresolution mode |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 1.584 µm / 最小 デフォーカス(補正後): 0.535 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X