+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20273 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
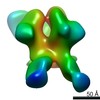
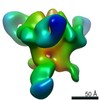
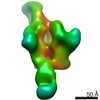
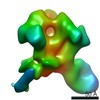
| タイトル | Negative-stain reconstruction of HIV-1 Env BG505 NFL CC+ trimer in complex with rabbit antibody E70 fragment antigen binding | |||||||||||||||
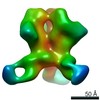
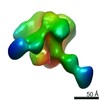
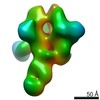
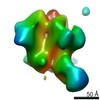
 マップデータ マップデータ | negative-stain 3D reconstruction | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  | |||||||||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 21.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Torres JL / Ozorowski G / Andrew AB | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2019 ジャーナル: Immunity / 年: 2019タイトル: Vaccination with Glycan-Modified HIV NFL Envelope Trimer-Liposomes Elicits Broadly Neutralizing Antibodies to Multiple Sites of Vulnerability. 著者: Viktoriya Dubrovskaya / Karen Tran / Gabriel Ozorowski / Javier Guenaga / Richard Wilson / Shridhar Bale / Christopher A Cottrell / Hannah L Turner / Gemma Seabright / Sijy O'Dell / Jonathan ...著者: Viktoriya Dubrovskaya / Karen Tran / Gabriel Ozorowski / Javier Guenaga / Richard Wilson / Shridhar Bale / Christopher A Cottrell / Hannah L Turner / Gemma Seabright / Sijy O'Dell / Jonathan L Torres / Lifei Yang / Yu Feng / Daniel P Leaman / Néstor Vázquez Bernat / Tyler Liban / Mark Louder / Krisha McKee / Robert T Bailer / Arlette Movsesyan / Nicole A Doria-Rose / Marie Pancera / Gunilla B Karlsson Hedestam / Michael B Zwick / Max Crispin / John R Mascola / Andrew B Ward / Richard T Wyatt /    要旨: The elicitation of broadly neutralizing antibodies (bNAbs) against the HIV-1 envelope glycoprotein (Env) trimer remains a major vaccine challenge. Most cross-conserved protein determinants are ...The elicitation of broadly neutralizing antibodies (bNAbs) against the HIV-1 envelope glycoprotein (Env) trimer remains a major vaccine challenge. Most cross-conserved protein determinants are occluded by self-N-glycan shielding, limiting B cell recognition of the underlying polypeptide surface. The exceptions to the contiguous glycan shield include the conserved receptor CD4 binding site (CD4bs) and glycoprotein (gp)41 elements proximal to the furin cleavage site. Accordingly, we performed heterologous trimer-liposome prime:boosting in rabbits to drive B cells specific for cross-conserved sites. To preferentially expose the CD4bs to B cells, we eliminated proximal N-glycans while maintaining the native-like state of the cleavage-independent NFL trimers, followed by gradual N-glycan restoration coupled with heterologous boosting. This approach successfully elicited CD4bs-directed, cross-neutralizing Abs, including one targeting a unique glycan-protein epitope and a bNAb (87% breadth) directed to the gp120:gp41 interface, both resolved by high-resolution cryoelectron microscopy. This study provides proof-of-principle immunogenicity toward eliciting bNAbs by vaccination. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20273.map.gz emd_20273.map.gz | 11.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20273-v30.xml emd-20273-v30.xml emd-20273.xml emd-20273.xml | 21.3 KB 21.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20273.png emd_20273.png | 54.2 KB | ||
| その他 |  emd_20273_half_map_1.map.gz emd_20273_half_map_1.map.gz emd_20273_half_map_2.map.gz emd_20273_half_map_2.map.gz | 11.9 MB 11.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20273 http://ftp.pdbj.org/pub/emdb/structures/EMD-20273 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20273 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20273 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20273_validation.pdf.gz emd_20273_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20273_full_validation.pdf.gz emd_20273_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_20273_validation.xml.gz emd_20273_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20273 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20273 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20273 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20273 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20273.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20273.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | negative-stain 3D reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half map 1
| ファイル | emd_20273_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_20273_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Env BG505 NFL CC+ trimer in complex with rabbit antibody E7...
| 全体 | 名称: HIV-1 Env BG505 NFL CC+ trimer in complex with rabbit antibody E70 fragment antigen binding |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 Env BG505 NFL CC+ trimer in complex with rabbit antibody E7...
| 超分子 | 名称: HIV-1 Env BG505 NFL CC+ trimer in complex with rabbit antibody E70 fragment antigen binding タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 570 KDa |
-分子 #1: HIV-1 Env BG505 NFL CC+ trimer
| 分子 | 名称: HIV-1 Env BG505 NFL CC+ trimer / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK DAETTLFCAS DAKAYETEKH NVWATHACVP TDPNPQEIHL ENVTEEFNMW KNNMVEQMHT DIISLWDQSL KPCVKLTPLC VTLQCTNVTN NITDDMRGEL KNCSFNMTTE LRDKKQKVYS LFYRLDVVQI ...文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK DAETTLFCAS DAKAYETEKH NVWATHACVP TDPNPQEIHL ENVTEEFNMW KNNMVEQMHT DIISLWDQSL KPCVKLTPLC VTLQCTNVTN NITDDMRGEL KNCSFNMTTE LRDKKQKVYS LFYRLDVVQI NENQGNRSNN SNKEYRLINC NTSACTQACP KVSFEPIPIH YCAPAGFAIL KCKDKKFNGT GPCPSVSTVQ CTHGIKPVVS TQLLLNGSLA EEEVMIRSEN ITNNAKNILV QFNTPVQINC TRPNNYTRKS IRIGPGQAFY ATGDIIGDIR QAHCNVSKAT WNETLGKVVK QLRKHFGNNT IIRFANSSGG DLEVTTHSFN CGGEFFYCNT SGLFNSTWIS NTSVQGSNST GSNDSITLPC RIKQIINMWQ RIGQCMYAPP IQGVIRCVSN ITGLILTRDG GSTNSTTETF RPGGGDMRDN WRSELYKYKV VKIEPLGVAP TRAKRRVVGG GGGSGGGGSA VGIGAVRRGF LGAAGSTMGA ASMTLTVQAR NLLSGIVQQQ SNLLRAPEAQ QHLLKLTVWG IKQLQARVLA VERYLRDQQL LGIWGCSGKL ICTTNVPWNS SWSNRNLSEI WDNMTWLQWD KEISNYTQII YGLLEESQNQ QEKNEQDLLA LDGGGGSHHH HHHHH |
-分子 #2: Rabbit antibody E70 fragment antigen binding heavy chain
| 分子 | 名称: Rabbit antibody E70 fragment antigen binding heavy chain タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSLEESGGGL VKPGGTLTLT CKASGIDFTS GYDMCWVRQA PGKGLEWVAC IYLGDGNTYY ASWAKGQFTI SKTSSTTVTL QMTSLTAADT ATYFCARFAG YRYSVWSYPD LWGPGTLVTV SSGQPKAPSV FPLAPCCGDT PSSTVTLGCL VKGYLPEPVT VTWNSGTLTN ...文字列: QSLEESGGGL VKPGGTLTLT CKASGIDFTS GYDMCWVRQA PGKGLEWVAC IYLGDGNTYY ASWAKGQFTI SKTSSTTVTL QMTSLTAADT ATYFCARFAG YRYSVWSYPD LWGPGTLVTV SSGQPKAPSV FPLAPCCGDT PSSTVTLGCL VKGYLPEPVT VTWNSGTLTN GVRTFPSVRQ SSGLYSLSSV VSVTSSSQPV TCNVAHPATN TKVDKTVAPS TCSK |
-分子 #3: Rabbit antibody E70 fragment antigen binding kappa chain
| 分子 | 名称: Rabbit antibody E70 fragment antigen binding kappa chain タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIVMTQTPAS VEADVGGTVT IKCQASQRIV NLVAWYQHKP GQPPKLLIIG ASDLASGVPS RFSGSGYGTE FTLTISDLEC ADAATYFCQS AYNGDGDNAF GGGTEVVVKG DPVAPSVLIF PPAADQVATG TVTIVCVANK YFPDVTVTWE VDGTTQTTGI ENSKTPQNSA ...文字列: DIVMTQTPAS VEADVGGTVT IKCQASQRIV NLVAWYQHKP GQPPKLLIIG ASDLASGVPS RFSGSGYGTE FTLTISDLEC ADAATYFCQS AYNGDGDNAF GGGTEVVVKG DPVAPSVLIF PPAADQVATG TVTIVCVANK YFPDVTVTWE VDGTTQTTGI ENSKTPQNSA DCTYNLSSTL TLTSTQYNSH KEYTCKVTQG TTSVVQSFNR GDC |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: Grids were negatively stained with 2% (w/v) uranyl formate for 50 s | ||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)