+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12155 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KBV empty particle missing pentamer at acidic pH (C1) | |||||||||
 マップデータ マップデータ | KBV empty particle missing pentamer at acidic pH (C1) | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Apis mellifera (セイヨウミツバチ) / Apis mellifera (セイヨウミツバチ) /   Kashmir bee virus (腐蛆病) Kashmir bee virus (腐蛆病) | |||||||||
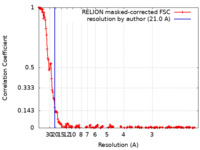
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 21.0 Å クライオ電子顕微鏡法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Mukhamedova L / Plevka P | |||||||||
| 資金援助 |  チェコ, 2件 チェコ, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2021 ジャーナル: J Virol / 年: 2021タイトル: Virion structure and genome release mechanism of dicistrovirus Kashmir bee virus. 著者: Liya Mukhamedova / Tibor Füzik / Jiří Nováček / Dominik Hrebík / Antonín Přidal / Gerardo A Marti / Diego M A Guérin / Pavel Plevka /    要旨: Infections of Kashmir bee virus (KBV) are lethal for honeybees and have been associated with colony collapse disorder. KBV and closely related viruses contribute to the ongoing decline in the number ...Infections of Kashmir bee virus (KBV) are lethal for honeybees and have been associated with colony collapse disorder. KBV and closely related viruses contribute to the ongoing decline in the number of honeybee colonies in North America, Europe, Australia, and other parts of the world. Despite the economic and ecological impact of KBV, its structure and infection process remain unknown. Here we present the structure of the virion of KBV determined to a resolution of 2.8 Å. We show that the exposure of KBV to acidic pH induces a reduction in inter-pentamer contacts within capsids and the reorganization of its RNA genome from a uniform distribution to regions of high and low density. Capsids of KBV crack into pieces at acidic pH, resulting in the formation of open particles lacking pentamers of capsid proteins. The large openings of capsids enable the rapid release of genomes and thus limit the probability of their degradation by RNases. The opening of capsids may be a shared mechanism for the genome release of viruses from the family The western honeybee () is indispensable for maintaining agricultural productivity as well as the abundance and diversity of wild flowering plants. However, bees suffer from environmental pollution, parasites, and pathogens, including viruses. Outbreaks of virus infections cause the deaths of individual honeybees as well as collapses of whole colonies. Kashmir bee virus has been associated with colony collapse disorder in the US, and no cure of the disease is currently available. Here we report the structure of an infectious particle of Kashmir bee virus and show how its protein capsid opens to release the genome. Our structural characterization of the infection process determined that therapeutic compounds stabilizing contacts between pentamers of capsid proteins could prevent the genome release of the virus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12155.map.gz emd_12155.map.gz | 74.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12155-v30.xml emd-12155-v30.xml emd-12155.xml emd-12155.xml | 14.4 KB 14.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12155_fsc.xml emd_12155_fsc.xml | 18.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12155.png emd_12155.png | 144.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12155 http://ftp.pdbj.org/pub/emdb/structures/EMD-12155 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12155 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12155 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12155.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12155.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | KBV empty particle missing pentamer at acidic pH (C1) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Kashmir bee virus
| 全体 | 名称:   Kashmir bee virus (腐蛆病) Kashmir bee virus (腐蛆病) |
|---|---|
| 要素 |
|
-超分子 #1: Kashmir bee virus
| 超分子 | 名称: Kashmir bee virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 68876 / 生物種: Kashmir bee virus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Apis mellifera (セイヨウミツバチ) Apis mellifera (セイヨウミツバチ) |
| 分子量 | 理論値: 5.932 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Full virus / 直径: 350.0 Å / T番号(三角分割数): 3 |
-分子 #1: Icosahedral virus
| 分子 | 名称: Icosahedral virus / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Apis mellifera (セイヨウミツバチ) Apis mellifera (セイヨウミツバチ) |
| 配列 | 文字列: NFKYLRCDVQ VKIVLNANPF IAGRLYLAYS PYDDKVAPER RIIYTSRAGV TGYPGVELDF QLDNSVEMTI PYASFQEAY DLVSGTEDFV QLYLFTITPV LGPSAESANS KVDLSVYMWL DNISLVIPTY RLNPNLPTGQ T LTRIVQNS DSDKLKEALK IAKSKNPSGY ...文字列: NFKYLRCDVQ VKIVLNANPF IAGRLYLAYS PYDDKVAPER RIIYTSRAGV TGYPGVELDF QLDNSVEMTI PYASFQEAY DLVSGTEDFV QLYLFTITPV LGPSAESANS KVDLSVYMWL DNISLVIPTY RLNPNLPTGQ T LTRIVQNS DSDKLKEALK IAKSKNPSGY KYIMGVLEQY NPSVKQVSMQ IATPNKSKST KPTSENPKIG PI SEVASGV KTAANGIERI PVLGEIAKPV TAAVKWFADI VGGVAAIFGW SKPRNQNQVM PYQNVPGWGY SLY KGIDMS VPLAYDPNNE LGDLRDVFPS AVDEMAIGYV CGNPAIKHVL TWNTTDAVQN PISNGDDWGG VIPV GMPCY SKTIRAVKGD TSTSKTEVMD PAPCEYVANL FSYWRATMCY RITVVKTAFH TGRLEIFFEP GSIPT VRTA DNLGPDQTQL NGTVAPSDNN YKYILDLTND TEVTIKVPYV SNKMFMKTVG IYGANDENNW DFDESF TGF LCIRPITKLM APDTVSQKVS IVVWKWAEDV VVVEPKPLTS GPTQVYNPPA VARDQVKQID VSMQINL SN KTDENTISFF DSGDPERMNS EALMRGCGEQ IVNLRPLLRT FRTINDNWSL AANTKTPITD LTNTADAE G RDYMSYLSFL YRFYRGGRRY KFFNTTPLKQ SQTCYVRSFL IPRNYTADEI NTDGPSHITY PVINPVHEV EVPFYSQYRK IPIASTTDKG YDSSLMYYTN VGTQQIVARA GNDDFTFGWM IGTPQLQGIT KEVAN |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | tissue |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 6 |
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): -0.004 µm / 最小 デフォーカス(補正後): -0.0002 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): -0.0018000000000000002 µm 最小 デフォーカス(公称値): -0.001 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 103.15 K / 最高: 108.15 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 8193 / 平均露光時間: 1.0 sec. / 平均電子線量: 94.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 27.78 / 当てはまり具合の基準: Cross- correlation |
 ムービー
ムービー コントローラー
コントローラー