+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11268 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E2 core of the fungal Pyruvate dehydrogenase complex with flexible/oversym. periphery, structured core and S4Y-structured interior. | ||||||||||||||||||
 マップデータ マップデータ | Fungal PDC (N. crassa). Endogenous preparation-E1 E2 E3 PX. Enforced symmetry: T. Periphery is flexible/oversym. Core is structured. Interior is structured in arrangement Y / S4Y. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | acetyl transferase / pyruvate dehydrogenase / protein complex / mitochondria / metabolism / tetrahedral icosahedral / TRANSFERASE | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dihydrolipoyllysine-residue acetyltransferase / dihydrolipoyllysine-residue acetyltransferase activity / pyruvate decarboxylation to acetyl-CoA / pyruvate dehydrogenase complex / mitochondrial matrix 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Forsberg BO / Aibara S / Howard RJ / Mortezaei N / Lindahl E | ||||||||||||||||||
| 資金援助 |  スウェーデン, European Union, 5件 スウェーデン, European Union, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Arrangement and symmetry of the fungal E3BP-containing core of the pyruvate dehydrogenase complex. 著者: B O Forsberg / S Aibara / R J Howard / N Mortezaei / E Lindahl /   要旨: The pyruvate dehydrogenase complex (PDC) is a multienzyme complex central to aerobic respiration, connecting glycolysis to mitochondrial oxidation of pyruvate. Similar to the E3-binding protein (E3BP) ...The pyruvate dehydrogenase complex (PDC) is a multienzyme complex central to aerobic respiration, connecting glycolysis to mitochondrial oxidation of pyruvate. Similar to the E3-binding protein (E3BP) of mammalian PDC, PX selectively recruits E3 to the fungal PDC, but its divergent sequence suggests a distinct structural mechanism. Here, we report reconstructions of PDC from the filamentous fungus Neurospora crassa by cryo-electron microscopy, where we find protein X (PX) interior to the PDC core as opposed to substituting E2 core subunits as in mammals. Steric occlusion limits PX binding, resulting in predominantly tetrahedral symmetry, explaining previous observations in Saccharomyces cerevisiae. The PX-binding site is conserved in (and specific to) fungi, and complements possible C-terminal binding motifs in PX that are absent in mammalian E3BP. Consideration of multiple symmetries thus reveals a differential structural basis for E3BP-like function in fungal PDC. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11268.map.gz emd_11268.map.gz | 73.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11268-v30.xml emd-11268-v30.xml emd-11268.xml emd-11268.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11268_fsc.xml emd_11268_fsc.xml | 13 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11268.png emd_11268.png | 192.4 KB | ||
| Filedesc metadata |  emd-11268.cif.gz emd-11268.cif.gz | 5.7 KB | ||
| その他 |  emd_11268_half_map_1.map.gz emd_11268_half_map_1.map.gz emd_11268_half_map_2.map.gz emd_11268_half_map_2.map.gz | 97.6 MB 97.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11268 http://ftp.pdbj.org/pub/emdb/structures/EMD-11268 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11268 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11268 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6zlmMC  6zloC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10489 (タイトル: Native Pyruvate Dehydrogenase Complex from Neurospora crassa EMPIAR-10489 (タイトル: Native Pyruvate Dehydrogenase Complex from Neurospora crassaData size: 305.5 Data #1: Aligned and dose-weighted micrographs of native N. crassa Pyruvate dehydrogenase [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11268.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11268.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Fungal PDC (N. crassa). Endogenous preparation-E1 E2 E3 PX. Enforced symmetry: T. Periphery is flexible/oversym. Core is structured. Interior is structured in arrangement Y / S4Y. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.25 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half-map 1. Fungal PDC (N. crassa). Endogenous preparation-E1...
| ファイル | emd_11268_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1. Fungal PDC (N. crassa). Endogenous preparation-E1 E2 E3 PX. Enforced symmetry: T. Periphery is flexible/oversym. Core is structured. Interior is structured in arrangement Y / S4Y. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
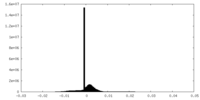
| 密度ヒストグラム |
-ハーフマップ: Half-map 2. Fungal PDC (N. crassa). Endogenous preparation-E1...
| ファイル | emd_11268_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2. Fungal PDC (N. crassa). Endogenous preparation-E1 E2 E3 PX. Enforced symmetry: T. Periphery is flexible/oversym. Core is structured. Interior is structured in arrangement Y / S4Y. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
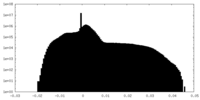
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : endogenous pyruvate dehydrogenase complex form Neurospora crassa
| 全体 | 名称: endogenous pyruvate dehydrogenase complex form Neurospora crassa |
|---|---|
| 要素 |
|
-超分子 #1: endogenous pyruvate dehydrogenase complex form Neurospora crassa
| 超分子 | 名称: endogenous pyruvate dehydrogenase complex form Neurospora crassa タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Catalytic (C-terminal) domain of Dihydrolipoyllysine-residue acetyltransferase (E2-component of pyruvate dehydrogenase complex) |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類)Organelle: mitochondria |
| 分子量 | 理論値: 7 MDa |
-分子 #1: Dihydrolipoyllysine-residue acetyltransferase component of pyruva...
| 分子 | 名称: Dihydrolipoyllysine-residue acetyltransferase component of pyruvate dehydrogenase complex, mitochondrial タイプ: protein_or_peptide / ID: 1 / コピー数: 60 / 光学異性体: LEVO / EC番号: dihydrolipoyllysine-residue acetyltransferase |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) |
| 分子量 | 理論値: 48.677395 KDa |
| 配列 | 文字列: MIVPVLSRQA LRHASVARVA LPSLTRWYAS YPPHTVVKMP ALSPTMTSGG IGAWQKKPGD KIEPGEVLVE IETDKAQMDF EFQEEGVLA KILKDSGEKD VAVGNPIAIL VEEGTDVNAF KDFTLKDAGG ETSPAVPKDE PKNESTASAP TPAPTPAPEP E NTSFTGRF ...文字列: MIVPVLSRQA LRHASVARVA LPSLTRWYAS YPPHTVVKMP ALSPTMTSGG IGAWQKKPGD KIEPGEVLVE IETDKAQMDF EFQEEGVLA KILKDSGEKD VAVGNPIAIL VEEGTDVNAF KDFTLKDAGG ETSPAVPKDE PKNESTASAP TPAPTPAPEP E NTSFTGRF QTALEREPNA LPAAKRLARE KGIDLRNVKG SGPGGKITEE DVKKALASAP AAGAAAAAYT DVPISGMRKT IA ARLKESV TENPHFFVST NLSVSKLLKL RQALNSSADG RYKLSVNDFL IKAMGIASKR VPTVNSSWRD GVIRQFETVD VSV AVATPN GLITPIVKGV EGKGLESISA AVKELAKKAR DGKLKPEEYQ GGSISISNMG MNPAVQSFTA IINPPQAAIL AVGA PQKVA VPVENEDGTT GVSWDEQIIV TASFDHKVVD GAVGAEWIRE LKKVIENPLE LLL UniProtKB: Dihydrolipoyllysine-residue acetyltransferase component of pyruvate dehydrogenase complex, mitochondrial |
-分子 #2: Pyruvate dehydrogenase X component
| 分子 | 名称: Pyruvate dehydrogenase X component / タイプ: protein_or_peptide / ID: 2 / 詳細: Uniprot Q7RWS2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) |
| 分子量 | 理論値: 1.124378 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)