+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10617 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
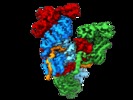
| タイトル | Subunits BBS 1,4,8,9,18 of the human BBSome complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ciliary transport / Arl6 effector / adaptor protein / complex / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of non-motile cilium assembly / protein localization to photoreceptor outer segment / RNA polymerase II-specific DNA-binding transcription factor binding => GO:0061629 / receptor localization to non-motile cilium / BBSome / retinal rod cell development / negative regulation of appetite by leptin-mediated signaling pathway / photoreceptor cell outer segment organization / microtubule anchoring at centrosome / smoothened binding ...regulation of non-motile cilium assembly / protein localization to photoreceptor outer segment / RNA polymerase II-specific DNA-binding transcription factor binding => GO:0061629 / receptor localization to non-motile cilium / BBSome / retinal rod cell development / negative regulation of appetite by leptin-mediated signaling pathway / photoreceptor cell outer segment organization / microtubule anchoring at centrosome / smoothened binding / regulation of cilium beat frequency involved in ciliary motility / sensory processing / protein localization to organelle / ciliary transition zone / melanosome transport / photoreceptor connecting cilium / patched binding / ventricular system development / negative regulation of actin filament polymerization / BBSome-mediated cargo-targeting to cilium / striatum development / Golgi to plasma membrane protein transport / positive regulation of cilium assembly / positive regulation of multicellular organism growth / response to stimulus / non-motile cilium assembly / maintenance of protein location in nucleus / photoreceptor cell maintenance / protein localization to cilium / regulation of stress fiber assembly / brain morphogenesis / non-motile cilium / negative regulation of systemic arterial blood pressure / retina homeostasis / motile cilium / centrosome cycle / protein localization to centrosome / negative regulation of GTPase activity / ciliary membrane / fat pad development / adult behavior / pericentriolar material / beta-tubulin binding / heart looping / fat cell differentiation / dendrite development / face development / axoneme / dynactin binding / social behavior / regulation of lipid metabolic process / centriolar satellite / spermatid development / mitotic cytokinesis / photoreceptor outer segment / alpha-tubulin binding / cilium assembly / intracellular transport / visual perception / centriole / photoreceptor inner segment / ciliary basal body / regulation of cytokinesis / neural tube closure / hippocampus development / neuron migration / cerebral cortex development / cilium / microtubule cytoskeleton organization / sensory perception of smell / protein transport / protein-macromolecule adaptor activity / negative regulation of gene expression / centrosome / membrane / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Klink BU / Raunser S | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Structure of the human BBSome core complex. 著者: Björn Udo Klink / Christos Gatsogiannis / Oliver Hofnagel / Alfred Wittinghofer / Stefan Raunser /  要旨: The BBSome is a heterooctameric protein complex that plays a central role in primary cilia homeostasis. Its malfunction causes the severe ciliopathy Bardet-Biedl syndrome (BBS). The complex acts as a ...The BBSome is a heterooctameric protein complex that plays a central role in primary cilia homeostasis. Its malfunction causes the severe ciliopathy Bardet-Biedl syndrome (BBS). The complex acts as a cargo adapter that recognizes signaling proteins such as GPCRs and links them to the intraflagellar transport machinery. The underlying mechanism is poorly understood. Here we present a high-resolution cryo-EM structure of a human heterohexameric core subcomplex of the BBSome. The structure reveals the architecture of the complex in atomic detail. It explains how the subunits interact with each other and how disease-causing mutations hamper this interaction. The complex adopts a conformation that is open for binding to membrane-associated GTPase Arl6 and a large positively charged patch likely strengthens the interaction with the membrane. A prominent negatively charged cleft at the center of the complex is likely involved in binding of positively charged signaling sequences of cargo proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10617.map.gz emd_10617.map.gz | 2.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10617-v30.xml emd-10617-v30.xml emd-10617.xml emd-10617.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10617.png emd_10617.png | 166.1 KB | ||
| マスクデータ |  emd_10617_msk_1.map emd_10617_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10617.cif.gz emd-10617.cif.gz | 8 KB | ||
| その他 |  emd_10617_half_map_1.map.gz emd_10617_half_map_1.map.gz emd_10617_half_map_2.map.gz emd_10617_half_map_2.map.gz | 39.4 MB 39.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10617 http://ftp.pdbj.org/pub/emdb/structures/EMD-10617 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10617 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10617 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10617_validation.pdf.gz emd_10617_validation.pdf.gz | 612.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10617_full_validation.pdf.gz emd_10617_full_validation.pdf.gz | 611.7 KB | 表示 | |
| XML形式データ |  emd_10617_validation.xml.gz emd_10617_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_10617_validation.cif.gz emd_10617_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10617 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10617 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10617 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10617 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10617.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10617.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10617_msk_1.map emd_10617_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10617_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10617_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BBSome core complex
| 全体 | 名称: BBSome core complex |
|---|---|
| 要素 |
|
-超分子 #1: BBSome core complex
| 超分子 | 名称: BBSome core complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: BBSome core complex containing BBS1,4, 8, 9 and 18. BBS5 was also present in the sample preparation, but was only visible in a subset of particles (see related entry) |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Bardet-Biedl syndrome 1 protein
| 分子 | 名称: Bardet-Biedl syndrome 1 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 65.159266 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAASSSDSD ACGAESNEAN SKWLDAHYDP MANIHTFSAC LALADLHGDG EYKLVVGDLG PGGQQPRLKV LKGPLVMTES PLPALPAAA ATFLMEQHEP RTPALALASG PCVYVYKNLR PYFKFSLPQL PPNPLEQDLW NQAKEDRIDP LTLKEMLESI R ETAEEPLS ...文字列: MAAASSSDSD ACGAESNEAN SKWLDAHYDP MANIHTFSAC LALADLHGDG EYKLVVGDLG PGGQQPRLKV LKGPLVMTES PLPALPAAA ATFLMEQHEP RTPALALASG PCVYVYKNLR PYFKFSLPQL PPNPLEQDLW NQAKEDRIDP LTLKEMLESI R ETAEEPLS IQSLRFLQLE LSEMEAFVNQ HKSNSIKRQT VITTMTTLKK NLADEDAVSC LVLGTENKEL LVLDPEAFTI LA KMSLPSV PVFLEVSGQF DVEFRLAAAC RNGNIYILRR DSKHPKYCIE LSAQPVGLIR VHKVLVVGST QDSLHGFTHK GKK LWTVQM PAAILTMNLL EQHSRGLQAV MAGLANGEVR IYRDKALLNV IHTPDAVTSL CFGRYGREDN TLIMTTRGGG LIIK ILKRT AVFVEGGSEV GPPPAQAMKL NVPRKTRLYV DQTLREREAG TAMHRAFQTD LYLLRLRAAR AYLQALESSL SPLST TARE PLKLHAVVQG LGPTFKLTLH LQNTSTTRPV LGLLVCFLYN EALYSLPRAF FKVPLLVPGL NYPLETFVES LSNKGI SDI IKVLVLREGQ SAPLLSAHVN MPGSEGLAAA UniProtKB: Bardet-Biedl syndrome 1 protein |
-分子 #2: Bardet-Biedl syndrome 4 protein
| 分子 | 名称: Bardet-Biedl syndrome 4 protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 59.46302 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MHHHHHHGPM AEERVATRTQ FPVSTESQKP RQKKAPEFPI LEKQNWLIHL HYIRKDYEAC KAVIKEQLQE TQGLCEYAIY VQALIFRLE GNIQESLELF QTCAVLSPQS ADNLKQVARS LFLLGKHKAA IEVYNEAAKL NQKDWEISHN LGVCYIYLKQ F NKAQDQLH ...文字列: MHHHHHHGPM AEERVATRTQ FPVSTESQKP RQKKAPEFPI LEKQNWLIHL HYIRKDYEAC KAVIKEQLQE TQGLCEYAIY VQALIFRLE GNIQESLELF QTCAVLSPQS ADNLKQVARS LFLLGKHKAA IEVYNEAAKL NQKDWEISHN LGVCYIYLKQ F NKAQDQLH NALNLNRHDL TYIMLGKIHL LEGDLDKAIE VYKKAVEFSP ENTELLTTLG LLYLQLGIYQ KAFEHLGNAL TY DPTNYKA ILAAGSMMQT HGDFDVALTK YRVVACAVPE SPPLWNNIGM CFFGKKKYVA AISCLKRANY LAPFDWKILY NLG LVHLTM QQYASAFHFL SAAINFQPKM GELYMLLAVA LTNLEDIENA KRAYAEAVHL DKCNPLVNLN YAVLLYNQGE KKNA LAQYQ EMEKKVSLLK DNSSLEFDSE MVEMAQKLGA ALQVGEALVW TKPVKDPKSK HQTTSTSKPA SFQQPLGSNQ ALGQA MSSA AAYRTLPSGA GGTSQFTKPP SLPLEPEPAV ESSPTETSEQ IREK UniProtKB: Bardet-Biedl syndrome 4 protein |
-分子 #3: Tetratricopeptide repeat domain 8 isoform 2
| 分子 | 名称: Tetratricopeptide repeat domain 8 isoform 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 58.702539 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MDYKDDDDKA GPMSSEMEPL LLAWSYFRRR KFQLCADLCT QMLEKSPYDQ AAWILKARAL TEMVYIDEID VDQEGIAEMM LDENAIAQV PRPGTSLKLP GTNQTGGPSQ AVRPITQAGR PITGFLRPST QSGRPGTMEQ AIRTPRTAYT ARPITSSSGR F VRLGTASM ...文字列: MDYKDDDDKA GPMSSEMEPL LLAWSYFRRR KFQLCADLCT QMLEKSPYDQ AAWILKARAL TEMVYIDEID VDQEGIAEMM LDENAIAQV PRPGTSLKLP GTNQTGGPSQ AVRPITQAGR PITGFLRPST QSGRPGTMEQ AIRTPRTAYT ARPITSSSGR F VRLGTASM LTSPDGPFIN LSRLNLTKYS QKPKLAKALF EYIFHHENDV KTALDLAALS TEHSQYKDWW WKVQIGKCYY RL GMYREAE KQFKSALKQQ EMVDTFLYLA KVYVSLDQPV TALNLFKQGL DKFPGEVTLL CGIARIYEEM NNMSSAAEYY KEV LKQDNT HVEAIACIGS NHFYSDQPEI ALRFYRRLLQ MGIYNGQLFN NLGLCCFYAQ QYDMTLTSFE RALSLAENEE EAAD VWYNL GHVAVGIGDT NLAHQCFRLA LVNNNNHAEA YNNLAVLEMR KGHVEQARAL LQTASSLAPH MYEPHFNFAT ISDKI GDLQ RSYVAAQKSE AAFPDHVDTQ HLIKQLRQHF AML UniProtKB: Tetratricopeptide repeat domain 8 isoform 2 |
-分子 #4: Protein PTHB1
| 分子 | 名称: Protein PTHB1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 99.383914 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSLFKARDWW STILGDKEEF DQGCLCLANV DNSGNGQDKI IVGSFMGYLR IFSPHPAKTG DGAQAEDLLL EVDLRDPVLQ VEVGKFVSG TEMLHLAVLH SRKLCVYSVS GTLGNVEHGN QCQMKLMYEH NLQRTACNMT YGSFGGVKGR DLICIQSMDG M LMVFEQES ...文字列: MSLFKARDWW STILGDKEEF DQGCLCLANV DNSGNGQDKI IVGSFMGYLR IFSPHPAKTG DGAQAEDLLL EVDLRDPVLQ VEVGKFVSG TEMLHLAVLH SRKLCVYSVS GTLGNVEHGN QCQMKLMYEH NLQRTACNMT YGSFGGVKGR DLICIQSMDG M LMVFEQES YAFGRFLPGF LLPGPLAYSS RTDSFLTVSS CQQVESYKYQ VLAFATDADK RQETEQQKLG SGKRLVVDWT LN IGEQALD ICIVSFNQSA SSVFVLGERN FFCLKDNGQI RFMKKLDWSP SCFLPYCSVS EGTINTLIGN HNNMLHIYQD VTL KWATQL PHIPVAVRVG CLHDLKGVIV TLSDDGHLQC SYLGTDPSLF QAPNVQSREL NYDELDVEMK ELQKIIKDVN KSQG VWPMT EREDDLNVSV VVSPNFDSVS QATDVEVGTD LVPSVTVKVT LQNRVILQKA KLSVYVQPPL ELTCDQFTFE FMTPD LTRT VSFSVYLKRS YTPSELEGNA VVSYSRPTDR NPDGIPRVIQ CKFRLPLKLI CLPGQPSKTA SHKITIDTNK SPVSLL SLF PGFASQSDDD QVNVMGFHFL GGARITVLAS KTSQRYRIQS EQFEDLWLIT NELILRLQEY FEKQGVKDFA CSFSGSI PL QEYFELIDHH FELRINGEKL EELLSERAVQ FRAIQRRLLA RFKDKTPAPL QHLDTLLDGT YKQVIALADA VEENQGNL F QSFTRLKSAT HLVILLIALW QKLSADQVAI LEAAFLPLQE DTQELGWEET VDAAISHLLK TCLSKSSKEQ ALNLNSQLN IPKDTSQLKK HITLLCDRLS KGGRLCLSTD AAAPQTMVMP GGCTTIPESD LEERSVEQDS TELFTNHRHL TAETPRPEVS PLQGVSE UniProtKB: Protein PTHB1 |
-分子 #5: BBSome-interacting protein 1
| 分子 | 名称: BBSome-interacting protein 1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.430824 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MASWSHPQFE KGSAGSAAGS GAGWSHPQFE KGAGLEVLFQ GPKRAEFMLK AAAKRPELSG KNTISNNSDM AEVKSMFREV LPKQGPLFV EDIMTMVLCK PKLLPLKSLT LEKLEKMHQA AQNTIRQQEM AEKDQRQITH UniProtKB: BBSome-interacting protein 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.08 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 286 K / 装置: FEI VITROBOT MARK III 詳細: double blot with 2 minutes incubation after first sample application. | ||||||||
| 詳細 | The sample was cross linked with 0.5% glutaraldehyde |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-50 / 実像数: 15266 / 平均露光時間: 15.0 sec. / 平均電子線量: 67.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 1.0 µm / 最小 デフォーカス(補正後): 0.3 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X