+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10240 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the native full-length HIV-1 capsid protein A92E in helical assembly (-13,11) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus ...integrase activity / Integration of viral DNA into host genomic DNA / Autointegration results in viral DNA circles / Minus-strand DNA synthesis / Plus-strand DNA synthesis / 2-LTR circle formation / Uncoating of the HIV Virion / Vpr-mediated nuclear import of PICs / Early Phase of HIV Life Cycle / Integration of provirus / APOBEC3G mediated resistance to HIV-1 infection / Binding and entry of HIV virion / viral process / viral life cycle / HIV-1 retropepsin / symbiont-mediated activation of host apoptosis / Assembly Of The HIV Virion / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / Budding and maturation of HIV virion / host multivesicular body / protein processing / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / symbiont-mediated suppression of host gene expression / viral penetration into host nucleus / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / host cell / viral capsid / peptidase activity / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / 加水分解酵素; エステル加水分解酵素 / aspartic-type endopeptidase activity / DNA-directed DNA polymerase activity / symbiont entry into host cell / viral translational frameshifting / lipid binding / host cell nucleus / host cell plasma membrane / structural molecule activity / virion membrane / DNA binding / zinc ion binding / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
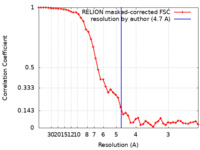
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Ni T / Gerard S / Zhao G / Ning J / Zhang P | |||||||||
| 資金援助 |  英国, 英国,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Intrinsic curvature of the HIV-1 CA hexamer underlies capsid topology and interaction with cyclophilin A. 著者: Tao Ni / Samuel Gerard / Gongpu Zhao / Kyle Dent / Jiying Ning / Jing Zhou / Jiong Shi / Jordan Anderson-Daniels / Wen Li / Sooin Jang / Alan N Engelman / Christopher Aiken / Peijun Zhang /   要旨: The mature retrovirus capsid consists of a variably curved lattice of capsid protein (CA) hexamers and pentamers. High-resolution structures of the curved assembly, or in complex with host factors, ...The mature retrovirus capsid consists of a variably curved lattice of capsid protein (CA) hexamers and pentamers. High-resolution structures of the curved assembly, or in complex with host factors, have not been available. By devising cryo-EM methodologies for exceedingly flexible and pleomorphic assemblies, we have determined cryo-EM structures of apo-CA hexamers and in complex with cyclophilin A (CypA) at near-atomic resolutions. The CA hexamers are intrinsically curved, flexible and asymmetric, revealing the capsomere and not the previously touted dimer or trimer interfaces as the key contributor to capsid curvature. CypA recognizes specific geometries of the curved lattice, simultaneously interacting with three CA protomers from adjacent hexamers via two noncanonical interfaces, thus stabilizing the capsid. By determining multiple structures from various helical symmetries, we further revealed the essential plasticity of the CA molecule, which allows formation of continuously curved conical capsids and the mechanism of capsid pattern sensing by CypA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10240.map.gz emd_10240.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10240-v30.xml emd-10240-v30.xml emd-10240.xml emd-10240.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10240_fsc.xml emd_10240_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10240.png emd_10240.png | 86.2 KB | ||
| マスクデータ |  emd_10240_msk_1.map emd_10240_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10240_half_map_1.map.gz emd_10240_half_map_1.map.gz emd_10240_half_map_2.map.gz emd_10240_half_map_2.map.gz | 5.4 MB 5.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10240 http://ftp.pdbj.org/pub/emdb/structures/EMD-10240 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10240 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10240 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10240_validation.pdf.gz emd_10240_validation.pdf.gz | 503.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10240_full_validation.pdf.gz emd_10240_full_validation.pdf.gz | 503.2 KB | 表示 | |
| XML形式データ |  emd_10240_validation.xml.gz emd_10240_validation.xml.gz | 11 KB | 表示 | |
| CIF形式データ |  emd_10240_validation.cif.gz emd_10240_validation.cif.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10240 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10240 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10240 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10240 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6sluMC  6skkC  6skmC  6sknC  6slqC  6smuC  6y9vC  6y9wC  6y9xC  6y9yC  6y9zC  6yj5C  6zdjC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10240.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10240.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.22 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10240_msk_1.map emd_10240_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
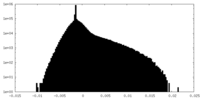
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10240_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
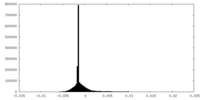
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10240_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : In vitro assembled HIV-1 capsid in tubular assembly
| 全体 | 名称: In vitro assembled HIV-1 capsid in tubular assembly |
|---|---|
| 要素 |
|
-超分子 #1: In vitro assembled HIV-1 capsid in tubular assembly
| 超分子 | 名称: In vitro assembled HIV-1 capsid in tubular assembly / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  |
-分子 #1: Gag protein
| 分子 | 名称: Gag protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 25.66442 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PIVQNIQGQM VHQAISPRTL NAWVKVVEEK AFSPEVIPMF SALSEGATPQ DLNTMLNTVG GHQAAMQMLK ETINEEAAEW DRVHPVHAG PIEPGQMREP RGSDIAGTTS TLQEQIGWMT NNPPIPVGEI YKRWIILGLN KIVRMYSPTS ILDIRQGPKE P FRDYVDRF ...文字列: PIVQNIQGQM VHQAISPRTL NAWVKVVEEK AFSPEVIPMF SALSEGATPQ DLNTMLNTVG GHQAAMQMLK ETINEEAAEW DRVHPVHAG PIEPGQMREP RGSDIAGTTS TLQEQIGWMT NNPPIPVGEI YKRWIILGLN KIVRMYSPTS ILDIRQGPKE P FRDYVDRF YKTLRAEQAS QEVKNWMTET LLVQNANPDC KTILKALGPA ATLEEMMTAC QGVGGPGHKA RVL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Purified capsid protein self-assembled in 50 mM Tris-HCl pH8.0 1M NaCl |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 6797 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 72.14 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6slu: |
 ムービー
ムービー コントローラー
コントローラー
































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)